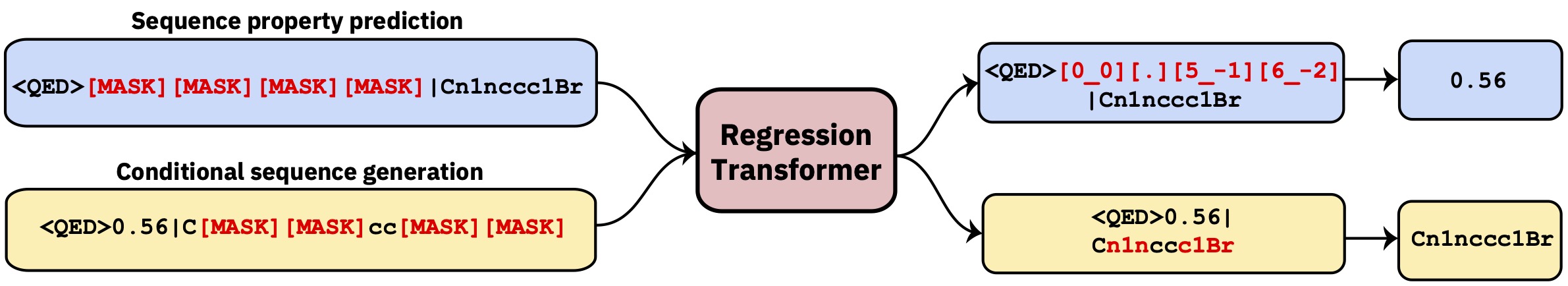
Un transformador multitarea que reformula la regresión como una tarea de modelado de secuencia condicional. Esto produce un modelo de lenguaje dicotómico que integra perfectamente la regresión con la generación condicional basada en la propiedad.
Este repositorio contiene el código de desarrollo. Lea el documento en la inteligencia de la máquina de la naturaleza .
? Una demostración de Gradio con una interfaz de usuario simple está disponible en los espacios Huggingface 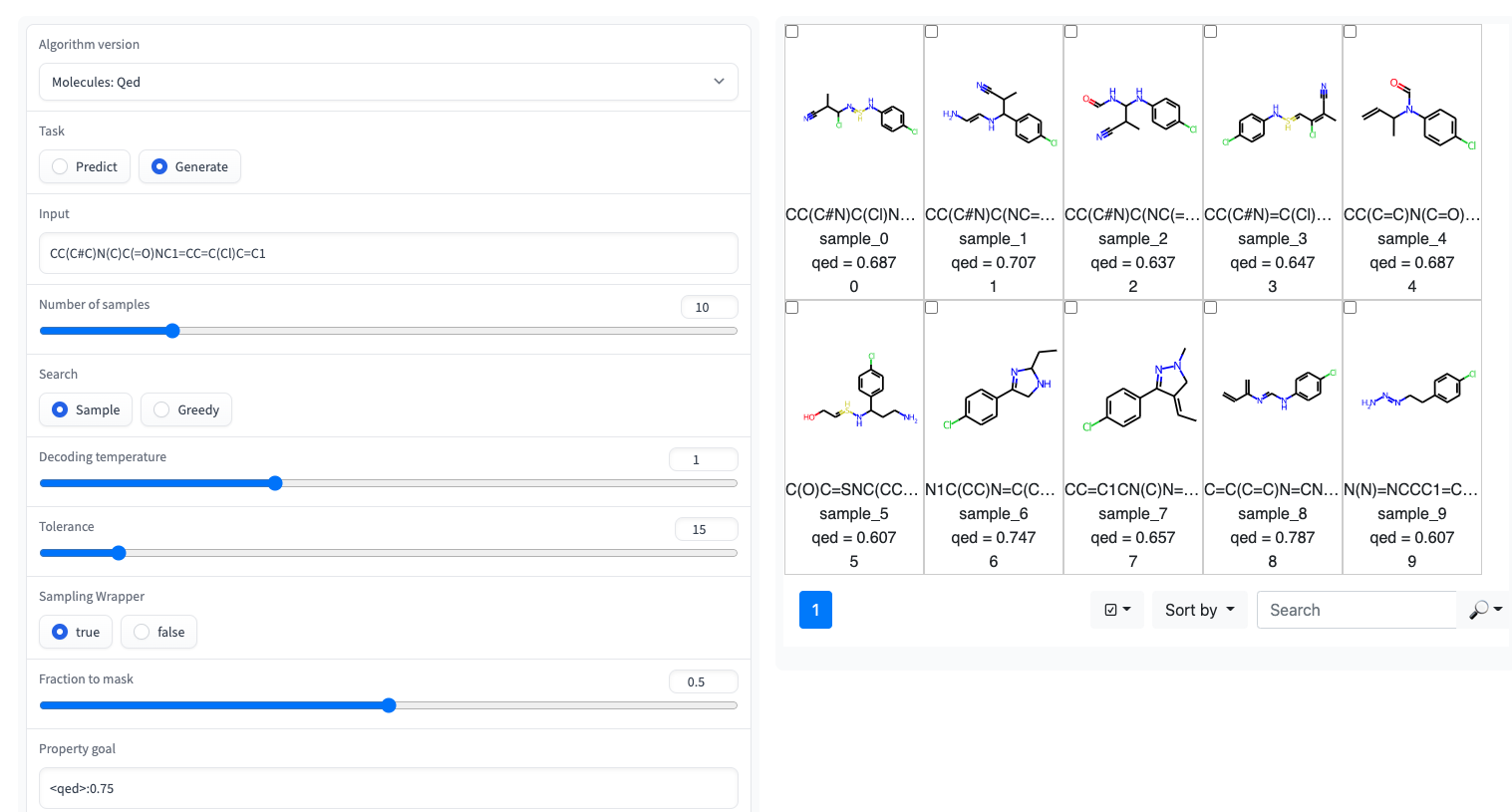
El transformador de regresión se implementa en la biblioteca GT4SD. A través de GT4SD, el uso de varios Transformadores de regresión previos a la aparición es una cuestión de unas pocas líneas de código. Se puede encontrar un tutorial completo de inferencia de ejecución, Fineting un modelo RT (o capacitarlo desde cero) y compartirlo y implementarlo en el Hub del modelo GT4SD aquí.
Por ejemplo, a través de GT4SD, puede usar el RT previamente provocado en moléculas pequeñas con algunas propiedades como se muestra en el papel, en particular QED y ESOL (solubilidad en agua). También hay varias variantes multipropertía de la RT: por ejemplo, un modelo entrenado conjuntamente en LOGP y sintetización (también conocido como SCSCORE). Para el modelado de lenguaje de proteínas, también encontrará una RT entrenada en un conjunto de datos de estabilidad de péptidos desde el punto de referencia de cinta. En resumen, GT4SD proporciona modelos RT previamente en:
qed , esol , crippen_logp ) o múltiples ( logp_and_synthesizability , cosmo_acdl , pfas ). Todos esos modelos usan selfies aparte de crippen_logp que usa sonrisas.stabilityuspto (usando sonrisas de reacción)rop_catalyst y block_copolymer se describen en Park et al., (2023; Nature Communications ). El rop_catalyst usa selfies convencionales, pero el modelo block_copolymer utiliza un nuevo lenguaje de polímero llamado CMDL descrito también en Park et al., (2023; Nature Communications ).También se proporciona un cuaderno de Jupyter con una base de juguete para adaptar una molécula hacia la solubilidad en GT4SD. Si usa GT4SD, puede generar moléculas como esta:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) Explore la solubilidad del espacio químico local alrededor de Buturon. Al variar los cebadores de la propiedad, puede obtener algo como esto: 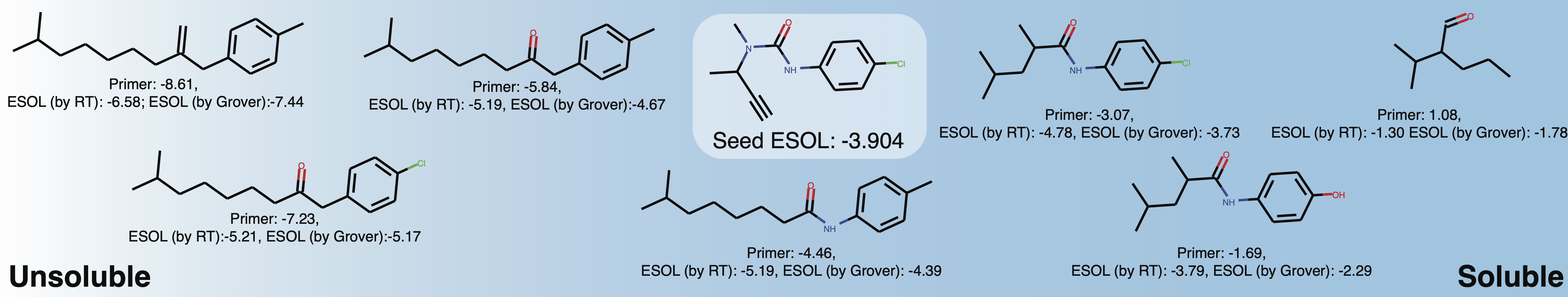
Esto es principalmente destinado a reproducir o extender los resultados del documento.
conda env create -f conda.yml
conda activate terminator
pip install -e .Los datos procesados utilizados para capacitar a los modelos están disponibles a través de la caja.
Puede descargar los datos y lanzar una capacitación señalando para entrenar y probar datos:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path apunta a un archivo que especifica el régimen de entrenamiento. Esto es opcional, si no se da el argumento, nos quedamos de forma predeterminada a la capacitación de vainilla PLM que enmascaras en todas partes con igual probabilidad (recomendado solo para el pretrénste inicial). Para ejemplos refinados, consulte la carpeta training_configs .
También tenga en cuenta que la carpeta vocabs contiene los archivos de vocabulario para entrenamiento en pequeñas moléculas, proteínas y reacciones químicas.
Se pueden encontrar configuraciones ejemplares del modelo (número de cabezas, capas, etc.) en la carpeta Configs.
Para evaluar un modelo capacitado, por ejemplo, en la tarea QED, ejecute lo siguiente:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonLos modelos previos a la aparición están disponibles a través del centro de modelos GT4SD. Hay un total de 9 modelos que también se pueden usar a través de espacios Huggingface. Los modelos que forman parte de la publicación también están disponibles a través de la carpeta de caja mencionada anteriormente.
Para generar datos personalizados para la tarea QED en un formato compatible con RT, ejecute scripts/generate_example_data.py y apunte a un archivo .smi con sonrisas en la primera columna.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtPara las propiedades definidas por el usuario, adapte el archivo o abra un problema.
Si necesita crear un nuevo vocabulario para un conjunto de datos, puede usar scripts/create_vocabulary.py, también agregará automáticamente algunos tokens especiales en la parte superior de su archivo de vocabulario.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt En este punto, la carpeta que contiene el archivo de vocabulario se puede usar para cargar un tokenizer compatible con cualquier ExpressionBertTokenizer :
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]Si usa el transformador de regresión, cite:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}