該存儲庫包含Docker圖像中NCBI BLAST+命令行應用程序的文檔。我們將使用一個小的基本示例和更高級的生產級示例演示如何在Google Cloud Platform(GCP)和Amazon Web Services(AWS)上運行BLAST分析。 UNIX/Linux命令和BLAST+的一些基本知識可用於完成本教程。
run命令選項run命令結構
國家生物技術信息中心(NCBI)基本的本地對齊搜索工具(BLAST)找到了序列之間局部相似性的區域。該程序將核苷酸或蛋白質序列與序列數據庫進行比較,併計算匹配的統計意義。爆炸可用於推斷序列之間的功能和進化關係,並幫助識別基因家族的成員。
Blast+於2009年推出,是BLAST命令行應用程序的改進版本。有關BLAST+的功能和功能的完整說明,請參閱Blast命令行應用程序用戶手冊。
雲計算通過使用按需,可擴展和彈性計算資源可節省潛在的成本。儘管此存儲庫的範圍不超出各種雲技術和好處的詳細描述,但以下各節包含開始運行Blast+ Docker映像(GCP)上運行Blast+ Docker映像所需的信息。
Docker是使用軟件容器執行操作系統級別虛擬化的工具。在容器化技術*中,圖像是封裝應用程序和依賴項的分析環境的快照。從本質上是根據指令列表構建的文件,可以保存並容易共享,以使其他人跨平台和操作系統重新創建確切的分析環境。容器是圖像的運行時實例。通過使用容器化,用戶可以繞過編譯,配置和安裝基於UNIX的工具(例如Blast+)的經常複雜的步驟。除可移植性外,容器化是一種輕巧的方法,可以使分析更加可訪問,可訪問,可互操作,可重複使用(公平),並且最終可重複。
*有許多容器化工具和標準,例如Docker和Singularity。我們將僅專注於Docker,該Docker被許多領域的許多人認為是事實上的標準。
以下各節包括使用Docker Image創建Google Virtual Machine,安裝Docker並運行Blast+命令的說明。
本節在Google實例上的Docker環境中提供了快速的爆炸分析。對於那些只想了解解決方案原理的人來說,這旨在作為概述。如果您使用Amazon實例,請轉到本文檔的Amazon Web服務設置部分。 Google Cloud Shell是一個交互式外殼環境,將用於此示例,這使得可以運行以下小示例而無需執行其他設置,例如創建計費帳戶或計算實例。本文檔的後面部分涵蓋了分析步驟,替代命令和更高級主題的更詳細的描述。
要求:Google帳戶
任務流: 
輸入數據:
首先,在單獨的瀏覽器窗口或選項卡中,在https://console.cloud.google.com/上登錄。
單擊Google Cloud Platform控制台右上角的激活雲外殼按鈕。 
現在,您將看到您的雲外殼會話窗口: 
下一步是在雲外殼會話中復制下面的命令。
請注意:在Github中,您可以使用鼠標複製;但是,在命令外殼中,您必須使用鍵盤。在Windows或Unix/Linux中,使用快捷鍵Control+C將其複制和Control+V複製到粘貼。在macOS上,使用Command+C將其複制和Command+V複製到粘貼。
要在雲外殼中滾動,請使用扳手圖標在Terminal settings中啟用滾動條。 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
此時,您應該在屏幕上看到輸出。通過查詢,BLAST將蛋白質序列P80049.1識別為分數為14.2,電子價值為0.96的匹配。
為了進行更大的分析,建議使用-out標誌將輸出保存到文件中。例如,將-out /blast/results/blastp.out blastp.out附加到上面步驟3中的最後一個命令,並使用more $HOME/results/blastp.out查看此輸出文件的內容。
您還可以對PDB查詢P01349.FSA,如以下代碼塊所示。
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
您現在已經完成了一個簡單的任務,並看到了docker的blast+如何工作。要了解Docker和Blast+在生產規模上,請繼續下一節。
在第2節中 - 使用Blast+ Docker映像的逐步指南,我們將使用上一節中的相同小示例,並討論替代方法,其他有用的Docker和Blast+命令以及Docker命令選項和結構。在第3節中,我們將演示如何在生產規模上運行Blast+ Docker圖像。
首先,您需要設置Google Cloud平台(GCP)虛擬機(VM)進行分析。
GCP目前提供300美元的信用額,從激活中12個月到期,以激勵新的雲用戶。以下步驟將向您展示如何激活此信用。將要求您獲取計費信息,但是一旦審判結束,GCP就不會自動收取您的費用;您必須選擇手動升級到付費帳戶。
登錄後,單擊激活以激活300美元的信用。 
例如,輸入您的國家,例如美國,並選中該框,指示您已閱讀並接受服務條款。
在“帳戶類型”下,選擇“個人”。 (這可以在您的Google帳戶中進行預選)
輸入您的姓名和地址。
在“您的付款方式”下,選擇“自動付款”。 (這可以在您的Google帳戶中預先選擇),這表明您使用該服務後需要支付費用,無論您是在達到帳單閾值時還是每30天(以先到者為準)支付費用。
在“付款方式”下,選擇“添加信用卡或借記卡”,然後輸入您的信用卡信息。試驗結束後,您將不會自動收費。您必須選擇在收取付款方式之前升級到付費帳戶。
單擊“開始我的免費試用”以完成註冊。此過程完成後,您應該看到GCP歡迎屏幕。
在這一點上,您應該在窗口右側看到此實例的成本估算。 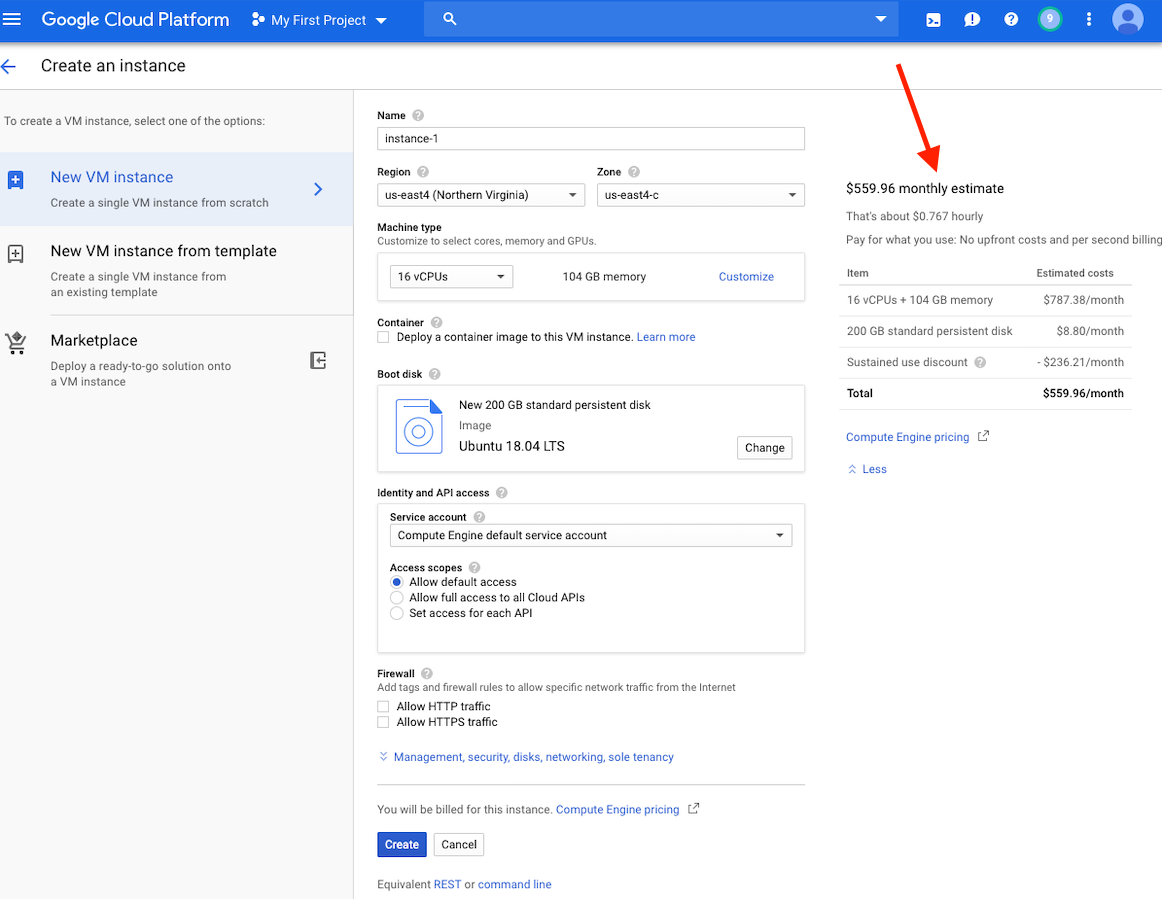
請注意:在同一區域中創建VM,因為存儲可以提供更好的性能。我們建議在US-EAST4地區創建VM。如果您的工作將需要幾個小時,但不到24小時,則可以利用可享有的VM。
可以在此處找到有關創建GCP帳戶和啟動VM的詳細說明。
創建VM後,必須從本地計算機訪問它。根據您想要使用的方式,有許多訪問VM的方法。在GCP上,最直接的方法是從瀏覽器中進行SSH。
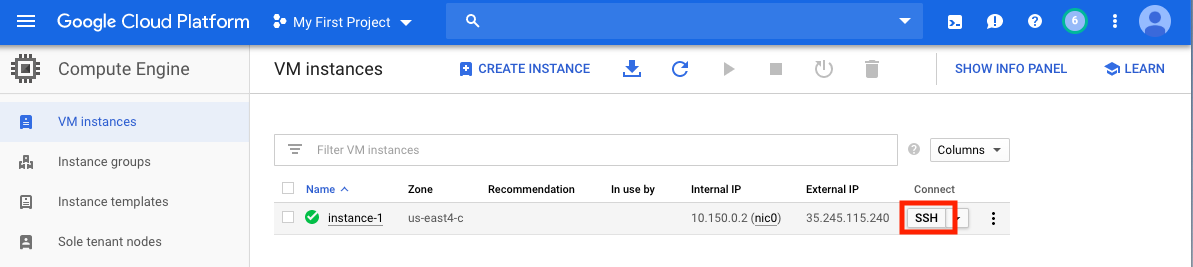
現在,您可以運行一個命令殼,並且可以繼續進行。
切記停止或刪除VM,以防止產生額外費用。
在本節中,我們將介紹Docker安裝,討論各種docker run命令選項,並檢查Docker命令的結構。我們將使用第1節中的相同示例,並探索運行Blast+ Docker映像的替代方法。但是,我們使用的是一個真實的VM實例,該實例比Google Cloud Shell提供了更大的性能和功能。
輸入數據
在生產系統中,必須將Docker作為應用程序安裝。
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
要確認Docker的正確安裝,請運行命令docker run hello-world 。如果正確安裝,您應該看到“來自Docker的Hello!...”(https://docs.docker.com/samples/library/hello-world/)
本節是可選的。
以下是本教程中使用的docker run命令行選項的列表。
| 姓名,短手(如果有) | 描述 |
|---|---|
--rm | 退出時自動卸下容器 |
--volume , -v | 綁定音量 |
--workdir , -w | 容器內的工作目錄 |
本節是可選的。
對於本教程,了解Docker命令的結構將很有用。以下命令包括三個部分。
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
命令docker run --rm ncbi/blast是運行Docker Image ncbi/blast並在運行完成後卸下容器的指令。
命令的第二部分使查詢序列數據可在容器中訪問。 Docker Bind Mounts使用-v將本地目錄安裝到容器內的目錄中,並提供訪問權限RW(讀寫)或RO(僅讀取)。例如,假設您的主題序列存儲在本地主機上的$ home/fasta目錄中,則可以使用以下參數使該目錄在/bast/fasta中可訪問/bast/fasta作為一個只讀目錄-v $HOME/fasta:/blast/fasta:ro 。 -w /blast/blastdb_custom標誌設置容器內的工作目錄。
命令的第三部分是Blast+命令。在這種情況下,它正在執行makeblastDB來創建BLAST數據庫文件。
您可以使用docker run -it ncbi/blast /bin/bash啟動此圖像的交互式BASH會話。對於Blast+ Docker映像,可執行文件位於文件夾 /Blast /bin和 /root /edirect中,並添加到變量$路徑中。
有關docker run命令的其他文檔,請參閱文檔。
本節是可選的。
| Docker命令 | 描述 |
|---|---|
docker ps -a | 顯示一個容器列表 |
docker rm $(docker ps -q -f status=exited) | 卸下所有退出的容器,如果您至少有1個退出的容器 |
docker rm <CONTAINER_ID> | 卸下容器 |
docker images | 顯示圖像列表 |
docker rmi <REPOSITORY (IMAGE_NAME)> | 刪除圖像 |
本節是可選的。
使用此Docker圖像,您可以在孤立的容器中運行BLAST+,從而促進爆炸結果的可重複性。作為此Docker映像的用戶,您有望提供BLAST數據庫和查詢序列以運行BLAST以及容器外部的位置以保存結果。以下是Blast+使用的目錄列表。您將在步驟2中創建它們。
| 目錄 | 目的 | 筆記 |
|---|---|---|
$HOME/blastdb | 存儲NCBI提供的BLAST數據庫 | 如果設置為單個絕對路徑,則可以使用$BLASTDB環境變量(請參閱通過環境變量配置BLAST。) |
$HOME/queries | 存儲用戶提供的查詢序列 | |
$HOME/fasta | 存儲用戶提供的FASTA序列以創建BLAST數據庫 | |
$HOME/results | 存儲爆炸結果 | 帶有rw權限安裝 |
$HOME/blastdb_custom | 存儲用戶提供的BLAST數據庫 |
本節是可選的。
以下命令顯示最新的BLAST版本。
docker run --rm ncbi/blast blastn -version
將標籤附加到圖像名稱( ncbi/blast )允許您使用其他版本的Blast+(有關支持的版本,請參見“支持的標籤和相應的發行說明”部分)。
不同版本的Blast+存在於不同的Docker圖像中。以下命令將啟動BLAST+版本2.9.0 Docker Image的下載。
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
例如,要使用BLAST+版本2.9.0 Docker Image而不是最新版本,請替換命令的第一部分
docker run --rm ncbi/blast docker run --rm ncbi/blast:2.9.0
本節是可選的。
在此示例中,我們將從獲取查詢和數據庫序列開始,然後創建一個自定義BLAST數據庫。
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
要驗證上面的新創建的BLAST數據庫,您可以運行以下命令以顯示數據庫中序列的訪問,序列長度和通用名稱。
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
作為替代方案,您還可以從NCBI或NCBI Google存儲存儲桶中下載預製的BLAST數據庫。
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
有關update_blastdb.pl的詳細說明,請參閱文檔。默認情況下,如果您不使用支持的雲提供商, update_blastdb.pl情況下將從雲下載或從NCBI下載。
本節是可選的。
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
本節是可選的。
下面的命令將$HOME/blastdb路徑安裝在本地機器上的AS /blast/blastdb上,並且blastdbcmd在此位置顯示了可用的BLAST數據庫。
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
您應該看到輸出/blast/blastdb/pdbaa Protein 。
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
您應該看到輸出/blast/blastdb_custom/nurse-shark-proteins Protein 。
在Docker容器中運行BLAST時,請注意指定到docker run命令的安裝座,以使輸入和輸出可訪問。在下面的示例中,前兩個安裝座提供了對BLAST數據庫的訪問權限,第三座安裝座提供了對查詢序列的訪問權限,第四座安裝座提供了一個目錄來保存結果。 (請注意:ro和:rw選項,將目錄分別固定為只讀和讀寫。)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
此時,您應該看到輸出文件$HOME/results/blastp.out 。通過查詢,BLAST將蛋白質序列P80049.1識別為分數為14.2,電子價值為0.96的匹配。要查看此輸出文件的內容,請使用命令more $HOME/results/blastp.out 。
切記停止或刪除VM,以防止產生額外費用。您可以在GCP控制台上執行此操作,如下所示。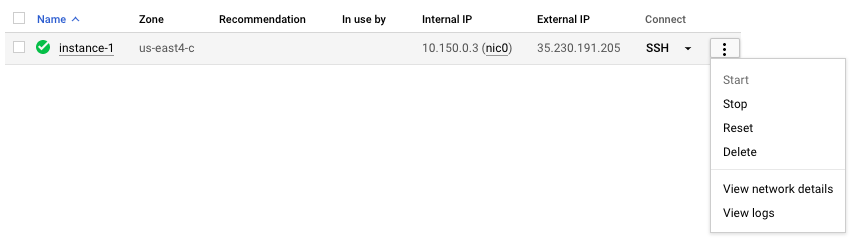
雲計算的承諾之一是可擴展性。在本節中,我們將演示如何在Google Cloud Platform上使用Bast+ Docker圖像。我們將執行類似於本出版物中描述的方法,以比較從從頭對齊的重疊群與細菌16S-23S測序與核苷酸收集(NT)數據庫相比。
為了測試可伸縮性,我們將使用不同尺寸的輸入來估計下載核苷酸收集數據庫並使用最新版本的Blast+ Docker映像的BLAST搜索的時間。以下表中總結了預期結果。
輸入文件:28個樣本(多-Fasta文件),其中包含從頭出版物中的重疊群。
(下載和創建輸入文件的說明在下面的代碼塊中描述。)
數據庫:預製的爆炸核苷酸收集數據庫,版本5(NT):68.7217 GB(2019年5月)
| 輸入文件名 | 文件內容 | 文件大小 | 序列數 | 核苷酸的數量 | 預期輸出大小 | |
|---|---|---|---|---|---|---|
| 分析1 | query1.fa | 僅樣品1 | 59 kb | 121 | 51,119 | 3.1 GB |
| 分析2 | query5.fa | 只有樣品1-5 | 422 kb | 717 | 375,154 | 10.4 GB |
| 分析3 | query.fa | 所有28個樣本 | 2.322 MB | 3798 | 2,069,892 | 47.8 GB |
| VM類型/區域 | 中央處理器 | 內存(GB) | 每小時費用* | 下載NT(最小) | 分析1(最小) | 分析2(最小) | 分析3(最小) | 總成本** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-EAST4C | 8 | 30 | $ 0.312 | 9 | 22 | - | - | - |
| N1-標準16 US-EAST4C | 16 | 60 | $ 0.611 | 9 | 14 | 53 | 205 | $ 2.86 |
| N1-HighMem-16 US-EAST4C | 16 | 104 | $ 0.767 | 9 | 9 | 30 | 143 | $ 2.44 |
| N1-HighMem-16 US-WEST2A | 16 | 104 | $ 0.809 | 11 | 9 | 30 | 147 | $ 2.60 |
| N1-HighMem-16 US-West1b | 16 | 104 | $ 0.674 | 11 | 9 | 30 | 147 | $ 2.17 |
| Blast網站(BLASTN) | - | - | - | - | 搜索超出了當前對使用的限制 | 搜索超出了當前對使用的限制 | 搜索超出了當前對使用的限制 | - |
所有GCP實例均配置為200 GB的持久標準磁盤。
*創建VM並可能更改時,Google Cloud Platform(2019年5月)提供了小時費用。
**使用小時成本和下載NT和運行分析1,分析2和分析3的總成本進行估算。估計僅用於比較;您的費用可能會有所不同,是您的監控和管理責任。
有關機器類型,區域和區域以及計算成本的更多信息,請參考GCP。
請注意,運行blastn二進製文件而不指定其-task參數調用Megablast算法。
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
您已經完成了整個教程。在這一點上,如果您不需要下載的數據進行進一步分析,請刪除VM以防止產生額外費用。
要刪除實例,請按照部分中的說明停止GCP實例。
有關其他信息,請參閱實例生命週期的Google Cloud平台的文檔。
要運行這些示例,您需要一個Amazon Web Services(AWS)帳戶。如果您還沒有一個,則可以創建一個帳戶,該帳戶可以免費提供探索和嘗試AWS服務的能力,並為每個服務提供指定的限制。首先,訪問免費級別網站,這將需要一張有效的信用卡,但是如果您在免費層中計算,則不會收取費用。選擇免費層產品時,請確保它在產品類別中。
這些說明創建了基於包括Docker及其依賴關係的Amazon Machine Image(AMI)的EC2 VM。
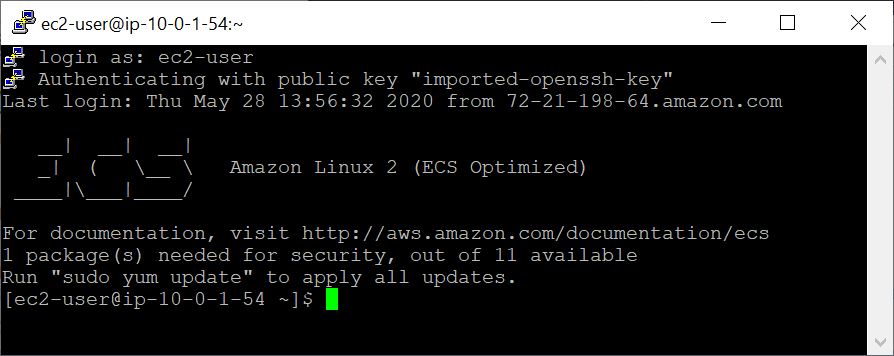
使用VM創建的VM,您可以使用SSH從本地計算機訪問它。您的密鑰對 / .pem文件可作為您的憑據。
建立SSH連接有幾種方法。從AWS控制台中的EC2實例列表中,選擇Connect ,然後按照連接方法的指令a獨立的SSH客戶端。
可以在此處找到有關連接到Linux VM的詳細說明。
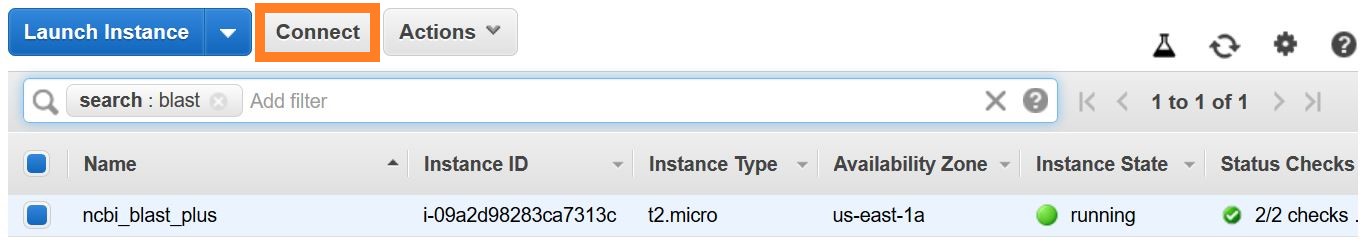
將EC2用戶指定為用戶名,而不是在您的SSH命令行中或提示登錄時將EC2用戶指定為用戶名。
在此示例中,我們將從獲取查詢和數據庫序列開始,然後創建一個自定義BLAST數據庫。
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
要驗證上面的新創建的BLAST數據庫,您可以運行以下命令以顯示數據庫中序列的訪問,序列長度和通用名稱。
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
在Docker容器中運行BLAST時,請注意指定在docker run命令中指定的安裝座( -v選項),以使輸入並輸出輸出。在下面的示例中,前兩個安裝座提供了對BLAST數據庫的訪問權限,第三座安裝座提供了對查詢序列的訪問權限,第四座安裝座提供了一個目錄來保存結果。 (請注意:ro和:rw選項,將目錄分別固定為只讀和讀寫。)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
此時,您應該看到輸出文件$HOME/results/blastp.out 。通過查詢,BLAST將蛋白質序列P80049.1識別為分數為14.2,電子價值為0.96的匹配。要查看此輸出文件的內容,請使用命令more $HOME/results/blastp.out 。
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
預期的輸出是BLAST DB的列表,包括其名稱,描述,大小和最後更新日期。
有關update_blastdb.pl的詳細說明,請參閱文檔。默認情況下,如果您不使用支持的雲提供商, update_blastdb.pl情況下將從雲下載或從NCBI下載。
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
預期的輸出是BLAST DBS名稱的列表。
請記住停止或終止VM,以防止產生額外費用。您可以從AWS控制台中的EC2實例列表中執行此操作,如下所示。
此示例需要一個多核主機。因此,通過執行此示例,將實現EC2計算費用。使用的實例類型的當前速率-t2.large-為$ 0.093/hr。
這些說明創建了基於包括Docker及其依賴關係的Amazon Machine Image(AMI)的EC2 VM。
使用VM創建的VM,您可以使用SSH從本地計算機訪問它。您的密鑰對 / .pem文件可作為您的憑據。
建立SSH連接有幾種方法。從AWS控制台中的EC2實例列表中,選擇Connect ,然後按照連接方法的指令a獨立的SSH客戶端。
可以在此處找到有關連接到Linux VM的詳細說明。
將EC2用戶指定為用戶名,而不是在您的SSH命令行中或提示登錄時將EC2用戶指定為用戶名。

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
下面的命令安裝命令(使用-v選項) $HOME/blastdb路徑AS /blast/blastdb在容器上, blastdbcmd在此位置顯示了可用的BLAST數據庫。
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
您應該看到輸出/blast/blastdb/pdbaa Protein 。
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
此時,您應該看到輸出文件$HOME/results/blastp_pdbaa.out 。要查看此輸出文件的內容,請使用命令more $HOME/results/blastp_pdbaa.out 。
在本地計算機和Linux實例之間傳輸文件的一種方法是使用安全複製協議(SCP)。
使用Amazon EC2用戶指南的Linux將文件傳輸到Linux實例的部分為此過程提供了詳細的說明。
NCBI在AWS,GCP和NCBI FTP網站上託管相同的數據庫。下表具有截至2022年11月的數據庫列表。
也可以通過命令獲得當前列表:
Docker Run -RM NCBI/BLAST UPDATE_BLASTDB.PL--SHOWALL漂亮
或者
update_blastdb.pl-下載Blast+軟件包後,ShowAll漂亮#。
如上所示,update_blastdb.pl也可以用於下載這些數據庫。它將自動選擇適當的資源(例如,如果您在該提供商中)。
這些數據庫也可以在GCP和AWS上使用ElasticBlast搜索。
訪問AWS或GCP上的數據庫以外的數據庫可能會導致您的帳戶出口費用。如果您不在雲提供商中,則應在NCBI FTP網站上使用數據庫。
| 姓名 | 類型 | 標題 |
|---|---|---|
| 16S_RIBOSOMAL_RNA | 脫氧核糖核酸 | 16S核醣體RNA(細菌和古細菌型菌株) |
| 18S_FUNGAL_SECONCES | 脫氧核糖核酸 | 來自真菌類型和參考材料的18S核醣體RNA序列(SSU) |
| 28S_FUNGAL_SECONCES | 脫氧核糖核酸 | 真菌類型和參考材料的28S核醣體RNA序列(LSU) |
| Betacoronavirus | 脫氧核糖核酸 | Betacoronavirus |
| GCF_000001405.38_TOP_LEVEL | 脫氧核糖核酸 | HOMO SAPIENS GRCH38.P12 [GCF_000001405.38]染色體加上未放置的腳手架 |
| gcf_000001635.26_top_level | 脫氧核糖核酸 | mus musculus grcm38.p6 [GCF_000001635.26]染色體加上未放置的腳手架 |
| is_refseq_fungi | 脫氧核糖核酸 | 內部轉錄的間隔區(ITS)來自真菌類型和參考材料 |
| is_eukaryote_sequences | 脫氧核糖核酸 | 它的真核生物爆炸 |
| lsu_eukaryote_rrna | 脫氧核糖核酸 | 真核生物的大型亞基核糖核酸 |
| lsu_prokaryote_rrna | 脫氧核糖核酸 | 大型亞基核醣體核酸用於原核生物 |
| ssu_eukaryote_rrna | 脫氧核糖核酸 | 小型亞基核醣體核酸,用於真核生物 |
| env_nt | 脫氧核糖核酸 | 環境樣品 |
| nt | 脫氧核糖核酸 | 核苷酸收集(NT) |
| 帕特 | 脫氧核糖核酸 | 源自GenBank的專利劃分的核苷酸序列 |
| PDBNT | 脫氧核糖核酸 | PDB核苷酸數據庫 |
| ref_euk_rep_genomes | 脫氧核糖核酸 | RefSeq真核代表性基因組數據庫 |
| ref_prok_rep_genomes | 脫氧核糖核酸 | RefSeq原核生物代表性基因組(包含RefSeq組裝) |
| ref_viroids_rep_genomes | 脫氧核糖核酸 | RefSEQ病毒代表性基因組 |
| ref_viruess_rep_genomes | 脫氧核糖核酸 | RefSEQ病毒代表性基因組 |
| refseq_rna | 脫氧核糖核酸 | NCBI成績單參考序列 |
| refseq_select_rna | 脫氧核糖核酸 | RefSeq選擇RNA序列 |
| tsa_nt | 脫氧核糖核酸 | 轉錄組shot彈槍裝配(TSA)序列 |
| env_nr | 蛋白質 | WGS元基因組項目的蛋白質 |
| 地標 | 蛋白質 | 標誌性的標誌性數據庫 |
| nr | 蛋白質 | 所有非冗餘GenBank CD翻譯+PDB+SwissProt+PIR+PRF不包括WGS項目中的環境樣本 |
| pdbaa | 蛋白質 | PDB蛋白數據庫 |
| 帕塔 | 蛋白質 | 源自GenBank的專利劃分的蛋白質序列 |
| RefSeq_protein | 蛋白質 | NCBI蛋白參考序列 |
| refseq_select_prot | 蛋白質 | RefSeq選擇蛋白質 |
| 瑞士語 | 蛋白質 | 非冗餘uniprotkb/瑞士語序列 |
| tsa_nr | 蛋白質 | 轉錄組shot彈槍裝配(TSA)序列 |
| CDD | 蛋白質 | 保守的域數據庫(CDD)是井被通知的多個序列比對模型的集合,被代表為特定位置的得分矩陣 |
NCBI為AWS,GCP和NCBI FTP站點的可用BLAST數據庫提供了元數據。
訪問AWS或GCP上的數據庫以外的數據庫可能會導致您的帳戶出口費用。如果您不在雲提供商中,則應在NCBI FTP網站上使用數據庫。
在AWS和GCP上,該文件與數據庫的日期相關子目錄。要查找最新有效的子目錄,請先讀取s3://ncbi-blast-databases/latest-dir (在AWS上)或gs://blast-db/latest-dir (在GCP上)。 latest-dir是一個文本文件,其中包含日期郵票(例如,2020-09-29-01-05-01),指定了最新目錄。適當的目錄將是BLAST數據庫的AWS或GCP基礎URI(例如s3://ncbi-blast-databases/ for AWS)以及latest-dir文件中的文本。在AWS中,URI將是s3://ncbi-blast-databases/2020-09-29-01-05-01 。 GCP URI將是相似的。
元數據文件的摘錄如下所示。大多數字段具有明顯的含義。這些文件包含BLAST數據庫。 bytes-total字段表示字節中的總BLAST數據庫大小,並旨在指定需要多少磁盤空間。
下面的示例來自AWS,但是GCP上的元數據文件具有相同的格式。 FTP網站上的數據庫中有GZPIPPECT TARFILE,每卷BLAST數據庫中有一個,因此將其列出而不是單個文件。
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
或給我們發送電子郵件。
國家生物技術信息中心(NCBI)
國家醫學圖書館(NLM)
國立衛生研究院(NIH)
查看此圖像中包含的軟件的許可證和版權信息。
與所有Docker映像一樣,這些可能還包含其他軟件,這些軟件可能來自其他許可(例如,從基本分配中,以及所包含的主要軟件的任何直接或間接依賴項)。
與任何預構建的圖像使用情況一樣,圖像用戶的責任是確保此圖像的任何使用都符合其中包含的所有軟件的任何相關許可。
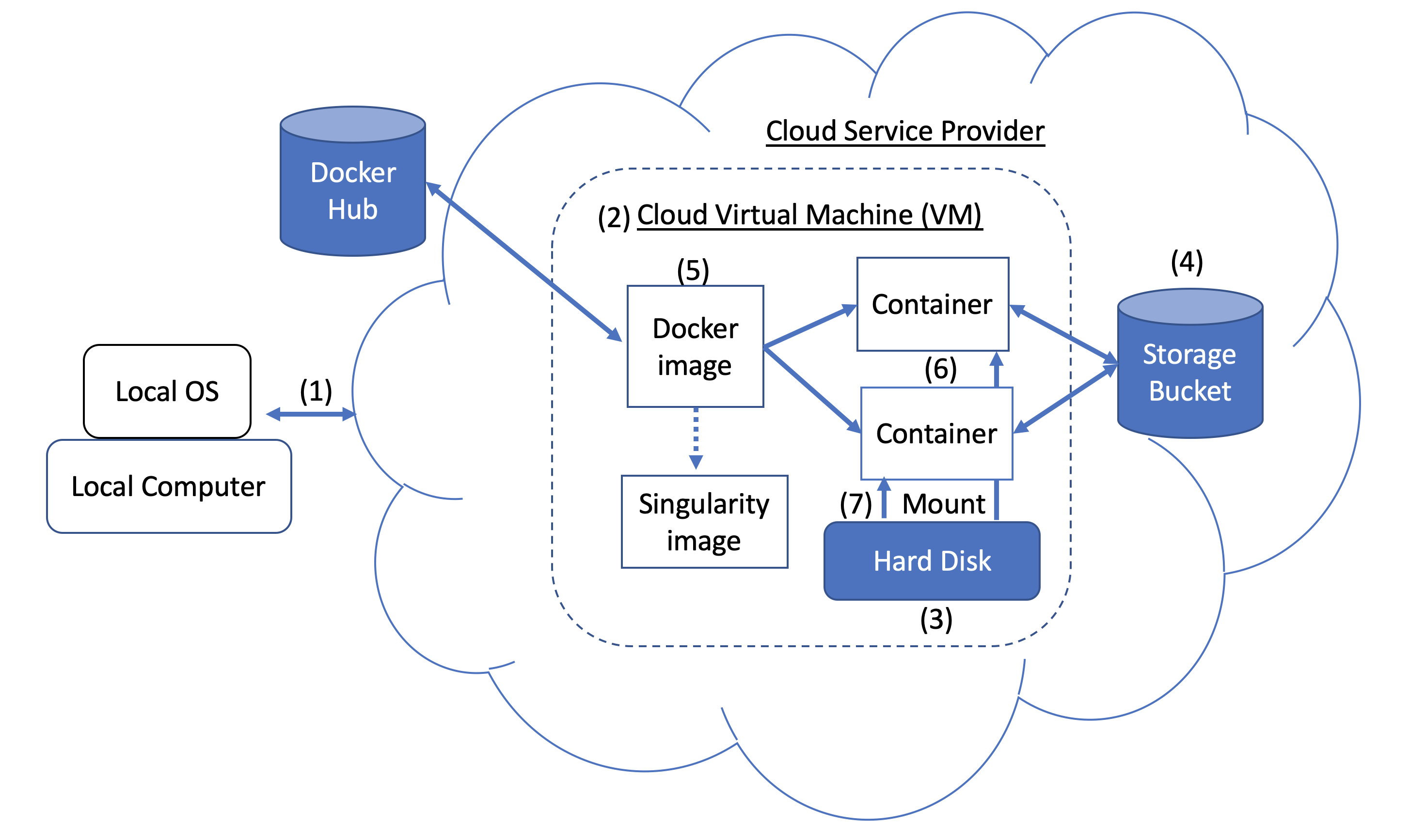 圖1。碼頭和雲計算概念。用戶可以使用SSH隧道(1)訪問云服務提供商(CSP)(例如Google Cloud平台)提供的計算資源。當您創建VM(2)時,將硬盤(也稱為啟動/持久磁盤)(3)附加到該VM上。有了正確的權限,VM還可以訪問公共領域中的其他存儲存儲桶(4)或其他數據存儲庫。一旦安裝了帶Docker的VM,您就可以運行Docker Image(5),例如NCBI的BLAST圖像。圖像可用於創建多個運行實例或容器(6)。每個容器在一個孤立的環境中。為了使數據在容器內訪問,您需要使用本教程中描述的Docker Bind Mount(7)。
圖1。碼頭和雲計算概念。用戶可以使用SSH隧道(1)訪問云服務提供商(CSP)(例如Google Cloud平台)提供的計算資源。當您創建VM(2)時,將硬盤(也稱為啟動/持久磁盤)(3)附加到該VM上。有了正確的權限,VM還可以訪問公共領域中的其他存儲存儲桶(4)或其他數據存儲庫。一旦安裝了帶Docker的VM,您就可以運行Docker Image(5),例如NCBI的BLAST圖像。圖像可用於創建多個運行實例或容器(6)。每個容器在一個孤立的環境中。為了使數據在容器內訪問,您需要使用本教程中描述的Docker Bind Mount(7)。
Docker圖像可用於創建奇異圖像。有關更多詳細信息,請參閱Singularity的文檔。
作為上述內容的替代方法,您還可以在容器內交互式運行爆炸。
何時使用:這對於在小型BLAST數據庫上運行一些(例如,少於5-10)的爆炸搜索很有用,您期望搜索在幾秒鐘/分鐘內完成。
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
此外,您可以通過在後台運行一個容器,以獨立模式運行爆炸。
何時使用:如果您有很多(例如,10個或更多)爆炸搜索要運行,或者您希望搜索需要很長時間才能執行,這是一種更實用的方法。在這種情況下,最好以獨立模式啟動爆炸容器並在其上執行命令。
注意:請確保安裝所有必需的目錄,因為啟動容器時需要指定這些目錄。
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
確認容器以獨立模式運行後,運行以下BLAST命令。
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
如果您遇到docker stop blast命令的問題,請從GCP控制台重置VM或重新啟動SSH會話。
My First Project在本地計算機上的Home Directory中復製文件$HOME/script.out instance-1
GCP文檔
首先為操作系統安裝GCP Cloud SDK命令行工具。
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.