Este repositório contém documentação para os aplicativos da linha de comando NCBI BLAST+ em uma imagem do Docker. Demonstraremos como usar a imagem do Docker para executar a análise de explosão na plataforma do Google Cloud (GCP) e na Amazon Web Services (AWS) usando um pequeno exemplo básico e um exemplo mais avançado de nível de produção. Alguns conhecimentos básicos dos comandos Unix/Linux e Blast+ são úteis para concluir este tutorial.
runrun
O Centro Nacional de Informações sobre Biotecnologia (NCBI) Ferramenta de Pesquisa de Alinhamento Local (BLAST) encontra regiões de similaridade local entre sequências. O programa compara seqüências de nucleotídeos ou proteínas a bancos de dados de sequência e calcula a significância estatística das correspondências. A explosão pode ser usada para inferir relações funcionais e evolutivas entre sequências, além de ajudar a identificar membros de famílias de genes.
Introduzido em 2009, o BLAST+ é uma versão aprimorada dos aplicativos de linha de comando BLAST. Para obter uma descrição completa dos recursos e recursos do BLAST+, consulte o manual do usuário Aplicativos de linha de comando BLAST.
A computação em nuvem oferece economia de custos potenciais usando recursos computacionais sob demanda, escaláveis e elásticos. Embora uma descrição detalhada de várias tecnologias e benefícios em nuvem esteja fora do escopo deste repositório, as seções a seguir contêm informações necessárias para começar a executar a imagem BLAST+ Docker na plataforma do Google Cloud (GCP).
O Docker é uma ferramenta para executar a virtualização do nível de sistema operacional usando contêineres de software. Na tecnologia de contêinerização * , uma imagem é um instantâneo de um ambiente analítico que encapsula aplicativos e dependências. Uma imagem, que é essencialmente um arquivo construído a partir de uma lista de instruções, pode ser salvo e facilmente compartilhado para que outras pessoas recriem o ambiente analítico exato em plataformas e sistemas operacionais. Um contêiner é uma instância de tempo de execução de uma imagem. Ao usar a contêinerização, os usuários podem ignorar as etapas frequentemente complicadas na compilação, configuração e instalação de uma ferramenta baseada em UNIX como o BLAST+. Além da portabilidade, a contêiner é uma abordagem leve para tornar a análise mais encontrada, acessível, interoperável, reutilizável (justa) e, finalmente, reproduzível.
*Existem muitas ferramentas e padrões de contêinerização, como docker e singularidade. Vamos nos concentrar apenas no Docker, que é considerado o padrão de fato de muitos no campo.
As seções a seguir incluem instruções para criar uma máquina virtual do Google, instalar o Docker e executar comandos Blast+ usando a imagem do Docker.
Esta seção fornece uma análise rápida de uma análise de explosão no ambiente do Docker em uma instância do Google. Isso se destina a uma visão geral para aqueles que desejam apenas uma compreensão dos princípios da solução. Se você trabalha com as instâncias da Amazon, vá a seção de configuração da Amazon Web Services desta documentação. O Google Cloud Shell, um ambiente de shell interativo, será usado para este exemplo, o que possibilita a execução do pequeno exemplo a seguir sem precisar executar uma configuração adicional, como criar uma conta de cobrança ou instância de computação. Descrições mais detalhadas das etapas de análise, comandos alternativos e tópicos mais avançados são abordados nas seções posteriores desta documentação.
Requisitos: uma conta do Google
Fluxo da tarefa: 
Dados de entrada:
Primeiro, em uma janela ou guia do navegador separado, faça login em https://console.cloud.google.com/
Clique no botão Active Cloud Shell no canto superior direito do console da plataforma do Google Cloud. 
Agora você verá a janela da sessão da Shell Cloud: 
A próxima etapa é copiar e contornar os comandos abaixo na sua sessão de shell em nuvem.
Observação: no github, você pode usar o mouse para copiar; No entanto, no shell de comando, você deve usar o teclado. No Windows ou Unix/Linux, use o Control+C para copiar e Control+V para colar. No macOS, use Command+C para copiar e Command+V para colar.
Para rolar no shell da nuvem, ative a barra de rolagem nas Terminal settings com o ícone da chave inglesa. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
Neste ponto, você deve ver a saída na tela. Com sua consulta, o BLAST identificou a sequência de proteínas P80049.1 como uma correspondência com uma pontuação de 14,2 e um valor E de 0,96.
Para análises maiores, é recomendável usar o sinalizador -out para salvar a saída em um arquivo. Por exemplo, append -out /blast/results/blastp.out para o último comando na etapa 3 acima e visualize o conteúdo desse arquivo de saída usando more $HOME/results/blastp.out .
Você também pode consultar o P01349.FSA contra o PDB, como mostrado no bloco de código a seguir.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
Agora você concluiu uma tarefa simples e viu como o Blast+ com o Docker funciona. Para aprender sobre o Docker e o BLAST+ em escala de produção, prossiga para a próxima seção.
Na Seção 2-Um guia passo a passo usando a imagem BLAST+ Docker, usaremos o mesmo pequeno exemplo da seção anterior e discutiremos abordagens alternativas, comandos e estruturas de comando do Docker e BLAST úteis e BLAST e as opções de comando do docker. Na Seção 3, demonstraremos como executar a imagem Blast+ Docker em escala de produção.
Primeiro, você precisa configurar uma máquina virtual (GCP) (VM) para análise.
Atualmente, o GCP está oferecendo um crédito de US $ 300, que expira 12 meses após a ativação, para incentivar novos usuários de nuvem. As etapas a seguir mostrarão como ativar esse crédito. Você receberá informações de cobrança, mas o GCP não o cobrará quando o teste terminar; Você deve optar por atualizar manualmente para uma conta paga.
Depois de fazer login, clique em Ativar para ativar o crédito de US $ 300. 
Digite seu país, por exemplo, Estados Unidos, e verifique a caixa indicando que você leu e aceita os termos de serviço.
Em "Tipo de conta", selecione "individual". (Isso pode ser pré-selecionado em sua conta do Google)
Digite seu nome e endereço.
Em "Como você paga", selecione "Pagamentos automáticos". (Isso pode ser pré-selecionado em sua conta do Google) Isso indica que você pagará custos depois de usar o serviço, quando atingir seu limite de cobrança ou a cada 30 dias, o que ocorrer primeiro.
Em "Método de pagamento", selecione "Adicionar um cartão de crédito ou débito" e insira as informações do seu cartão de crédito. Você não será carregado automaticamente assim que o teste terminar. Você deve optar por atualizar para uma conta paga antes que seu método de pagamento seja cobrado.
Clique em "Iniciar minha avaliação gratuita" para terminar o registro. Quando esse processo for concluído, você deve ver uma tela de boas -vindas do GCP.
Neste ponto, você verá uma estimativa de custo para esta instância no lado direito da sua janela. 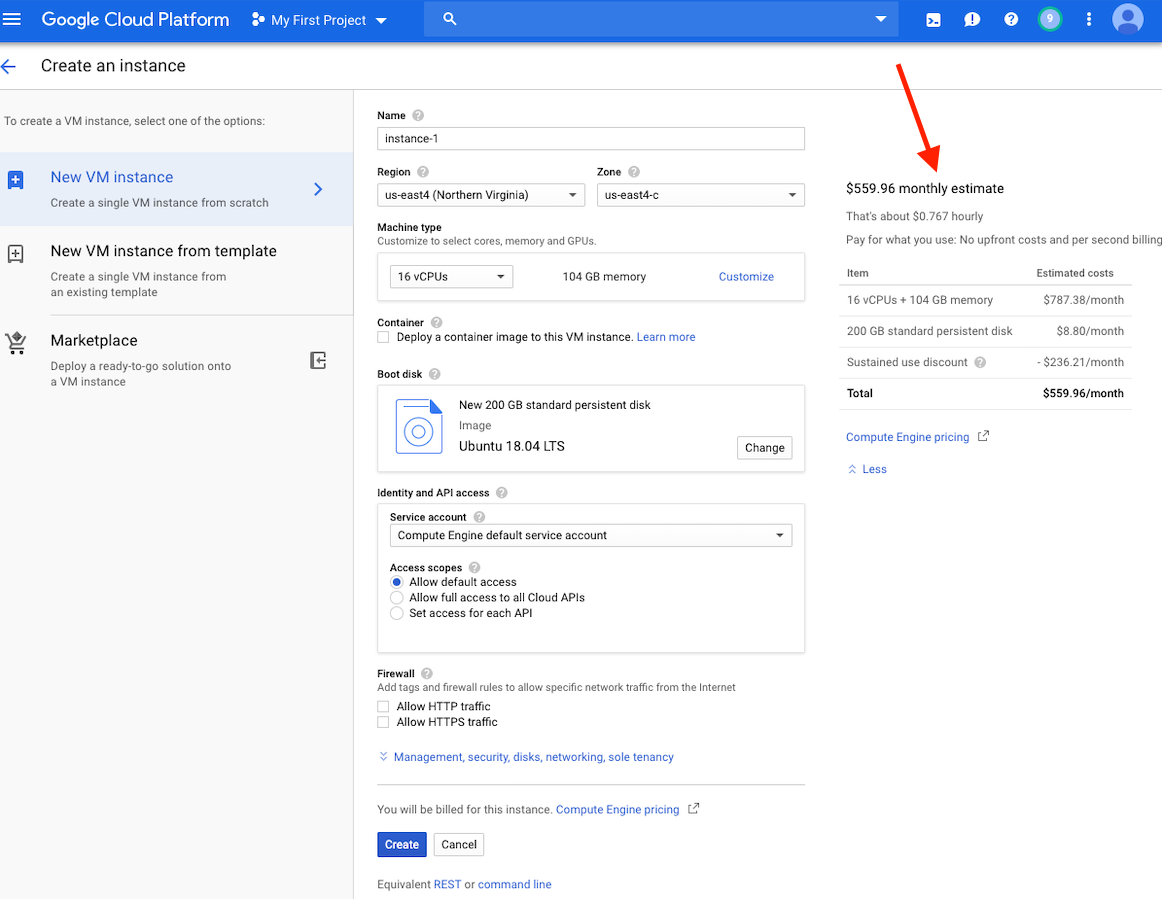
Observação: a criação de uma VM na mesma região que o armazenamento pode fornecer melhor desempenho. Recomendamos a criação de uma VM na região dos EUA-East4. Se você tiver um emprego que levará várias horas, mas menos de 24 horas, você pode aproveitar as VMs preventíveis.
Instruções detalhadas para criar uma conta GCP e iniciar uma VM podem ser encontradas aqui.
Depois de criar sua VM, você deve acessá -la no seu computador local. Existem muitos métodos para acessar sua VM, dependendo das maneiras pelas quais você gostaria de usá -lo. No GCP, a maneira mais direta é a SSH do navegador.
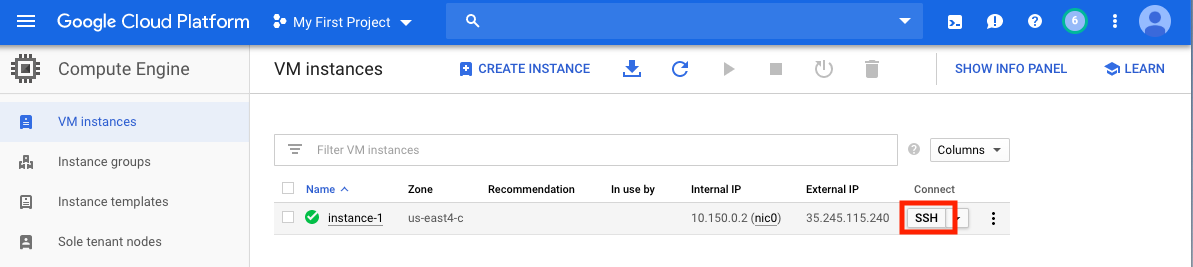
Agora você tem um shell de comando em execução e está pronto para prosseguir.
Lembre -se de parar ou excluir a VM para evitar incorrer em um custo adicional.
Nesta seção, abordaremos a instalação do Docker, discutiremos várias opções de comando docker run e examinaremos a estrutura de um comando Docker. Usaremos o mesmo pequeno exemplo da Seção 1 e exploraremos abordagens alternativas na execução da imagem Blast+ Docker. No entanto, estamos usando uma instância real da VM, que fornece maior desempenho e funcionalidade do que o shell do Google Cloud.
Dados de entrada
Em um sistema de produção, o Docker deve ser instalado como um aplicativo.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
Para confirmar a instalação correta do Docker, execute o comando docker run hello-world . Se instalado corretamente, você deve ver "Olá do Docker! ..." (https://docs.docker.com/samples/library/hello-world/)
Esta seção é opcional.
Abaixo está uma lista de opções de linha de comando docker run usadas neste tutorial.
| Nome, de mão curta (se disponível) | Descrição |
|---|---|
--rm | Remova automaticamente o contêiner quando sair |
--volume , -v | Vincular um volume |
--workdir , -w | Diretório de trabalho dentro do contêiner |
Esta seção é opcional.
Para este tutorial, seria útil entender a estrutura de um comando Docker. O comando a seguir consiste em três partes.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
A primeira parte do comando docker run --rm ncbi/blast é uma instrução para executar a imagem do Docker ncbi/blast e remover o contêiner quando a execução for concluída.
A segunda parte do comando torna os dados da sequência de consulta acessíveis no contêiner. O Docker Bind Mounts usa -v para montar os diretórios locais aos diretórios dentro do contêiner e fornecer permissão de acesso RW (Leia e escrever) ou RO (somente leitura). Por exemplo, assumindo que as seqüências de assunto são armazenadas no diretório $ home/fasta no host local, você pode usar o seguinte parâmetro para tornar esse diretório acessível dentro do contêiner em/blast/fasta como um diretório somente leitura -v $HOME/fasta:/blast/fasta:ro . O sinalizador -w /blast/blastdb_custom define o diretório de trabalho dentro do contêiner.
A terceira parte do comando é o comando BLAST+. Nesse caso, ele está executando MakeBlastDB para criar arquivos de banco de dados BLAST.
Você pode iniciar uma sessão interativa de bash para esta imagem usando docker run -it ncbi/blast /bin/bash . Para a imagem Blast+ Docker, os executáveis estão na pasta /explosão /bin e /root /edirect e adicionados ao caminho variável $.
Para documentação adicional no comando docker run , consulte a documentação.
Esta seção é opcional.
| Comando do Docker | Descrição |
|---|---|
docker ps -a | Exibe uma lista de contêineres |
docker rm $(docker ps -q -f status=exited) | Remove todos os recipientes saídos, se você tiver pelo menos 1 contêiner saiu |
docker rm <CONTAINER_ID> | Remove um recipiente |
docker images | Exibe uma lista de imagens |
docker rmi <REPOSITORY (IMAGE_NAME)> | Remove uma imagem |
Esta seção é opcional.
Com esta imagem do Docker, você pode executar o BLAST+ em um recipiente isolado, facilitando a reprodutibilidade dos resultados da explosão. Como usuário desta imagem do Docker, espera -se que você forneça bancos de dados de explosão e seqüências de consulta para executar a explosão, bem como um local fora do contêiner para salvar os resultados. A seguir, é apresentada uma lista de diretórios usados pelo BLAST+. Você os criará na etapa 2.
| Diretório | Propósito | Notas |
|---|---|---|
$HOME/blastdb | Armazena bancos de dados de explosão fornecidos pelo NCBI | Se definido como um caminho único e absoluto , a variável de ambiente $BLASTDB poderá ser usada (consulte Configurando a explosão por meio de variáveis de ambiente.) |
$HOME/queries | Armazena sequências de consulta fornecidas pelo usuário | |
$HOME/fasta | Armazena sequências FASTA fornecidas pelo usuário para criar bancos de dados de explosão (s) | |
$HOME/results | BLAST RESULTADOS DO LOJAS | Montar com permissões rw |
$HOME/blastdb_custom | Armazena bancos de dados de explosão fornecidos pelo usuário |
Esta seção é opcional.
O comando a seguir exibe a versão mais recente da explosão.
docker run --rm ncbi/blast blastn -version
Anexar uma tag ao nome da imagem ( ncbi/blast ) permite que você use uma versão diferente do BLAST+ (consulte a seção “Tags suportadas e as respectivas notas de lançamento” para versões suportadas).
Versões diferentes do BLAST+ existem em diferentes imagens do Docker. O comando a seguir iniciará o download da imagem BLAST+ Versão 2.9.0 Docker.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
Por exemplo, para usar a imagem BLAST+ versão 2.9.0 Docker em vez da versão mais recente, substitua a primeira parte do comando
docker run --rm ncbi/blast com docker run --rm ncbi/blast:2.9.0
Esta seção é opcional.
Neste exemplo, começaremos buscando sequências de consulta e banco de dados e criaremos um banco de dados de explosão personalizado.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Para verificar o banco de dados BLAST recém -criado acima, você pode executar o seguinte comando para exibir os acessos, o comprimento da sequência e o nome comum das sequências no banco de dados.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Como alternativa, você também pode baixar bancos de dados de BLAST pré -formados do NCBI ou do NCBI Google Storage Bucket.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
Para uma descrição detalhada de update_blastdb.pl , consulte a documentação. Por padrão, update_blastdb.pl download da nuvem, desde que você esteja conectado ou do NCBI se você não estiver usando um provedor de nuvem suportado.
Esta seção é opcional.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Esta seção é opcional.
O comando abaixo monta o caminho $HOME/blastdb na máquina local como /blast/blastdb no contêiner, e blastdbcmd mostra os bancos de dados de explosão disponíveis neste local.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Você deve ver uma /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
Você deve ver uma /blast/blastdb_custom/nurse-shark-proteins Protein .
Ao executar a explosão em um recipiente do docker, observe as montagens especificadas no comando docker run para tornar a entrada e as saídas acessíveis. Nos exemplos abaixo, as duas primeiras montagens fornecem acesso aos bancos de dados de explosões, a terceira montagem fornece acesso às seqüências de consulta e a quarta montagem fornece um diretório para salvar os resultados. (Observe o :ro e :rw Options, que montam os diretórios como somente leitura e leitura, respectivamente.)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
Neste ponto, você deve ver o arquivo de saída $HOME/results/blastp.out . Com sua consulta, o BLAST identificou a sequência de proteínas P80049.1 como uma correspondência com uma pontuação de 14,2 e um valor E de 0,96. Para visualizar o conteúdo deste arquivo de saída, use o comando more $HOME/results/blastp.out .
Lembre -se de parar ou excluir a VM para evitar incorrer em um custo adicional. Você pode fazer isso no console GCP, como mostrado abaixo. 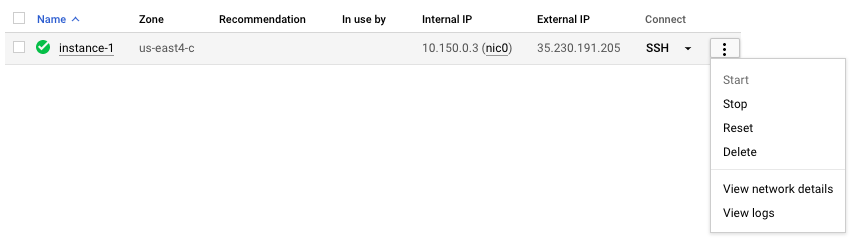
Uma das promessas da computação em nuvem é a escalabilidade. Nesta seção, demonstraremos como usar a imagem BLAST+ Docker em escala de produção na plataforma do Google Cloud. Realizaremos uma análise de explosão semelhante à abordagem descrita nesta publicação para comparar contigs alinhados de novo do sequenciamento bacteriano 16S-23S no banco de dados de coleta de nucleotídeos (NT).
Para testar a escalabilidade, usaremos as entradas de tamanhos diferentes para estimar a quantidade de tempo para baixar o banco de dados de coleta de nucleotídeos e executar a pesquisa de explosão usando a versão mais recente da imagem BLAST+ Docker. Os resultados esperados estão resumidos nas tabelas a seguir.
Arquivos de entrada: 28 amostras (arquivos multi-fasta) contendo contigs alinhados de novo da publicação.
(As instruções para baixar e criar os arquivos de entrada são descritas no bloco de código abaixo.)
Banco de dados: Banco de dados de coleta de nucleotídeos pré-formatados, versão 5 (NT): 68.7217 GB (de maio de 2019)
| Nome do arquivo de entrada | Conteúdo do arquivo | Tamanho do arquivo | Número de sequências | Número de nucleotídeos | Tamanho esperado da saída | |
|---|---|---|---|---|---|---|
| Análise 1 | Query1.fa | Somente amostra 1 | 59 KB | 121 | 51.119 | 3,1 GB |
| Análise 2 | Query5.fa | Apenas amostras 1-5 | 422 KB | 717 | 375.154 | 10,4 GB |
| Análise 3 | query.fa | Todas as 28 amostras | 2.322 MB | 3798 | 2.069.892 | 47,8 GB |
| VM tipo/zona | CPU | Memória (GB) | Custo horário* | Baixe NT (min) | Análise 1 (min) | Análise 2 (min) | Análise 3 (min) | Custo total ** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-leste4c | 8 | 30 | $ 0,312 | 9 | 22 | - | - | - |
| N1-Standard-16 EUA-East4C | 16 | 60 | $ 0,611 | 9 | 14 | 53 | 205 | US $ 2,86 |
| N1-Highmem-16 US-EAST4C | 16 | 104 | $ 0,767 | 9 | 9 | 30 | 143 | US $ 2,44 |
| N1-Highmem-16 US-West2a | 16 | 104 | $ 0,809 | 11 | 9 | 30 | 147 | US $ 2,60 |
| N1-Highmem-16 US-West1b | 16 | 104 | $ 0,674 | 11 | 9 | 30 | 147 | US $ 2,17 |
| Site de explosão (Blastn) | - | - | - | - | As pesquisas excedem as restrições atuais no uso | As pesquisas excedem as restrições atuais no uso | As pesquisas excedem as restrições atuais no uso | - |
Todas as instâncias do GCP estão configuradas com 200 GB de disco padrão persistente.
*Os custos horários foram fornecidos pelo Google Cloud Platform (maio de 2019) quando as VMs foram criadas e estão sujeitas a alterações.
** Os custos totais foram estimados usando o custo horário e o tempo total para baixar o NT e executar a análise 1, a análise 2 e a análise 3. As estimativas são usadas apenas para comparação; Seus custos podem variar e são sua responsabilidade de monitorar e gerenciar.
Consulte o GCP para obter mais informações sobre tipos de máquinas, regiões e zonas e calcular o custo.
Observe que a execução do blastn Binary sem especificar seu parâmetro -task chama o algoritmo Megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
Você concluiu o tutorial inteiro. Neste ponto, se você não precisar dos dados baixados para análises adicionais, exclua a VM para evitar o custo adicional de incorrer.
Para excluir uma instância, siga as instruções na seção, parem a instância do GCP.
Para obter informações adicionais, consulte a documentação do Google Cloud Platform no ciclo de vida da instância.
Para executar esses exemplos, você precisará de uma conta da Amazon Web Services (AWS). Se você ainda não possui um, pode criar uma conta que forneça a capacidade de explorar e experimentar os serviços da AWS gratuitamente para limites especificados para cada serviço. Para começar a visitar o site de nível gratuito, isso exigirá um cartão de crédito válido, mas não será cobrado se você calcular dentro da camada gratuita. Ao escolher um produto de camada gratuito, verifique se ele está na computação da categoria de produto.
Essas instruções criam uma VM EC2 com base em uma imagem da Amazon Machine (AMI) que inclui o Docker e suas dependências.
Com a VM criada, você a acessa no computador local usando o SSH. Seu par / .PEM FILE serve como sua credencial.
Existem várias maneiras de estabelecer uma conexão SSH. Na lista de instâncias do EC2 no console da AWS, selecione Connect e siga as instruções para o método de conexão um cliente SSH independente .
As instruções detalhadas para se conectar a uma VM Linux podem ser encontradas aqui.
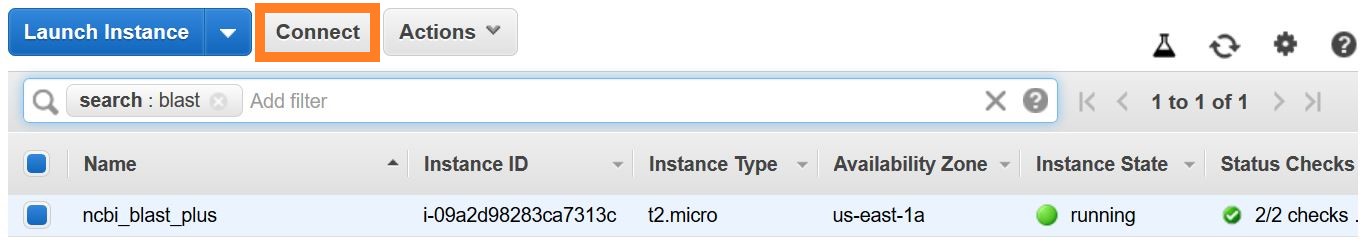
Especifique o EC2-User como o nome de usuário, em vez de root na sua linha de comando SSH ou quando solicitado a fazer login, especifique o EC2-User como o nome de usuário.
Neste exemplo, começaremos buscando sequências de consulta e banco de dados e criaremos um banco de dados de explosão personalizado.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Para verificar o banco de dados BLAST recém -criado acima, você pode executar o seguinte comando para exibir os acessos, o comprimento da sequência e o nome comum das sequências no banco de dados.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Ao executar a explosão em um contêiner do docker, observe as montagens ( -v opção) especificadas para o comando docker run para tornar a entrada e as saídas acessíveis. Nos exemplos abaixo, as duas primeiras montagens fornecem acesso aos bancos de dados de explosões, a terceira montagem fornece acesso às seqüências de consulta e a quarta montagem fornece um diretório para salvar os resultados. (Observe o :ro e :rw Options, que montam os diretórios como somente leitura e leitura, respectivamente.)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
Neste ponto, você deve ver o arquivo de saída $HOME/results/blastp.out . Com sua consulta, o BLAST identificou a sequência de proteínas P80049.1 como uma correspondência com uma pontuação de 14,2 e um valor E de 0,96. Para visualizar o conteúdo deste arquivo de saída, use o comando more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
A saída esperada é uma lista de DBs de explosão, incluindo seu nome, descrição, tamanho e última data atualizada.
Para uma descrição detalhada de update_blastdb.pl , consulte a documentação. Por padrão, update_blastdb.pl download da nuvem, desde que você esteja conectado ou do NCBI se você não estiver usando um provedor de nuvem suportado.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
A saída esperada é uma lista dos nomes do BLAST DBS.
Lembre -se de parar ou encerrar a VM para evitar incorrer em um custo adicional. Você pode fazer isso na lista de instâncias do EC2 no console da AWS, como mostrado abaixo.
Este exemplo requer um host com vários núcleos. Como tal, as cobranças de computação EC2 serão realizadas executando este exemplo. A taxa atual para o tipo de instância usada - T2.Large - é de US $ 0,093/hr.
Essas instruções criam uma VM EC2 com base em uma imagem da Amazon Machine (AMI) que inclui o Docker e suas dependências.
Com a VM criada, você a acessa no computador local usando o SSH. Seu par / .PEM FILE serve como sua credencial.
Existem várias maneiras de estabelecer uma conexão SSH. Na lista de instâncias do EC2 no console da AWS, selecione Connect e siga as instruções para o método de conexão um cliente SSH independente .
As instruções detalhadas para se conectar a uma VM Linux podem ser encontradas aqui.
Especifique o EC2-User como o nome de usuário, em vez de root na sua linha de comando SSH ou quando solicitado a fazer login, especifique o EC2-User como o nome de usuário.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
O comando abaixo é montado (usando a opção -v ) o caminho $HOME/blastdb na máquina local como /blast/blastdb no contêiner, e blastdbcmd mostra os bancos de dados de explosão disponíveis neste local.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Você deve ver uma /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
Neste ponto, você deve ver o arquivo de saída $HOME/results/blastp_pdbaa.out . Para visualizar o conteúdo deste arquivo de saída, use o comando more $HOME/results/blastp_pdbaa.out .
Uma maneira de transferir arquivos entre o computador local e uma instância do Linux é usar o Protocolo de Copiar Seguro (SCP).
A seção transferindo arquivos para instâncias do Linux do Linux usando o SCP do guia do usuário do Amazon EC2 para instâncias do Linux fornece instruções detalhadas para esse processo.
O NCBI hospeda os mesmos bancos de dados na AWS, GCP e no site NCBI FTP. A tabela abaixo possui a lista de bancos de dados atualizados em novembro de 2022.
Também é possível obter a lista atual com o comando:
Docker Run -RM NCBI/BLAST UPDATE_BLASTDB.PL -Showall Pretty
ou
UPDATE_BLASTDB.PL -Showall Pretty # Após o download do pacote Blast+.
Como mostrado acima, update_blastdb.pl também pode ser usado para baixar esses bancos de dados. Ele selecionará automaticamente o recurso apropriado (por exemplo, GCP se você estiver dentro desse provedor).
Esses bancos de dados também podem ser pesquisados com o ElasticBlast no GCP e na AWS.
O acesso aos bancos de dados na AWS ou GCP fora do provedor de nuvem provavelmente resultará em cobranças de saída em sua conta. Se você não estiver no provedor de nuvem, use os bancos de dados no site NCBI FTP.
| Nome | Tipo | Título |
|---|---|---|
| 16S_RIBOSOMAL_RNA | DNA | RNA ribossômico 16S (bactérias e tensões do tipo arquea) |
| 18s_fungal_seques | DNA | Sequências de RNA ribossômico 18S (SSU) do tipo de fungos e material de referência |
| 28s_fungal_seques | DNA | 28S sequências de RNA ribossômico (LSU) do tipo de fungos e material de referência |
| Betacoronavírus | DNA | Betacoronavírus |
| Gcf_000001405.38_top_level | DNA | HOMO SAPIENS GRCH38.P12 [GCF_000001405.38] Cromossomos mais andaimes não colocados e não abordados |
| Gcf_000001635.26_top_level | DNA | Mus musculus grcm38.p6 [gcf_000001635.26] cromossomos mais andaimes não colocados e não ocorros |
| Its_refseq_fungi | DNA | Região do espaçador transcrito interno (ITS) do tipo de fungos e material de referência |
| Its_eukaryote_seques | DNA | Sua explosão de eucarioto |
| Lsu_eukaryote_rrna | DNA | Grande ácido nucleico ribossômico subunidades para eucariotos |
| LSU_PROKARYOTE_RRNA | DNA | Grande ácido nucleico ribossômico subunidades para procariontes |
| SSU_EUKARYOTE_RRNA | DNA | Ácido nucleico ribossômico de pequena subunidade para eucariotos |
| Env_NT | DNA | amostras ambientais |
| nt | DNA | Coleção de nucleotídeos (NT) |
| Patnt | DNA | Sequências nucleotídicas derivadas da divisão de patentes do GenBank |
| pdbnt | DNA | Banco de dados de nucleotídeos PDB |
| REF_EUK_REP_GENOMES | DNA | Banco de dados do genoma representativo eucariótico refseq |
| REF_PROK_REP_GENOMES | DNA | Refseq Prokaryote Representative Genomes (contém a Assembléia Refseq) |
| ref_viroids_rep_genomes | DNA | Refseq viróides genomas representativos |
| REF_VIRUSES_REP_GENOMES | DNA | Genomas representativos dos vírus refseq |
| refseq_rna | DNA | Sequências de referência de transcrição do NCBI |
| refseq_select_rna | DNA | Refseq Selecione sequências de RNA |
| tsa_nt | DNA | Sequências de montagem de espingarda de transcriptoma (TSA) |
| Env_nr | Proteína | Proteínas de projetos metagenômicos do WGS |
| marco | Proteína | Banco de dados de marco para SmartBlast |
| nr | Proteína | Todas as traduções não redundantes do GenBank CDS+PDB+SWISSPROT+PIR+PRF excluindo amostras ambientais de projetos WGS |
| pdbaa | Proteína | Banco de dados de proteínas PDB |
| Pataa | Proteína | Sequências de proteínas derivadas da divisão de patentes do GenBank |
| refseq_protein | Proteína | Sequências de referência de proteínas NCBI |
| refseq_select_prot | Proteína | Refseq Selecione proteínas |
| SwissProt | Proteína | Sequências não redundantes uniprotkb/swissprot |
| tsa_nr | Proteína | Sequências de montagem de espingarda de transcriptoma (TSA) |
| CDD | Proteína | O banco de dados de domínio conservado (CDD) é uma coleção de modelos de alinhamento de sequência múltipla bem anotados representados como matrizes de pontuação específicas da posição |
O NCBI fornece metadados para os bancos de dados BLAST disponíveis na AWS, GCP e no site NCBI FTP.
O acesso aos bancos de dados na AWS ou GCP fora do provedor de nuvem provavelmente resultará em cobranças de saída em sua conta. Se você não estiver no provedor de nuvem, use os bancos de dados no site NCBI FTP.
Na AWS e GCP, o arquivo está em um subdiretório dependente de data com os bancos de dados. Para encontrar o subdiretório válido mais recente, primeiro leia s3://ncbi-blast-databases/latest-dir (na AWS) ou gs://blast-db/latest-dir (no GCP). latest-dir é um arquivo de texto com um carimbo de data (por exemplo, 2020-09-29-01-05-01) especificando o diretório mais recente. O diretório adequado será o URI base do AWS ou GCP para os bancos de dados BLAST (por exemplo, s3://ncbi-blast-databases/ for AWS) mais o texto no arquivo latest-dir . Um exemplo de URI, na AWS, seria s3://ncbi-blast-databases/2020-09-29-01-05-01 . O URI do GCP seria semelhante.
Um trecho de um arquivo de metadados é mostrado abaixo. A maioria dos campos tem significados óbvios. Os arquivos compreendem o banco de dados BLAST. O campo bytes-total representa o tamanho total do banco de dados BLAST em bytes e tem como objetivo especificar quanto espaço em disco é necessário.
O exemplo abaixo é da AWS, mas os arquivos de metadados no GCP têm o mesmo formato. Os bancos de dados no site FTP estão em lonas Gzipp, um por volume do banco de dados BLAST; portanto, eles são listados em vez dos arquivos individuais.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
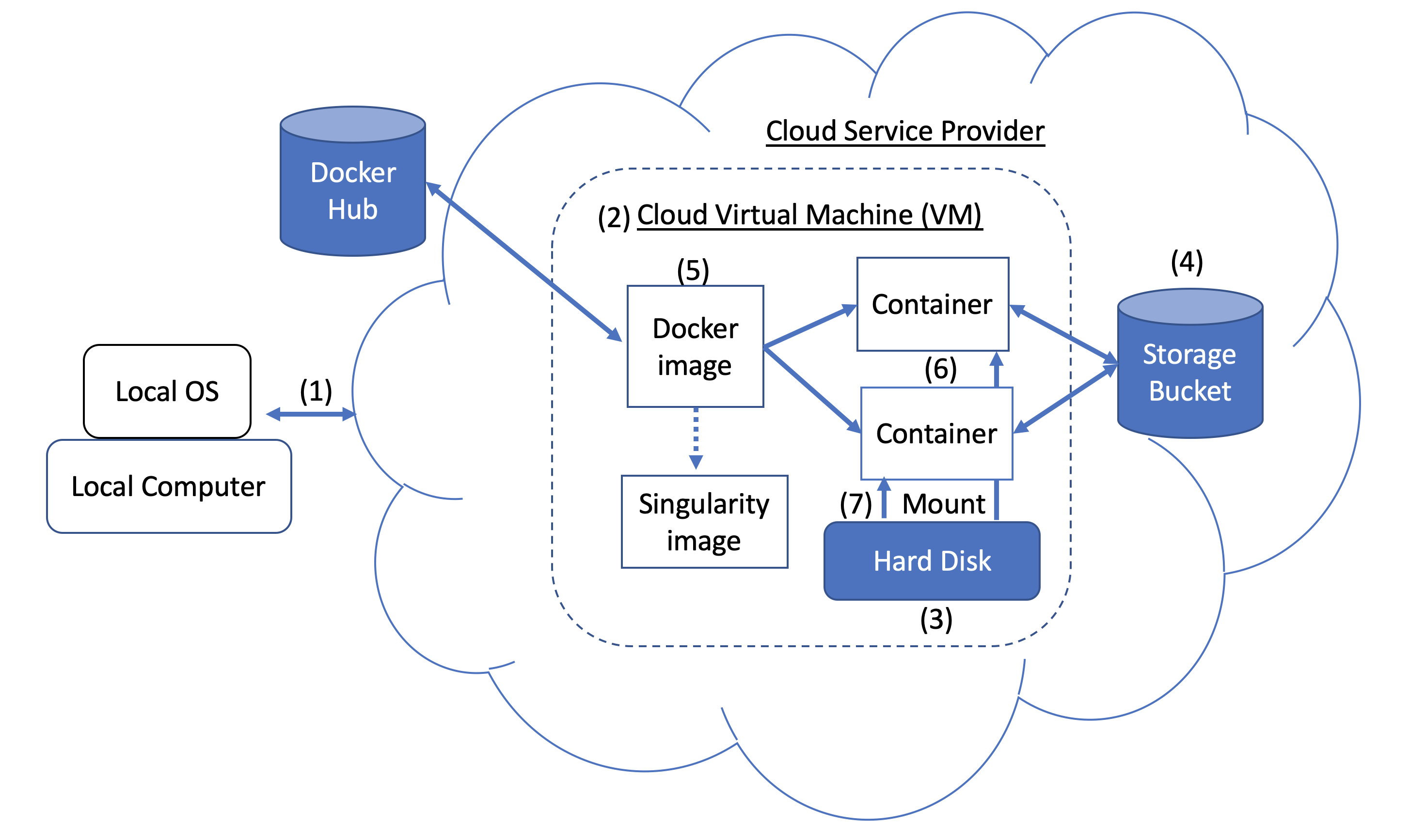 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.