Ce référentiel contient une documentation pour les applications de ligne de commande NCBI BLAST + dans une image Docker. Nous montrerons comment utiliser l'image Docker pour exécuter l'analyse BLAST sur la plate-forme Google Cloud (GCP) et les services Web d'Amazon (AWS) à l'aide d'un petit exemple de base et d'un exemple de niveau de production plus avancé. Certaines connaissances de base des commandes UNIX / Linux et BLAST + sont utiles pour terminer ce tutoriel.
runrun la structure de commande
Le National Center for Biotechnology Information (NCBI) L'outil de recherche d'alignement local de base (BLAST) trouve des régions de similitude locale entre les séquences. Le programme compare les séquences nucléotidiques ou protéiques aux bases de données de séquence et calcule la signification statistique des correspondances. Le souffle peut être utilisé pour déduire les relations fonctionnelles et évolutives entre les séquences ainsi que pour aider à identifier les membres des familles de gènes.
Introduit en 2009, BLAST + est une version améliorée des applications de ligne de commande BLAST. Pour une description complète des fonctionnalités et des capacités de BLAST +, veuillez vous référer au manuel d'utilisation des applications de ligne de commande BLAST.
Le cloud computing offre des économies de coûts potentielles en utilisant des ressources de calcul à la demande, évolutives et élastiques. Bien qu'une description détaillée de diverses technologies et avantages cloud soit hors de portée de ce référentiel, les sections suivantes contiennent des informations nécessaires pour commencer à exécuter l'image BLAST + Docker sur la plate-forme Google Cloud (GCP).
Docker est un outil pour effectuer une virtualisation au niveau du système opérationnel à l'aide de conteneurs logiciels. Dans la technologie de contenerisation * , une image est un instantané d'un environnement analytique encapsulant les applications et les dépendances. Une image, qui est essentiellement un fichier construit à partir d'une liste d'instructions, peut être enregistrée et facilement partagée pour que d'autres recréent l'environnement analytique exact sur les plates-formes et les systèmes d'exploitation. Un conteneur est une instance d'exécution d'une image. En utilisant la conteneurisation, les utilisateurs peuvent contourner les étapes souvent compliquées dans la compilation, la configuration et l'installation d'un outil basé sur Unix comme BLAST +. En plus de la portabilité, la conteneurisation est une approche légère pour rendre l'analyse plus trouvable, accessible, interopérable, réutilisable (équitable) et, finalement, reproductible.
* Il existe de nombreux outils et normes de contenerisation, tels que Docker et la singularité. Nous nous concentrerons uniquement sur Docker, qui est considéré comme la norme de facto par beaucoup dans le domaine.
Les sections suivantes incluent des instructions pour créer une machine virtuelle Google, installer Docker et Exécuter les commandes BLAST + à l'aide de l'image Docker.
Cette section fournit une analyse rapide d'une analyse BLAST dans l'environnement Docker sur une instance Google. Ceci est destiné à un aperçu de ceux qui veulent juste une compréhension des principes de la solution. Si vous travaillez avec Amazon Instances, veuillez consulter la section de configuration des services Web Amazon de cette documentation. Le Google Cloud Shell, un environnement de shell interactif, sera utilisé pour cet exemple, ce qui permet d'exécuter le petit exemple suivant sans avoir à effectuer une configuration supplémentaire, comme la création d'un compte de facturation ou d'une instance de calcul. Des descriptions plus détaillées des étapes d'analyse, des commandes alternatives et des sujets plus avancés sont couverts dans les sections ultérieures de cette documentation.
Exigences: un compte Google
Flux de la tâche:
Données d'entrée:
Tout d'abord, dans une fenêtre ou un onglet de navigateur séparé, connectez-vous à https://console.cloud.google.com/
Cliquez sur le bouton Activer le shell cloud dans le coin supérieur droit de la console Google Cloud Platform. 
Vous verrez maintenant votre fenêtre de session de coquille de cloud: 
L'étape suivante consiste à copier et à coller les commandes ci-dessous dans votre session de shell cloud.
Veuillez noter: Dans GitHub, vous pouvez utiliser votre souris pour copier; Cependant, dans le shell de commande, vous devez utiliser votre clavier. Dans Windows ou Unix / Linux, utilisez le Control+C pour copier et Control+V pour coller. Sur macOS, utilisez Command+C pour copier et Command+V pour coller.
Pour faire défiler le shell cloud, activez la barre de défilement dans Terminal settings avec l'icône de clé. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
À ce stade, vous devriez voir la sortie à l'écran. Avec votre requête, BLAST a identifié la séquence protéique P80049.1 comme un match avec un score de 14,2 et une valeur électronique de 0,96.
Pour une analyse plus grande, il est recommandé d'utiliser l'indicateur -out pour enregistrer la sortie dans un fichier. Par exemple, appelez -out /blast/results/blastp.out vers la dernière commande de l'étape 3 ci-dessus et affichez le contenu de ce fichier de sortie en utilisant more $HOME/results/blastp.out .
Vous pouvez également interroger P01349.FSA contre le PDB comme indiqué dans le bloc de code suivant.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
Vous avez maintenant terminé une tâche simple et vu comment fonctionne Blast + avec Docker. Pour en savoir plus sur Docker et Blast + à l'échelle de production, veuillez passer à la section suivante.
Dans la section 2 - un guide étape par étape utilisant l'image BLAST + Docker, nous utiliserons le même petit exemple de la section précédente et discuterons des approches alternatives, des commandes Docker et BLAST + utiles supplémentaires et des options et structures de commande Docker. Dans la section 3, nous montrerons comment exécuter l'image BLAST + Docker à l'échelle de production.
Tout d'abord, vous devez configurer une machine virtuelle (VM) de la plate-forme Google Cloud (GCP) pour l'analyse.
GCP offre actuellement un crédit de 300 $, qui expire 12 mois à partir de l'activation, pour inciter les nouveaux utilisateurs de cloud. Les étapes suivantes vous montreront comment activer ce crédit. On vous demandera des informations de facturation, mais le GCP ne vous fera pas automatiquement une fois la fin de l'essai; Vous devez choisir de passer manuellement à un compte payant.
Après avoir signé, cliquez sur Activer pour activer le crédit de 300 $.
Entrez votre pays, par exemple, les États-Unis et cochez la case indiquant que vous avez lu et accepté les conditions d'utilisation.
Sous «Type de compte», sélectionnez «Individu». (Cela peut être présélectionné dans votre compte Google)
Entrez votre nom et votre adresse.
Sous «Comment vous payez», sélectionnez «Paiements automatiques». (Cela peut être présélectionné dans votre compte Google) Cela indique que vous paierez les frais après avoir utilisé le service, soit lorsque vous avez atteint votre seuil de facturation ou tous les 30 jours, selon la première éventualité.
Dans le cadre du «mode de paiement», sélectionnez «Ajouter une carte de crédit ou de débit» et entrez les informations de votre carte de crédit. Vous ne serez pas automatiquement facturé une fois l'essai terminé. Vous devez choisir de passer à un compte payant avant que votre mode de paiement ne soit facturé.
Cliquez sur «Démarrer mon essai gratuit» pour terminer l'inscription. Une fois ce processus terminé, vous devriez voir un écran de bienvenue GCP.
À ce stade, vous devriez voir une estimation des coûts pour cette instance sur le côté droit de votre fenêtre. 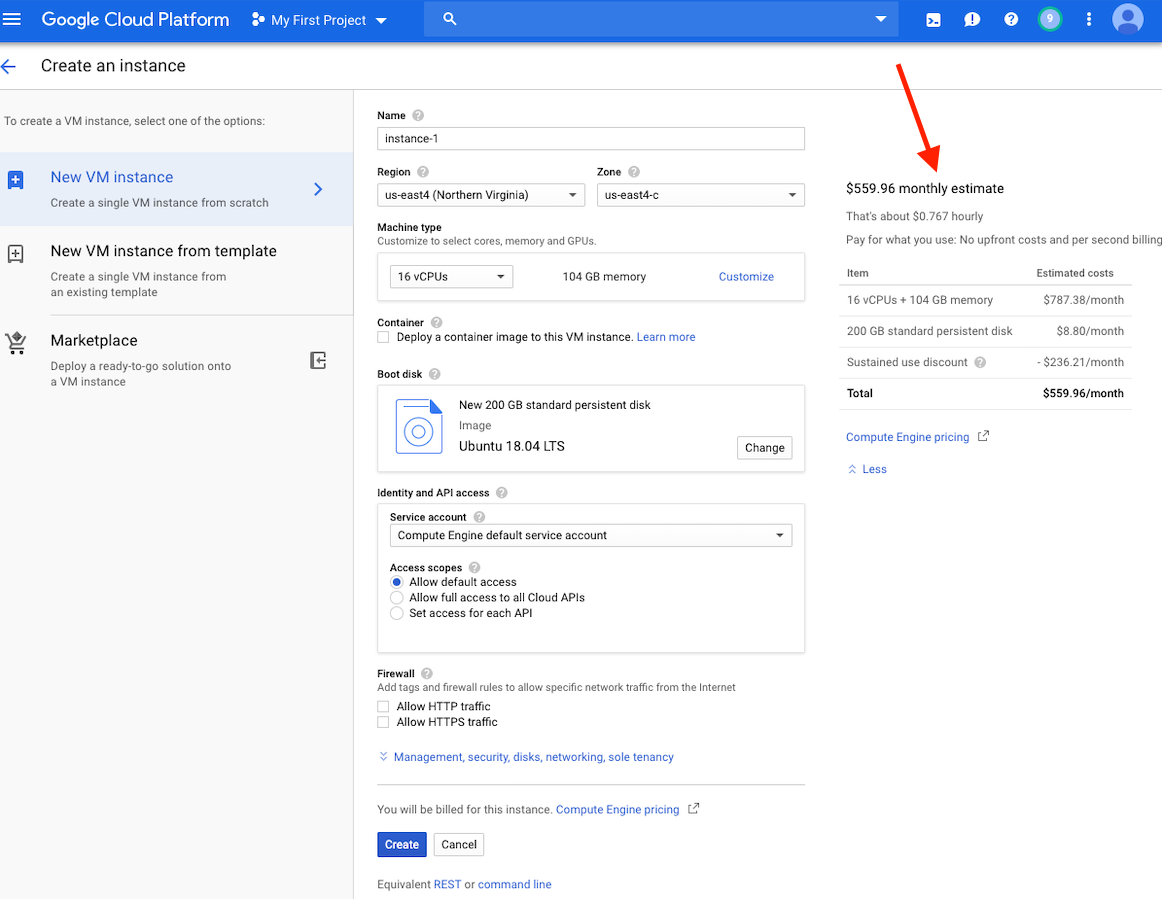
Veuillez noter: la création d'une machine virtuelle dans la même région que le stockage peut fournir de meilleures performances. Nous vous recommandons de créer une machine virtuelle dans la région des États-Unis. Si vous avez un emploi qui prendra plusieurs heures, mais moins de 24 heures, vous pouvez potentiellement profiter des machines virtuelles préemptives.
Des instructions détaillées pour créer un compte GCP et le lancement d'une machine virtuelle peuvent être trouvées ici.
Une fois votre machine virtuelle créée, vous devez y accéder depuis votre ordinateur local. Il existe de nombreuses méthodes pour accéder à votre machine virtuelle, selon les façons dont vous souhaitez l'utiliser. Sur le GCP, le moyen le plus simple est de SSH du navigateur.
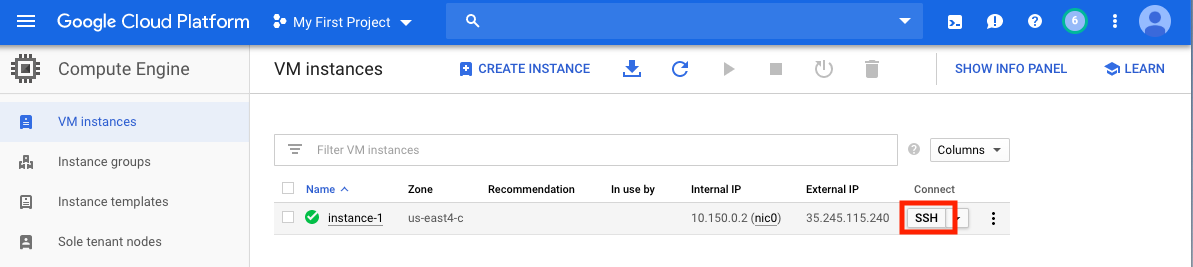
Vous avez maintenant un shell de commande en cours d'exécution et vous êtes prêt à continuer.
N'oubliez pas d'arrêter ou de supprimer la machine virtuelle pour éviter de contracter des coûts supplémentaires.
Dans cette section, nous couvrirons l'installation de Docker, discuterons de diverses options de commande docker run et examinerons la structure d'une commande docker. Nous utiliserons le même petit exemple de la section 1 et explorerons des approches alternatives pour exécuter l'image BLAST + Docker. Cependant, nous utilisons une véritable instance VM, qui offre des performances et des fonctionnalités plus importantes que le shell Google Cloud.
Données d'entrée
Dans un système de production, Docker doit être installé en tant qu'application.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
Pour confirmer l'installation correcte de Docker, exécutez la commande docker run hello-world . Si vous êtes correctement installé, vous devriez voir "Hello from docker! ..." (https://docs.docker.com/samples/library/hello-world/)
Cette section est facultative.
Vous trouverez ci-dessous une liste des options de ligne de commande docker run utilisée dans ce tutoriel.
| Nom, courte (si disponible) | Description |
|---|---|
--rm | Retirez automatiquement le conteneur lorsqu'il sort |
--volume , -v | Lier le montage un volume |
--workdir , -w | Répertoire de travail à l'intérieur du conteneur |
Cette section est facultative.
Pour ce tutoriel, il serait utile de comprendre la structure d'une commande docker. La commande suivante se compose de trois parties.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
La première partie de la commande docker run --rm ncbi/blast est une instruction pour exécuter l'image Docker ncbi/blast et supprimer le conteneur lorsque l'exécution est terminée.
La deuxième partie de la commande rend les données de séquence de requête accessibles dans le conteneur. Docker Bind Mounts utilise -v pour monter les répertoires locaux dans les répertoires à l'intérieur du conteneur et fournir l'autorisation d'accès RW (lire et écrire) ou RO (lire uniquement). Par exemple, en supposant que vos séquences de sujets sont stockées dans le répertoire $ Home / Fasta sur l'hôte local, vous pouvez utiliser le paramètre suivant pour rendre ce répertoire accessible à l'intérieur du conteneur dans / BLAST / FASTA en tant que répertoire en lecture seule -v $HOME/fasta:/blast/fasta:ro . L'indicateur -w /blast/blastdb_custom définit le répertoire de travail à l'intérieur du conteneur.
La troisième partie de la commande est la commande BLAST +. Dans ce cas, il exécute MakeBlastDB pour créer des fichiers de base de données BLAST.
Vous pouvez démarrer une session bash interactive pour cette image en utilisant docker run -it ncbi/blast /bin/bash . Pour l'image BLAST + Docker, les exécutables sont dans le dossier / souffle / bin et / root / edirect et ajoutés à la variable $ chemin.
Pour une documentation supplémentaire sur la commande docker run , veuillez vous référer à la documentation.
Cette section est facultative.
| Commande docker | Description |
|---|---|
docker ps -a | Affiche une liste de conteneurs |
docker rm $(docker ps -q -f status=exited) | Supprime tous les conteneurs sortants, si vous avez au moins 1 conteneur sorti |
docker rm <CONTAINER_ID> | Supprime un conteneur |
docker images | Affiche une liste d'images |
docker rmi <REPOSITORY (IMAGE_NAME)> | Supprime une image |
Cette section est facultative.
Avec cette image Docker, vous pouvez exécuter BLAST + dans un conteneur isolé, facilitant la reproductibilité des résultats de BLAST. En tant qu'utilisateur de cette image Docker, vous devez fournir des bases de données BLAST et des séquences de requête pour exécuter Blast ainsi qu'un emplacement à l'extérieur du conteneur pour enregistrer les résultats. Ce qui suit est une liste des répertoires utilisés par BLAST +. Vous les créerez à l'étape 2.
| Annuaire | But | Notes |
|---|---|---|
$HOME/blastdb | Stores Bases de données BLAST fournies par NCBI- | Si elle est définie sur un seul chemin absolu , la variable d'environnement $BLASTDB pourrait être utilisée à la place (voir Configurer BLAST via des variables d'environnement.) |
$HOME/queries | Stocke les séquences de requête fournies par l'utilisateur | |
$HOME/fasta | Stocke les séquences Fasta fournies par l'utilisateur pour créer une (s) base de données BLAST | |
$HOME/results | Résultats de l'explosion des magasins | Monter avec les autorisations rw |
$HOME/blastdb_custom | Stocke les bases de données de dalm fournies par l'utilisateur |
Cette section est facultative.
La commande suivante affiche la dernière version BLAST.
docker run --rm ncbi/blast blastn -version
L'ajout d'une balise au nom de l'image ( ncbi/blast ) vous permet d'utiliser une version différente de BLAST + (voir la section «Tags pris en charge et notes de version respectives» pour les versions prises en charge).
Différentes versions de BLAST + existent dans différentes images Docker. La commande suivante lancera le téléchargement de l'image docker BLAST + version 2.9.0.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
Par exemple, pour utiliser l'image docker BLAST + Version 2.9.0 au lieu de la dernière version, remplacez la première partie de la commande
docker run --rm ncbi/blast avec docker run --rm ncbi/blast:2.9.0
Cette section est facultative.
Dans cet exemple, nous commencerons par récupérer des séquences de requête et de base de données, puis créerons une base de données BLAST personnalisée.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Pour vérifier la base de données BLAST nouvellement créée ci-dessus, vous pouvez exécuter la commande suivante pour afficher les accessions, la longueur de séquence et le nom commun des séquences dans la base de données.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
En tant qu'alternative, vous pouvez également télécharger des bases de données BLAST préformattées à partir de NCBI ou du seau de stockage Google NCBI.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
Pour une description détaillée de update_blastdb.pl , veuillez vous référer à la documentation. Par défaut, update_blastdb.pl téléchargera à partir du cloud à condition que vous soyez connecté, ou depuis NCBI si vous n'utilisez pas de fournisseur de cloud pris en charge.
Cette section est facultative.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Cette section est facultative.
La commande ci-dessous monte le chemin $HOME/blastdb sur la machine locale AS /blast/blastdb sur le conteneur, et blastdbcmd affiche les bases de données BLAST disponibles à cet emplacement.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Vous devriez voir une /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
Vous devriez voir une /blast/blastdb_custom/nurse-shark-proteins Protein .
Lors de l'exécution de Blast dans un conteneur Docker, notez les supports spécifiés à la commande docker run pour rendre l'entrée et les sorties accessibles. Dans les exemples ci-dessous, les deux premiers supports donnent accès aux bases de données BLAST, le troisième support donne accès à la ou des séquences de requête, et le quatrième support fournit un répertoire pour enregistrer les résultats. (Notez les options :ro et :rw , qui montent respectivement les répertoires en lecture seule et en lecture.)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
À ce stade, vous devriez voir le fichier de sortie $HOME/results/blastp.out . Avec votre requête, BLAST a identifié la séquence protéique P80049.1 comme un match avec un score de 14,2 et une valeur électronique de 0,96. Pour afficher le contenu de ce fichier de sortie, utilisez la commande more $HOME/results/blastp.out .
N'oubliez pas d'arrêter ou de supprimer la machine virtuelle pour éviter de contracter des coûts supplémentaires. Vous pouvez le faire sur la console GCP comme indiqué ci-dessous. 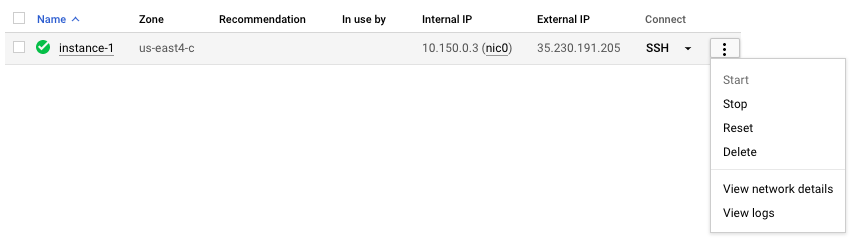
L'une des promesses du cloud computing est l'évolutivité. Dans cette section, nous montrerons comment utiliser l'image BLAST + Docker à l'échelle de production sur la plate-forme Google Cloud. Nous effectuerons une analyse BLAST similaire à l'approche décrite dans cette publication pour comparer les contigs alignés de novo du séquençage bactérien 16S-23 contre la base de données de collection de nucléotides (NT).
Pour tester l'évolutivité, nous utiliserons des entrées de différentes tailles pour estimer la durée de téléchargement de la base de données de collection de nucléotides et d'exécuter la recherche BLAST en utilisant la dernière version de l'image BLAST + Docker. Les résultats attendus sont résumés dans les tableaux suivants.
Fichiers d'entrée: 28 échantillons (fichiers multi-fasta) contenant des contigs alignés de novo de la publication.
(Les instructions pour télécharger et créer les fichiers d'entrée sont décrites dans le bloc de code ci-dessous.)
Base de données: Base de données pré-formatée de la collection de nucléotides Blast, version 5 (NT): 68.7217 Go (à partir de mai 2019)
| Nom de fichier d'entrée | Déposer un contenu | Taille de fichier | Nombre de séquences | Nombre de nucléotides | Taille de sortie attendue | |
|---|---|---|---|---|---|---|
| Analyse 1 | query1.fa | uniquement échantillon 1 | 59 Ko | 121 | 51 119 | 3,1 Go |
| Analyse 2 | Query5.fa | uniquement les échantillons 1-5 | 422 kb | 717 | 375 154 | 10,4 Go |
| Analyse 3 | query.fa | tous les 28 échantillons | 2,322 Mb | 3798 | 2 069 892 | 47,8 Go |
| Type / zone de machine virtuelle | Processeur | Mémoire (GB) | Coût horaire * | Télécharger NT (min) | Analyse 1 (min) | Analyse 2 (min) | Analyse 3 (min) | Coût total ** |
|---|---|---|---|---|---|---|---|---|
| N1-standard-8 US-East4c | 8 | 30 | 0,312 $ | 9 | 22 | - | - | - |
| N1-standard-16 US-East4c | 16 | 60 | 0,611 $ | 9 | 14 | 53 | 205 | 2,86 $ |
| N1-Highmem-16 US-East4c | 16 | 104 | 0,767 $ | 9 | 9 | 30 | 143 | 2,44 $ |
| N1-Highmem-16 US-West2a | 16 | 104 | 0,809 $ | 11 | 9 | 30 | 147 | 2,60 $ |
| N1-Highmem-16 US-West1b | 16 | 104 | 0,674 $ | 11 | 9 | 30 | 147 | 2,17 $ |
| Site Web BLAST (BLASTN) | - | - | - | - | Les recherches dépassent les restrictions actuelles sur l'utilisation | Les recherches dépassent les restrictions actuelles sur l'utilisation | Les recherches dépassent les restrictions actuelles sur l'utilisation | - |
Toutes les instances GCP sont configurées avec 200 Go de disque standard persistant.
* Les coûts horaires ont été fournis par Google Cloud Platform (mai 2019) lorsque les machines virtuelles ont été créées et sont sujettes à modification.
** Les coûts totaux ont été estimés en utilisant le coût horaire et le temps total pour télécharger NT et exécuter l'analyse 1, l'analyse 2 et l'analyse 3. Les estimations sont utilisées à titre de comparaison uniquement; Vos coûts peuvent varier et sont à votre responsabilité de surveiller et de gérer.
Veuillez vous référer à GCP pour plus d'informations sur les types de machines, les régions et les zones et calculer les coûts.
Veuillez noter que l'exécution du binaire blastn sans spécifier son paramètre -task invoque l'algorithme Megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
Vous avez terminé l'intégralité du tutoriel. À ce stade, si vous n'avez pas besoin des données téléchargées pour une analyse plus approfondie, veuillez supprimer la machine virtuelle pour éviter de contracter des coûts supplémentaires.
Pour supprimer une instance, suivez les instructions dans la section Arrêtez l'instance GCP.
Pour plus d'informations, veuillez consulter la documentation de Google Cloud Platform sur le cycle de vie des instances.
Pour exécuter ces exemples, vous aurez besoin d'un compte Amazon Web Services (AWS). Si vous n'en avez pas déjà un, vous pouvez créer un compte qui offre la possibilité d'explorer et d'essayer gratuitement les services AWS aux limites spécifiées pour chaque service. Pour commencer, visitez le site de niveau gratuit, cela nécessitera une carte de crédit valide, mais elle ne sera pas facturée si vous calculez le niveau gratuit. Lorsque vous choisissez un produit de niveau gratuit, assurez-vous qu'il se trouve dans la catégorie de produits Compute .
Ces instructions créent une machine virtuelle EC2 basée sur une image Amazon Machine (AMI) qui inclut Docker et ses dépendances.
Avec la machine virtuelle créée, vous y accédez depuis votre ordinateur local à l'aide de SSH. Votre fichier de paire de clés / .pem sert de compensation.
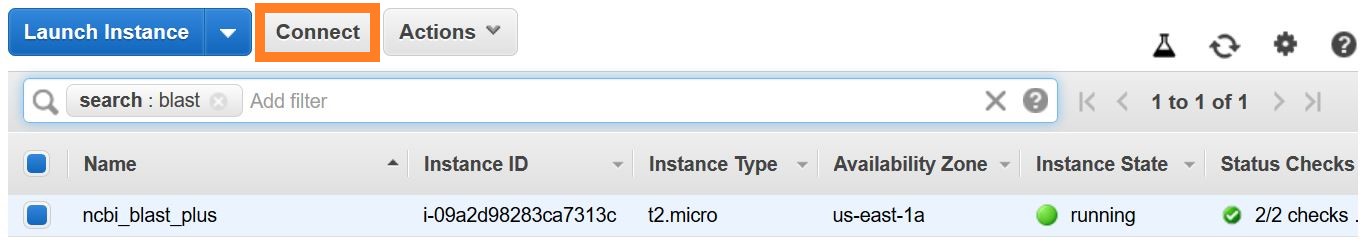
Il existe plusieurs façons d'établir une connexion SSH. Dans la liste des instances EC2 dans la console AWS, sélectionnez Connect , puis suivez les instructions de la méthode de connexion un client SSH autonome .
Les instructions détaillées pour se connecter à une machine virtuelle Linux peuvent être trouvées ici.
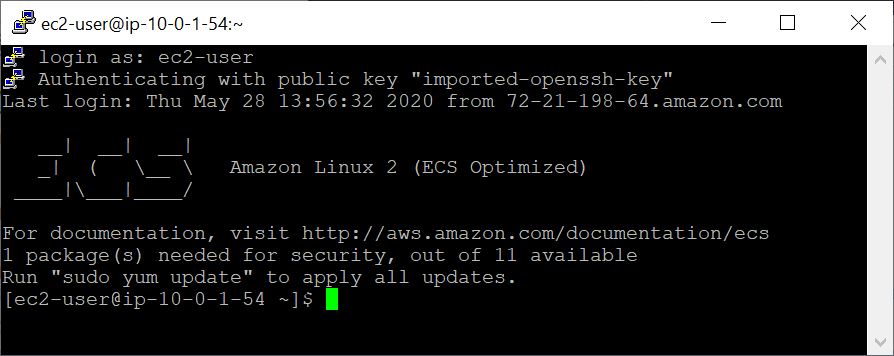
Spécifiez EC2-User comme nom d'utilisateur, au lieu de root dans votre ligne de commande SSH ou lorsque vous vous êtes invité à se connecter, spécifiez EC2-User comme nom d'utilisateur.
Dans cet exemple, nous commencerons par récupérer des séquences de requête et de base de données, puis créerons une base de données BLAST personnalisée.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Pour vérifier la base de données BLAST nouvellement créée ci-dessus, vous pouvez exécuter la commande suivante pour afficher les accessions, la longueur de séquence et le nom commun des séquences dans la base de données.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Lors de l'exécution de l'explosion dans un conteneur Docker, notez l'option Mounts (option -v ) spécifiée à la commande docker run pour rendre l'entrée et les sorties accessibles. Dans les exemples ci-dessous, les deux premiers supports donnent accès aux bases de données BLAST, le troisième support donne accès à la ou des séquences de requête, et le quatrième support fournit un répertoire pour enregistrer les résultats. (Notez les options :ro et :rw , qui montent respectivement les répertoires en lecture seule et en lecture.)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
À ce stade, vous devriez voir le fichier de sortie $HOME/results/blastp.out . Avec votre requête, BLAST a identifié la séquence protéique P80049.1 comme un match avec un score de 14,2 et une valeur électronique de 0,96. Pour afficher le contenu de ce fichier de sortie, utilisez la commande more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
La sortie attendue est une liste de DBS BLAST, y compris leur nom, leur description, sa taille et leur dernière date mise à jour.
Pour une description détaillée de update_blastdb.pl , veuillez vous référer à la documentation. Par défaut, update_blastdb.pl téléchargera à partir du cloud à condition que vous soyez connecté, ou depuis NCBI si vous n'utilisez pas de fournisseur de cloud pris en charge.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
La sortie attendue est une liste des noms de Blast DBS.
N'oubliez pas d'arrêter ou de résilier la machine virtuelle pour éviter de contracter des coûts supplémentaires. Vous pouvez le faire à partir de la liste d'instances EC2 dans la console AWS comme indiqué ci-dessous.
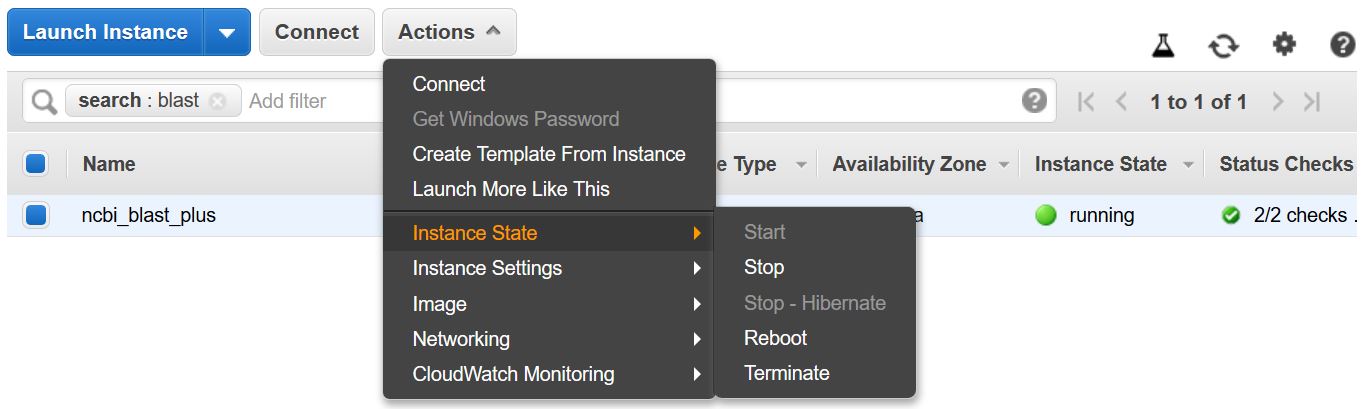
Cet exemple nécessite un hôte multi-fond. En tant que tels, les frais de calcul EC2 seront réalisés en exécutant cet exemple. Le taux actuel pour le type d'instance utilisé - T2.Large - est de 0,093 $ / h.
Ces instructions créent une machine virtuelle EC2 basée sur une image Amazon Machine (AMI) qui inclut Docker et ses dépendances.
Avec la machine virtuelle créée, vous y accédez depuis votre ordinateur local à l'aide de SSH. Votre fichier de paire de clés / .pem sert de compensation.
Il existe plusieurs façons d'établir une connexion SSH. Dans la liste des instances EC2 dans la console AWS, sélectionnez Connect , puis suivez les instructions de la méthode de connexion un client SSH autonome .
Les instructions détaillées pour se connecter à une machine virtuelle Linux peuvent être trouvées ici.
Spécifiez EC2-User comme nom d'utilisateur, au lieu de root dans votre ligne de commande SSH ou lorsque vous vous êtes invité à se connecter, spécifiez EC2-User comme nom d'utilisateur.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
La commande ci-dessous monte (en utilisant l'option -v ) le chemin $HOME/blastdb sur la machine locale AS /blast/blastdb sur le conteneur, et blastdbcmd affiche les bases de données BLAST disponibles à cet endroit.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Vous devriez voir une /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
À ce stade, vous devriez voir le fichier de sortie $HOME/results/blastp_pdbaa.out . Pour afficher le contenu de ce fichier de sortie, utilisez la commande more $HOME/results/blastp_pdbaa.out .
Une façon de transférer des fichiers entre votre ordinateur local et une instance Linux consiste à utiliser le protocole de copie sécurisé (SCP).
La section transférant des fichiers vers les instances Linux à partir de Linux à l'aide de SCP du Guide de l'utilisateur Amazon EC2 pour les instances Linux fournit des instructions détaillées pour ce processus.
Le NCBI héberge les mêmes bases de données sur AWS, GCP et le site NCBI FTP. Le tableau ci-dessous a la liste des bases de données à jour en novembre 2022.
Il est également possible d'obtenir la liste actuelle avec la commande:
Docker Run --RM NCBI / BLAST UPDATE_BLASTDB.PL --SHOWALL Pretty
ou
Update_blastdb.pl --showall Pretty # après avoir téléchargé le package BLAST +.
Comme indiqué ci-dessus, Update_blastdb.pl peut également être utilisé pour télécharger ces bases de données. Il sélectionnera automatiquement la ressource appropriée (par exemple, GCP si vous êtes au sein de ce fournisseur).
Ces bases de données peuvent également être recherchées avec ElasticBlast sur GCP et AWS.
L'accès aux bases de données sur AWS ou GCP en dehors du fournisseur de cloud entraînera probablement des frais de sortie sur votre compte. Si vous n'êtes pas sur le fournisseur de cloud, vous devez utiliser les bases de données sur le site NCBI FTP.
| Nom | Taper | Titre |
|---|---|---|
| 16S_RIBOSOMAL_RNA | ADN | ARN ribosomal 16S (souches de type bactéries et archées) |
| 18S_Fungal_Sequences | ADN | Séquences d'ARN ribosomales 18S (SSU) à partir de type champignons et de matériau de référence |
| 28S_FUNGAL_SERDENCES | ADN | Séquences d'ARN ribosomales 28S (LSU) à partir de type champignon et de matériau de référence |
| Bêtacoronavirus | ADN | Bêtacoronavirus |
| Gcf_000001405.38_top_level | ADN | Homo sapiens grch38.p12 [gcf_000001405.38] chromosomes plus échafaudages non placés et illocalisés |
| Gcf_000001635.26_top_level | ADN | Mus musculus grcm38.p6 [gcf_000001635.26] chromosomes plus échafaudages non placés et illocalisés |
| Its_refseq_fungi | ADN | Région interne de l'espaceur transcrit (ITS) à partir de type champignons et de matériau de référence |
| Its_eukaryote_ sequences | ADN | Son explosion eucaryote |
| LSU_EUKARYOTE_RRNA | ADN | Grande sous-unité acide nucléique ribosomal pour les eucaryotes |
| LSU_PROKARYOTE_RNNA | ADN | Grande sous-unité acide nucléique ribosomal pour les procaryotes |
| SSU_EUKARYOTE_RRNA | ADN | Petite sous-unité acide nucléique ribosomal pour les eucaryotes |
| Env_nt | ADN | échantillons environnementaux |
| NT | ADN | Collection de nucléotides (NT) |
| patnt | ADN | Séquences nucléotidiques dérivées de la division des brevets de Genbank |
| pdbnt | ADN | Base de données de nucléotides PDB |
| ref_euk_rep_genomes | ADN | Base de données RefSeq Eukaryotic Reprewinging Genome |
| Ref_prok_rep_genomes | ADN | RefSeq Prokaryote Représentant Genomes (contient RefSeq Assembly) |
| Ref_viroid_rep_genomes | ADN | Genomes représentatifs RefSeq Viroids |
| Ref_virus_rep_genomes | ADN | RefSeq Virus Genomes représentatifs |
| RefSeq_RNA | ADN | Séquences de référence de transcription NCBI |
| RefSeq_Select_RNA | ADN | RefSeq Select ARN séquences |
| tsa_nt | ADN | Séquences d'assemblage de fusil de chasse du transcriptome (TSA) |
| Env_nr | Protéine | Protéines des projets métagénomiques WGS |
| point de repère | Protéine | Base de données historique pour SmartBlast |
| NR | Protéine | Toutes les traductions CDS Genbank non redondantes + PDB + Swissprot + PIR + PRF excluant les échantillons environnementaux de projets WGS |
| PDBAA | Protéine | Base de données de protéines PDB |
| pataa | Protéine | Séquences protéiques dérivées de la division des brevets de Genbank |
| RefSeq_protein | Protéine | Séquences de référence de la protéine NCBI |
| RefSeq_Select_Prot | Protéine | RefSeq Select Proteins |
| swissprot | Protéine | Séquences uniprotkb / swissprot non redondantes |
| tsa_nr | Protéine | Séquences d'assemblage de fusil de chasse du transcriptome (TSA) |
| cdd | Protéine | La base de données de domaine conservée (CDD) est une collection de modèles d'alignement de séquences multiples bien annulés repris sous forme de score spécifique à la position |
Le NCBI fournit des métadonnées pour les bases de données BLAST disponibles sur AWS, GCP et le site NCBI FTP.
L'accès aux bases de données sur AWS ou GCP en dehors du fournisseur de cloud entraînera probablement des frais de sortie sur votre compte. Si vous n'êtes pas sur le fournisseur de cloud, vous devez utiliser les bases de données sur le site NCBI FTP.
Sur AWS et GCP, le fichier est dans un sous-répertoire dépendant de la date avec les bases de données. Pour trouver le dernier sous-répertoire valide, lisez d'abord s3://ncbi-blast-databases/latest-dir (sur AWS) ou gs://blast-db/latest-dir (sur GCP). latest-dir est un fichier texte avec un tampon de date (par exemple, 2020-09-29-01-05-01) spécifiant le répertoire le plus récent. Le répertoire approprié sera le URI de base AWS ou GCP pour les bases de données BLAST (par exemple, s3://ncbi-blast-databases/ pour AWS) plus le texte dans le fichier latest-dir . Un exemple URI, dans AWS, serait s3://ncbi-blast-databases/2020-09-29-01-05-01 . L'URI GCP serait similaire.
Un extrait à partir d'un fichier de métadonnées est illustré ci-dessous. La plupart des champs ont des significations évidentes. Les fichiers comprennent la base de données BLAST. Le champ bytes-total représente la taille totale de la base de données BLAST dans les octets et est destiné à spécifier la quantité d'espace disque requise.
L'exemple ci-dessous provient de AWS, mais les fichiers de métadonnées sur GCP ont le même format. Les bases de données sur le site FTP sont dans des tarfiles gzippés, un par volume de la base de données BLAST, donc ceux-ci sont répertoriés plutôt que les fichiers individuels.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
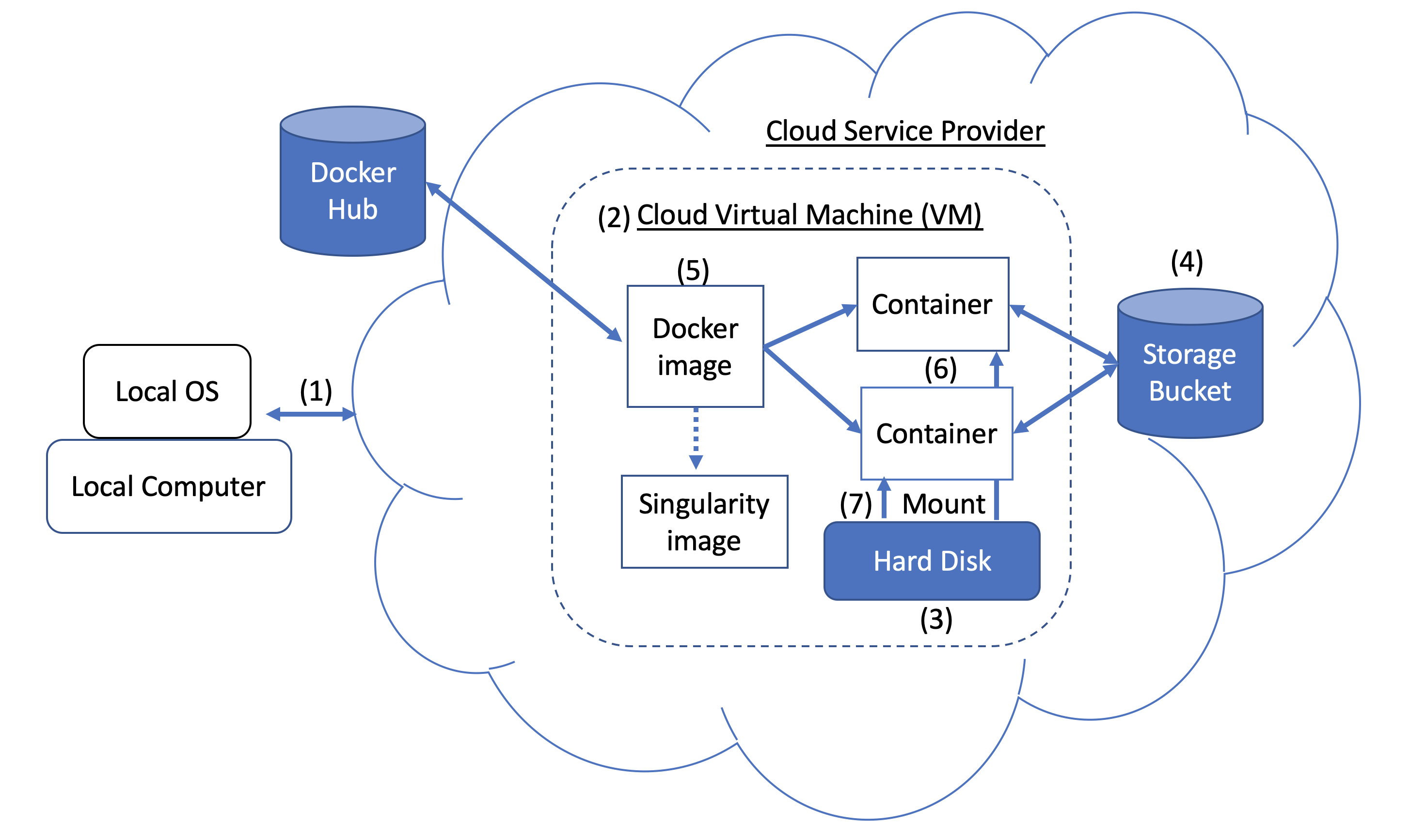 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.