Repositori ini berisi dokumentasi untuk aplikasi NCBI Blast+ Command Line dalam gambar Docker. Kami akan menunjukkan cara menggunakan gambar Docker untuk menjalankan analisis BLAST di Google Cloud Platform (GCP) dan Amazon Web Services (AWS) menggunakan contoh dasar kecil dan contoh tingkat produksi yang lebih canggih. Beberapa pengetahuan dasar dari perintah UNIX/Linux dan Blast+ berguna dalam menyelesaikan tutorial ini.
run opsi perintahrun struktur perintah
Pusat Informasi Bioteknologi Nasional (NCBI) Alat Pencarian Alignment Lokal Dasar (BLAST) menemukan daerah kesamaan lokal antara urutan. Program ini membandingkan urutan nukleotida atau protein dengan database urutan dan menghitung signifikansi statistik kecocokan. BLAST dapat digunakan untuk menyimpulkan hubungan fungsional dan evolusi antara sekuens serta membantu mengidentifikasi anggota keluarga gen.
Diperkenalkan pada tahun 2009, BLAST+ adalah versi yang lebih baik dari aplikasi baris perintah BLAST. Untuk deskripsi lengkap tentang fitur dan kemampuan BLAST+, silakan merujuk ke manual pengguna Line Command Line.
Cloud Computing menawarkan penghematan biaya potensial dengan menggunakan sumber daya komputasi sesuai permintaan, scalable, dan elastis. Sementara deskripsi terperinci tentang berbagai teknologi dan manfaat cloud berada di luar ruang lingkup untuk repositori ini, bagian berikut berisi informasi yang diperlukan untuk mulai menjalankan gambar Blast+ Docker di Google Cloud Platform (GCP).
Docker adalah alat untuk melakukan virtualisasi tingkat sistem operasi menggunakan wadah perangkat lunak. Dalam Teknologi Kontainerisasi * , gambar adalah snapshot dari lingkungan analitik yang merangkum aplikasi dan dependensi. Gambar, yang pada dasarnya adalah file yang dibangun dari daftar instruksi, dapat disimpan dan dengan mudah dibagikan untuk orang lain untuk menciptakan kembali lingkungan analitik yang tepat di seluruh platform dan sistem operasi. Wadah adalah instance runtime dari suatu gambar. Dengan menggunakan kontainerisasi, pengguna dapat melewati langkah-langkah yang sering rumit dalam menyusun, mengkonfigurasi, dan menginstal alat berbasis UNIX seperti Blast+. Selain portabilitas, kontainerisasi adalah pendekatan yang ringan untuk membuat analisis lebih dapat ditemukan, dapat diakses, dapat dioperasikan, dapat digunakan kembali (adil) dan, pada akhirnya, dapat direproduksi.
*Ada banyak alat dan standar kontainerisasi, seperti Docker dan Singularity. Kami hanya akan fokus pada Docker, yang dianggap sebagai standar de facto oleh banyak orang di lapangan.
Bagian berikut termasuk instruksi untuk membuat mesin virtual Google, menginstal Docker, dan menjalankan perintah Blast+ menggunakan gambar Docker.
Bagian ini memberikan run-through cepat dari analisis BLAST di lingkungan Docker pada instance Google. Ini dimaksudkan sebagai tinjauan umum bagi mereka yang hanya menginginkan pemahaman tentang prinsip -prinsip solusi. Jika Anda bekerja dengan instance Amazon, silakan buka bagian Pengaturan Layanan Web Amazon dari dokumentasi ini. Google Cloud Shell, lingkungan shell interaktif, akan digunakan untuk contoh ini, yang memungkinkan untuk menjalankan contoh kecil berikut tanpa harus melakukan pengaturan tambahan, seperti membuat akun penagihan atau menghitung instance. Deskripsi yang lebih rinci tentang langkah -langkah analisis, perintah alternatif, dan topik yang lebih canggih dibahas di bagian selanjutnya dari dokumentasi ini.
Persyaratan: Akun Google
Aliran tugas: 
Input Data:
Pertama, di jendela atau tab browser terpisah, masuk di https://console.cloud.google.com/
Klik tombol Activate Cloud Shell di sudut kanan atas konsol Google Cloud Platform. 
Anda sekarang akan melihat jendela sesi cloud shell Anda: 
Langkah selanjutnya adalah menyalin-dan-menempel perintah di bawah ini di sesi cloud shell Anda.
Harap dicatat: di GitHub Anda dapat menggunakan mouse Anda untuk menyalin; Namun, dalam cangkang perintah Anda harus menggunakan keyboard Anda. Di Windows atau Unix/Linux, gunakan Control+C untuk menyalin dan Control+V untuk menempel. Pada macOS, gunakan Command+C untuk menyalin dan Command+V untuk menempel.
Untuk menggulir di cloud shell, aktifkan scrollbar di Terminal settings dengan ikon kunci pas. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
Pada titik ini, Anda akan melihat output di layar. Dengan kueri Anda, Blast mengidentifikasi urutan protein P80049.1 sebagai kecocokan dengan skor 14,2 dan nilai-E 0,96.
Untuk analisis yang lebih besar, disarankan untuk menggunakan flag -out untuk menyimpan output ke file. Misalnya, tambahkan -out /blast/results/blastp.out ke perintah terakhir pada langkah 3 di atas dan lihat konten file output ini menggunakan more $HOME/results/blastp.out .
Anda juga dapat menanyakan P01349.FSA terhadap PDB seperti yang ditunjukkan pada blok kode berikut.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
Anda sekarang telah menyelesaikan tugas sederhana dan melihat bagaimana Blast+ dengan Docker bekerja. Untuk mempelajari tentang Docker dan Blast+ pada skala produksi, silakan lanjutkan ke bagian berikutnya.
Di Bagian 2-Panduan langkah demi langkah menggunakan gambar Blast+ Docker, kami akan menggunakan contoh kecil yang sama dari bagian sebelumnya dan membahas pendekatan alternatif, tambahan Docker dan perintah BLAST+ tambahan, dan opsi dan struktur perintah Docker. Di Bagian 3, kami akan menunjukkan cara menjalankan gambar Blast+ Docker pada skala produksi.
Pertama, Anda perlu mengatur mesin virtual Google Cloud Platform (GCP) untuk analisis.
GCP saat ini menawarkan kredit $ 300, yang berakhir 12 bulan dari aktivasi, untuk memberi insentif kepada pengguna cloud baru. Langkah -langkah berikut akan menunjukkan kepada Anda cara mengaktifkan kredit ini. Anda akan diminta untuk informasi penagihan, tetapi GCP tidak akan menagih otomatis Anda setelah uji coba berakhir; Anda harus memilih untuk meningkatkan secara manual ke akun berbayar.
Setelah masuk, klik Aktifkan untuk mengaktifkan kredit $ 300. 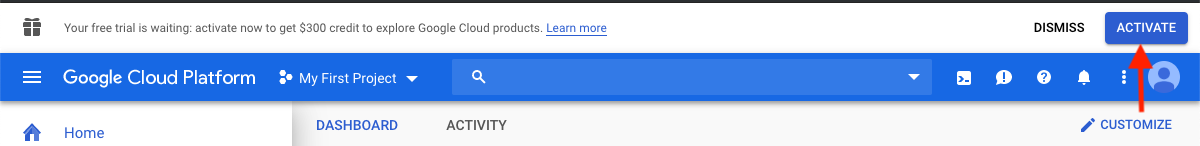
Masukkan negara Anda, misalnya, Amerika Serikat, dan centang kotak yang menunjukkan bahwa Anda telah membaca dan menerima ketentuan layanan.
Di bawah "Jenis Akun," pilih "Individu." (Ini mungkin dipilih sebelumnya di akun Google Anda)
Masukkan nama dan alamat Anda.
Di bawah "Bagaimana Anda Membayar," pilih "Pembayaran Otomatis." (Ini mungkin telah dipilih sebelumnya di akun Google Anda) Ini menunjukkan bahwa Anda akan membayar biaya setelah Anda menggunakan layanan ini, baik ketika Anda telah mencapai ambang tagihan Anda atau setiap 30 hari, mana yang lebih dulu.
Di bawah "Metode Pembayaran," pilih "Tambahkan kartu kredit atau debit" dan masukkan informasi kartu kredit Anda. Anda tidak akan dikenakan biaya secara otomatis setelah uji coba berakhir. Anda harus memilih untuk meningkatkan ke akun berbayar sebelum metode pembayaran Anda akan dibebankan.
Klik "Mulai Uji Coba Gratis Saya" untuk menyelesaikan pendaftaran. Ketika proses ini selesai, Anda akan melihat layar sambutan GCP.
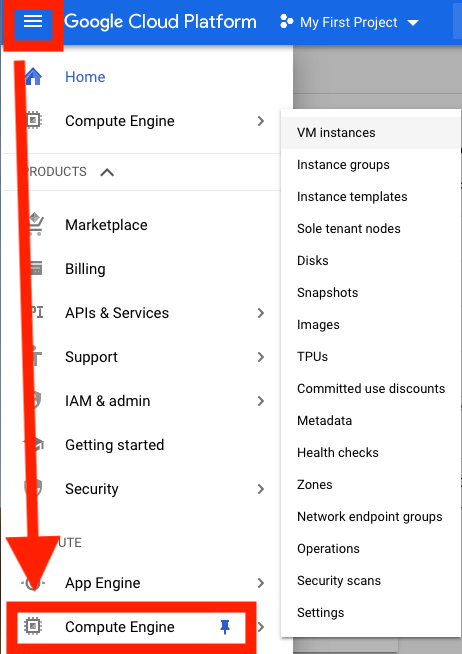
Pada titik ini, Anda akan melihat perkiraan biaya untuk contoh ini di sisi kanan jendela Anda. 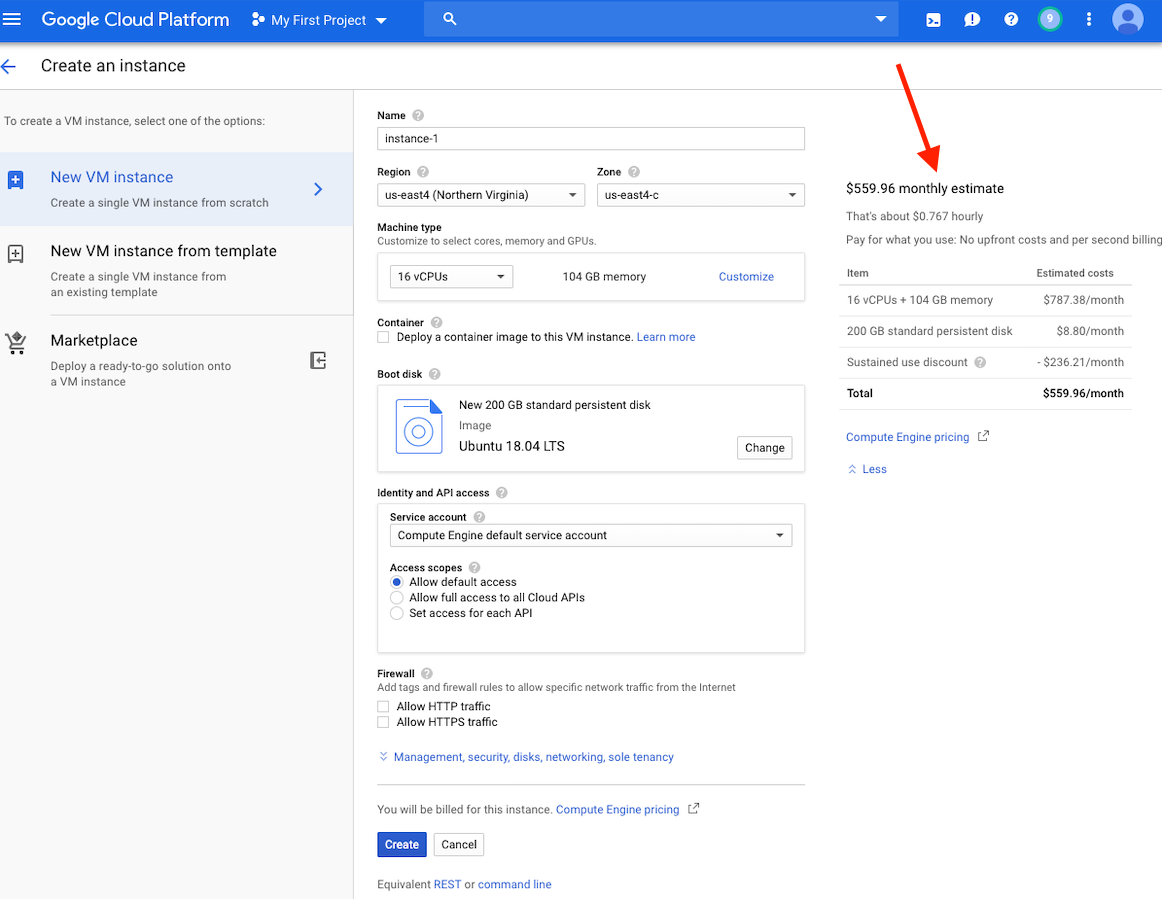
Harap dicatat: Membuat VM di wilayah yang sama dengan penyimpanan dapat memberikan kinerja yang lebih baik. Kami merekomendasikan membuat VM di wilayah AS-EAST4. Jika Anda memiliki pekerjaan yang akan memakan waktu beberapa jam, tetapi kurang dari 24 jam, Anda berpotensi memanfaatkan VM preemptible.
Instruksi terperinci untuk membuat akun GCP dan meluncurkan VM dapat ditemukan di sini.
Setelah VM Anda dibuat, Anda harus mengaksesnya dari komputer lokal Anda. Ada banyak metode untuk mengakses VM Anda, tergantung pada cara Anda ingin menggunakannya. Di GCP, cara yang paling mudah adalah dengan SSH dari browser.
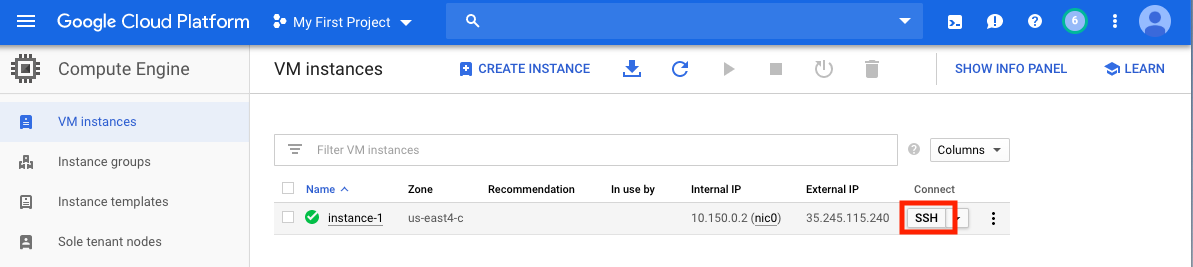
Anda sekarang memiliki cangkang perintah yang berjalan dan Anda siap untuk melanjutkan.
Ingatlah untuk menghentikan atau menghapus VM untuk mencegah biaya tambahan.
Di bagian ini, kami akan mencakup instalasi Docker, membahas berbagai opsi perintah docker run , dan memeriksa struktur perintah Docker. Kami akan menggunakan contoh kecil yang sama dari Bagian 1 dan mengeksplorasi pendekatan alternatif dalam menjalankan gambar Blast+ Docker. Namun, kami menggunakan instance VM nyata, yang memberikan kinerja dan fungsionalitas yang lebih besar daripada Google Cloud Shell.
Data masukan
Dalam sistem produksi, Docker harus diinstal sebagai aplikasi.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
Untuk mengonfirmasi pemasangan Docker yang benar, jalankan perintah docker run hello-world . Jika diinstal dengan benar, Anda akan melihat "Halo dari Docker! ..." (https://docs.docker.com/samples/library/hello-world/)
Bagian ini opsional.
Di bawah ini adalah daftar opsi baris perintah docker run yang digunakan dalam tutorial ini.
| Nama, tangan pendek (jika tersedia) | Keterangan |
|---|---|
--rm | Secara otomatis lepaskan wadah saat keluar |
--volume , -v | Mengikat volume |
--workdir , -w | Direktori Bekerja di dalam wadah |
Bagian ini opsional.
Untuk tutorial ini, akan berguna untuk memahami struktur perintah Docker. Perintah berikut terdiri dari tiga bagian.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Bagian pertama dari Command docker run --rm ncbi/blast adalah instruksi untuk menjalankan gambar Docker ncbi/blast dan menghapus wadah saat menjalankan selesai.
Bagian kedua dari perintah membuat data urutan kueri dapat diakses dalam wadah. Docker Bind Mounts menggunakan -v untuk memasang direktori lokal ke direktori di dalam wadah dan memberikan izin akses RW (baca dan tulis) atau ro (hanya baca). Misalnya, dengan asumsi urutan subjek Anda disimpan di direktori $ home/fasta pada host lokal, Anda dapat menggunakan parameter berikut untuk membuat direktori itu dapat diakses di dalam wadah di/blast/fasta sebagai direktori read -only -v $HOME/fasta:/blast/fasta:ro . Bendera -w /blast/blastdb_custom mengatur direktori kerja di dalam wadah.
Bagian ketiga dari perintah adalah perintah blast+. Dalam hal ini, ia menjalankan MakeblastDB untuk membuat file database BLAST.
Anda dapat memulai sesi bash interaktif untuk gambar ini dengan menggunakan docker run -it ncbi/blast /bin/bash . Untuk gambar Blast+ Docker, executable berada di folder /blast /bin dan /root /edirect dan ditambahkan ke variabel $ path.
Untuk dokumentasi tambahan pada perintah docker run , silakan merujuk ke dokumentasi.
Bagian ini opsional.
| Perintah Docker | Keterangan |
|---|---|
docker ps -a | Menampilkan daftar kontainer |
docker rm $(docker ps -q -f status=exited) | Menghapus semua wadah keluar, jika Anda memiliki setidaknya 1 wadah keluar |
docker rm <CONTAINER_ID> | Menghapus wadah |
docker images | Menampilkan daftar gambar |
docker rmi <REPOSITORY (IMAGE_NAME)> | Menghapus gambar |
Bagian ini opsional.
Dengan gambar Docker ini Anda dapat menjalankan Blast+ dalam wadah yang terisolasi, memfasilitasi reproduktifitas hasil ledakan. Sebagai pengguna gambar Docker ini, Anda diharapkan menyediakan basis data BLAST dan urutan kueri untuk menjalankan Blast serta lokasi di luar wadah untuk menyimpan hasilnya. Berikut ini adalah daftar direktori yang digunakan oleh Blast+. Anda akan membuatnya di Langkah 2.
| Direktori | Tujuan | Catatan |
|---|---|---|
$HOME/blastdb | Menyimpan database BLAST yang disediakan NCBI | Jika diatur ke jalur tunggal, absolut , variabel lingkungan $BLASTDB dapat digunakan sebagai gantinya (lihat Mengkonfigurasi BLAST melalui variabel lingkungan.) |
$HOME/queries | Menyimpan Urutan Kueri yang Disediakan Pengguna | |
$HOME/fasta | Menyimpan Urutan FASTA yang Disediakan Pengguna untuk Membuat Basis Data BLAST | |
$HOME/results | Stores Hasil Blast | Dipasang dengan izin rw |
$HOME/blastdb_custom | Menyimpan basis data BLAST yang disediakan pengguna |
Bagian ini opsional.
Perintah berikut menampilkan versi BLAST terbaru.
docker run --rm ncbi/blast blastn -version
Menambahkan tag ke nama gambar ( ncbi/blast ) memungkinkan Anda untuk menggunakan versi BLAST+ yang berbeda (lihat “Tag yang Didukung dan Catatan Rilis masing -masing” untuk versi yang didukung).
Versi BLAST+ yang berbeda ada dalam gambar Docker yang berbeda. Perintah berikut akan memulai pengunduhan gambar BLAST+ Versi 2.9.0 Docker.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
Misalnya, untuk menggunakan gambar BLAST+ versi 2.9.0 Docker alih -alih versi terbaru, ganti bagian pertama dari perintah
docker run --rm ncbi/blast dengan docker run --rm ncbi/blast:2.9.0
Bagian ini opsional.
Dalam contoh ini, kita akan mulai dengan mengambil urutan kueri dan basis data dan kemudian membuat database BLAST khusus.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Untuk memverifikasi basis data BLAST yang baru dibuat di atas, Anda dapat menjalankan perintah berikut untuk menampilkan aksesi, panjang urutan, dan nama umum dari urutan dalam database.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Sebagai alternatif, Anda juga dapat mengunduh basis data BLAST yang telah diformat sebelumnya dari NCBI atau NCBI Google Storage Bucket.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
Untuk deskripsi terperinci tentang update_blastdb.pl , silakan merujuk ke dokumentasi. Secara default update_blastdb.pl akan mengunduh dari cloud asalkan Anda terhubung, atau dari NCBI jika Anda tidak menggunakan penyedia cloud yang didukung.
Bagian ini opsional.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Bagian ini opsional.
Perintah di bawah ini memasang jalur $HOME/blastdb pada mesin lokal sebagai /blast/blastdb pada wadah, dan blastdbcmd menunjukkan database BLAST yang tersedia di lokasi ini.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Anda akan melihat /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
Anda akan melihat /blast/blastdb_custom/nurse-shark-proteins Protein .
Saat menjalankan BLAST dalam wadah Docker, perhatikan dudukan yang ditentukan untuk perintah docker run untuk membuat input dan output dapat diakses. Dalam contoh di bawah ini, dua dudukan pertama menyediakan akses ke basis data BLAST, dudukan ketiga menyediakan akses ke urutan kueri, dan dudukan keempat menyediakan direktori untuk menyimpan hasilnya. (Perhatikan :ro dan :rw Opsi, yang masing-masing memasang direktori sebagai baca dan baca-tulis.)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
Pada titik ini, Anda akan melihat file output $HOME/results/blastp.out . Dengan kueri Anda, Blast mengidentifikasi urutan protein P80049.1 sebagai kecocokan dengan skor 14,2 dan nilai-E 0,96. Untuk melihat konten file output ini, gunakan perintah more $HOME/results/blastp.out .
Ingatlah untuk menghentikan atau menghapus VM untuk mencegah biaya tambahan. Anda dapat melakukan ini di konsol GCP seperti yang ditunjukkan di bawah ini. 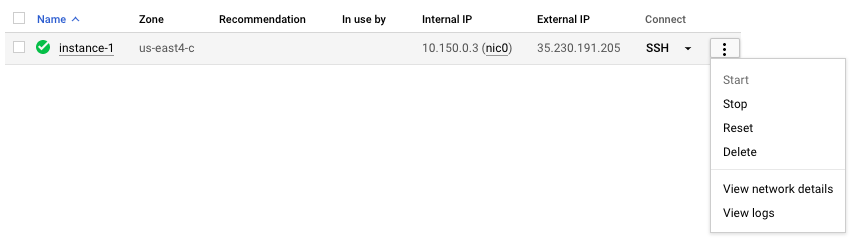
Salah satu janji komputasi awan adalah skalabilitas. Di bagian ini, kami akan menunjukkan cara menggunakan gambar Blast+ Docker pada skala produksi di Google Cloud Platform. Kami akan melakukan analisis BLAST yang mirip dengan pendekatan yang dijelaskan dalam publikasi ini untuk membandingkan de novo yang disejajarkan dari bakteri 16S-23S yang diurutkan terhadap database pengumpulan nukleotida (NT).
Untuk menguji skalabilitas, kami akan menggunakan input dari berbagai ukuran untuk memperkirakan jumlah waktu untuk mengunduh database koleksi nukleotida dan menjalankan pencarian BLAST menggunakan versi terbaru dari gambar Blast+ Docker. Hasil yang diharapkan dirangkum dalam tabel berikut.
File input: 28 sampel (file multi-fasta) yang berisi de novo disejajarkan dari publikasi.
(Instruksi untuk mengunduh dan membuat file input dijelaskan di blok kode di bawah ini.)
Database: Database Koleksi Nukleotida BLAST yang telah diformat sebelumnya, Versi 5 (NT): 68.7217 GB (dari Mei 2019)
| Nama file input | Konten file | Ukuran file | Jumlah urutan | Jumlah nukleotida | Ukuran output yang diharapkan | |
|---|---|---|---|---|---|---|
| Analisis 1 | query1.fa | hanya sampel 1 | 59 kb | 121 | 51.119 | 3.1 GB |
| Analisis 2 | kueri5.fa | Hanya sampel 1-5 | 422 kb | 717 | 375.154 | 10.4 GB |
| Analisis 3 | query.fa | Semua 28 sampel | 2.322 MB | 3798 | 2.069.892 | 47,8 GB |
| Tipe/Zona VM | CPU | Memori (GB) | Biaya per jam* | Unduh NT (min) | Analisis 1 (min) | Analisis 2 (min) | Analisis 3 (min) | Total biaya ** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-East4C | 8 | 30 | $ 0,312 | 9 | 22 | - | - | - |
| N1-Standard-16 US-EAST4C | 16 | 60 | $ 0,611 | 9 | 14 | 53 | 205 | $ 2,86 |
| N1-HighMem-16 US-EAST4C | 16 | 104 | $ 0,767 | 9 | 9 | 30 | 143 | $ 2,44 |
| N1-HighMem-16 US-WEST2A | 16 | 104 | $ 0,809 | 11 | 9 | 30 | 147 | $ 2,60 |
| N1-HighMem-16 US-West1b | 16 | 104 | $ 0,674 | 11 | 9 | 30 | 147 | $ 2,17 |
| Situs web Blast (Blastn) | - | - | - | - | Pencarian melebihi pembatasan saat ini pada penggunaan | Pencarian melebihi pembatasan saat ini pada penggunaan | Pencarian melebihi pembatasan saat ini pada penggunaan | - |
Semua instance GCP dikonfigurasi dengan 200 GB disk standar persisten.
*Biaya per jam disediakan oleh Google Cloud Platform (Mei 2019) ketika VM dibuat dan dapat berubah.
** Total biaya diperkirakan menggunakan biaya per jam dan total waktu untuk mengunduh NT dan menjalankan analisis 1, analisis 2, dan analisis 3. Perkiraan hanya digunakan untuk perbandingan; Biaya Anda dapat bervariasi dan merupakan tanggung jawab Anda untuk memantau dan mengelola.
Silakan merujuk ke GCP untuk informasi lebih lanjut tentang jenis mesin, wilayah dan zona, dan menghitung biaya.
Harap dicatat bahwa menjalankan biner blastn tanpa menentukan parameter -task memohon algoritma megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
Anda telah menyelesaikan seluruh tutorial. Pada titik ini, jika Anda tidak memerlukan data yang diunduh untuk analisis lebih lanjut, silakan hapus VM untuk mencegah biaya tambahan.
Untuk menghapus instance, ikuti instruksi di bagian tersebut hentikan instance GCP.
Untuk informasi tambahan, silakan merujuk ke dokumentasi Google Cloud Platform pada siklus hidup instan.
Untuk menjalankan contoh -contoh ini, Anda akan memerlukan akun Amazon Web Services (AWS). Jika Anda belum memilikinya, Anda dapat membuat akun yang menyediakan kemampuan untuk menjelajahi dan mencoba layanan AWS secara gratis hingga batas yang ditentukan untuk setiap layanan. Untuk memulai, kunjungi situs Tier GRATIS, ini akan membutuhkan kartu kredit yang valid namun tidak akan dikenakan biaya jika Anda menghitung di dalam tingkat gratis. Saat memilih produk tingkat gratis, pastikan itu ada di komputasi kategori produk.
Instruksi ini membuat EC2 VM berdasarkan gambar mesin Amazon (AMI) yang mencakup Docker dan ketergantungannya.
Dengan VM yang dibuat, Anda mengaksesnya dari komputer lokal Anda menggunakan SSH. File Kunci Pasangan / .pem Anda berfungsi sebagai kredensial Anda.
Ada beberapa cara untuk membuat koneksi SSH. Dari daftar instance EC2 di konsol AWS, pilih Connect , lalu ikuti instruksi untuk metode koneksi klien SSH mandiri .
Instruksi terperinci untuk menghubungkan ke Linux VM dapat ditemukan di sini.
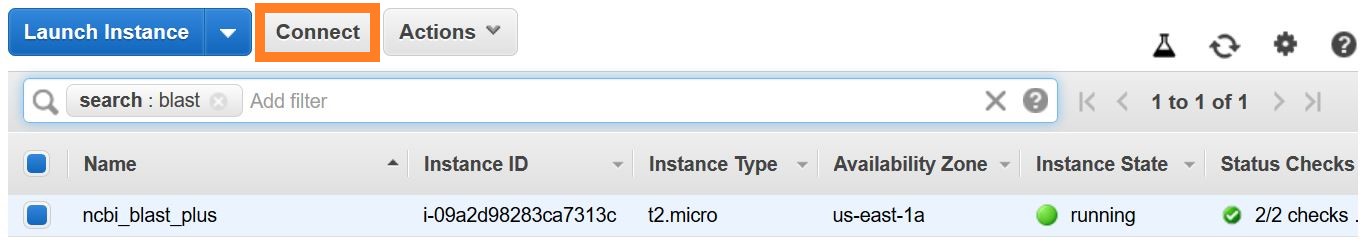
Tentukan EC2-User sebagai nama pengguna, alih-alih root di baris perintah SSH Anda atau ketika diminta untuk login, tentukan EC2-User sebagai nama pengguna.
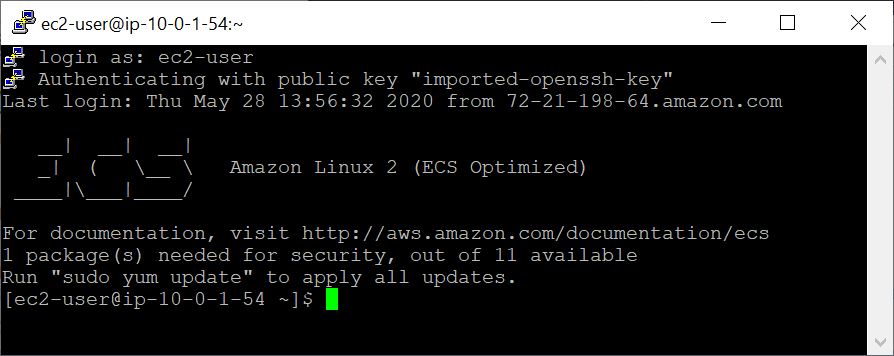
Dalam contoh ini, kita akan mulai dengan mengambil urutan kueri dan basis data dan kemudian membuat database BLAST khusus.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Untuk memverifikasi basis data BLAST yang baru dibuat di atas, Anda dapat menjalankan perintah berikut untuk menampilkan aksesi, panjang urutan, dan nama umum dari urutan dalam database.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Saat menjalankan BLAST dalam wadah Docker, catat mounts ( -v opsi) yang ditentukan untuk perintah docker run untuk membuat input dan output dapat diakses. Dalam contoh di bawah ini, dua dudukan pertama menyediakan akses ke basis data BLAST, dudukan ketiga menyediakan akses ke urutan kueri, dan dudukan keempat menyediakan direktori untuk menyimpan hasilnya. (Perhatikan :ro dan :rw Opsi, yang masing-masing memasang direktori sebagai baca dan baca-tulis.)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
Pada titik ini, Anda akan melihat file output $HOME/results/blastp.out . Dengan kueri Anda, Blast mengidentifikasi urutan protein P80049.1 sebagai kecocokan dengan skor 14,2 dan nilai-E 0,96. Untuk melihat konten file output ini, gunakan perintah more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
Output yang diharapkan adalah daftar DB BLAST, termasuk nama, deskripsi, ukuran, dan tanggal terakhir yang diperbarui.
Untuk deskripsi terperinci tentang update_blastdb.pl , silakan merujuk ke dokumentasi. Secara default update_blastdb.pl akan mengunduh dari cloud asalkan Anda terhubung, atau dari NCBI jika Anda tidak menggunakan penyedia cloud yang didukung.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Output yang diharapkan adalah daftar nama Blast DBS.
Ingatlah untuk menghentikan atau menghentikan VM untuk mencegah biaya tambahan. Anda dapat melakukan ini dari daftar instance EC2 di konsol AWS seperti yang ditunjukkan di bawah ini.
Contoh ini membutuhkan host multi-core. Dengan demikian, biaya komputasi EC2 akan direalisasikan dengan melaksanakan contoh ini. Tingkat saat ini untuk jenis instance yang digunakan - t2.large - adalah $ 0,093/jam.
Instruksi ini membuat EC2 VM berdasarkan gambar mesin Amazon (AMI) yang mencakup Docker dan ketergantungannya.
Dengan VM yang dibuat, Anda mengaksesnya dari komputer lokal Anda menggunakan SSH. File Kunci Pasangan / .pem Anda berfungsi sebagai kredensial Anda.
Ada beberapa cara untuk membuat koneksi SSH. Dari daftar instance EC2 di konsol AWS, pilih Connect , lalu ikuti instruksi untuk metode koneksi klien SSH mandiri .
Instruksi terperinci untuk menghubungkan ke Linux VM dapat ditemukan di sini.
Tentukan EC2-User sebagai nama pengguna, alih-alih root di baris perintah SSH Anda atau ketika diminta untuk login, tentukan EC2-User sebagai nama pengguna.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
Perintah di bawah memasang (menggunakan opsi -v ) jalur $HOME/blastdb pada mesin lokal sebagai /blast/blastdb pada wadah, dan blastdbcmd menunjukkan basis data BLAST yang tersedia di lokasi ini.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Anda akan melihat /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
Pada titik ini, Anda akan melihat file output $HOME/results/blastp_pdbaa.out . Untuk melihat konten file output ini, gunakan perintah more $HOME/results/blastp_pdbaa.out .
Salah satu cara untuk mentransfer file antara komputer lokal Anda dan instance Linux adalah dengan menggunakan Secure Copy Protocol (SCP).
Bagian yang mentransfer file ke instance Linux dari Linux menggunakan SCP dari panduan pengguna Amazon EC2 untuk instance Linux memberikan instruksi terperinci untuk proses ini.
NCBI meng -host database yang sama di AWS, GCP, dan situs NCBI FTP. Tabel di bawah ini memiliki daftar basis data saat ini pada November, 2022.
Dimungkinkan juga untuk mendapatkan daftar saat ini dengan perintah:
Docker Run --RM NCBI/Blast Update_BlastDB.pl --showall Pretty
atau
update_blastdb.pl --showall cantik # Setelah mengunduh paket blast+.
Seperti yang ditunjukkan di atas, update_blastdb.pl juga dapat digunakan untuk mengunduh database ini. Ini akan secara otomatis memilih sumber daya yang sesuai (misalnya, GCP jika Anda berada di dalam penyedia itu).
Basis data ini juga dapat dicari dengan ElasticBlast di GCP dan AWS.
Mengakses database pada AWS atau GCP di luar penyedia cloud kemungkinan akan menghasilkan biaya jalan keluar ke akun Anda. Jika Anda tidak berada di penyedia cloud, Anda harus menggunakan database di situs NCBI FTP.
| Nama | Jenis | Judul |
|---|---|---|
| 16S_RIBOSOMAL_RNA | DNA | 16S RNA ribosom (bakteri dan strain tipe archaea) |
| 18S_Fungal_SePreences | DNA | Urutan RNA ribosom 18S (SSU) dari jenis jamur dan bahan referensi |
| 28S_Fungal_Sequences | DNA | 28S Ribosomal RNA Sequences (LSU) dari Jenis Jamur dan Bahan Referensi |
| Betacoronavirus | DNA | Betacoronavirus |
| GCF_000001405.38_TOP_LEVEL | DNA | Homo Sapiens GRCH38.P12 [GCF_000001405.38] Kromosom ditambah perancah tanpa tempat dan tidak terlokalisasi |
| GCF_000001635.26_TOP_LEVEL | DNA | Mus musculus GRCM38.P6 [GCF_000001635.26] Kromosom ditambah perancah tanpa tempat dan tidak terlokalisasi |
| Its_refseq_fungi | DNA | Wilayah spacer transkrip internal (ITS) dari jenis jamur dan bahan referensi |
| ITS_EUKARYOTE_SEPERENCES | DNA | Ledakan eukariota |
| LSU_EUKARYOTE_RRNA | DNA | Asam nukleat ribosom subunit besar untuk eukariota |
| LSU_PROKARYOTE_RRNA | DNA | Asam nukleat ribosom subunit besar untuk prokariota |
| SSU_EUKARYOTE_RRNA | DNA | Asam nukleat ribosom subunit kecil untuk eukariota |
| Env_nt | DNA | Sampel lingkungan |
| nt | DNA | Pengumpulan Nukleotida (NT) |
| Patnt | DNA | Urutan nukleotida yang berasal dari divisi paten GenBank |
| pdbnt | DNA | Database nukleotida PDB |
| ref_euk_rep_genomes | DNA | Database genom representatif eukariotik refseq |
| ref_prok_rep_genomes | DNA | Refseq Prokaryote Representative Genom (berisi Majelis Refseq) |
| ref_viroids_rep_genomes | DNA | Refseq Viroids Genom Representatif |
| ref_viruses_rep_genomes | DNA | Virus Refseq Genom Perwakilan |
| refseq_rna | DNA | Urutan referensi transkrip NCBI |
| refseq_select_rna | DNA | Refseq Pilih Urutan RNA |
| tsa_nt | DNA | Urutan Transkriptome Shotgun Assembly (TSA) |
| Env_nr | Protein | Protein dari proyek metagenomik WGS |
| tengara | Protein | Database tengara untuk SmartBlast |
| nr | Protein | Semua terjemahan CDS GenBank Non-Redundan+PDB+Swissprot+PIR+PRF tidak termasuk sampel lingkungan dari proyek WGS |
| pdbaa | Protein | Database Protein PDB |
| Pataa | Protein | Urutan protein yang berasal dari divisi paten GenBank |
| refseq_protein | Protein | Urutan referensi protein NCBI |
| refseq_select_prot | Protein | Refseq SELECT protein |
| Swissprot | Protein | Urutan UnipRotKB/Swissprot yang tidak berlebihan |
| tsa_nr | Protein | Urutan Transkriptome Shotgun Assembly (TSA) |
| CDD | Protein | Database Domain Conserved (CDD) adalah kumpulan model penyelarasan beberapa urutan yang dianotasi dengan baik sebagai matriks skor spesifik posisi |
NCBI menyediakan metadata untuk database BLAST yang tersedia di AWS, GCP dan situs NCBI FTP.
Mengakses database pada AWS atau GCP di luar penyedia cloud kemungkinan akan menghasilkan biaya jalan keluar ke akun Anda. Jika Anda tidak berada di penyedia cloud, Anda harus menggunakan database di situs NCBI FTP.
Pada AWS dan GCP, file tersebut berada dalam subdirektori yang bergantung pada tanggal dengan database. Untuk menemukan subdirektori terbaru yang valid, pertama baca s3://ncbi-blast-databases/latest-dir (di AWS) atau gs://blast-db/latest-dir (di GCP). latest-dir adalah file teks dengan cap tanggal (mis., 2020-09-29-01-05-01) yang menentukan direktori terbaru. Direktori yang tepat adalah AWS atau GCP Base URI untuk database BLAST (misalnya, s3://ncbi-blast-databases/ untuk AWS) ditambah teks dalam file latest-dir . Contoh URI, dalam AWS, adalah s3://ncbi-blast-databases/2020-09-29-01-05-01 . GCP URI akan serupa.
Kutipan dari file metadata ditunjukkan di bawah ini. Sebagian besar bidang memiliki makna yang jelas. File tersebut terdiri dari database BLAST. Bidang bytes-total mewakili ukuran total basis data BLAST dalam byte dan dimaksudkan untuk menentukan berapa banyak ruang disk yang diperlukan.
Contoh di bawah ini adalah dari AWS, tetapi file metadata pada GCP memiliki format yang sama. Databases on the FTP site are in gzipped tarfiles, one per volume of the BLAST database, so those are listed rather than the individual files.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
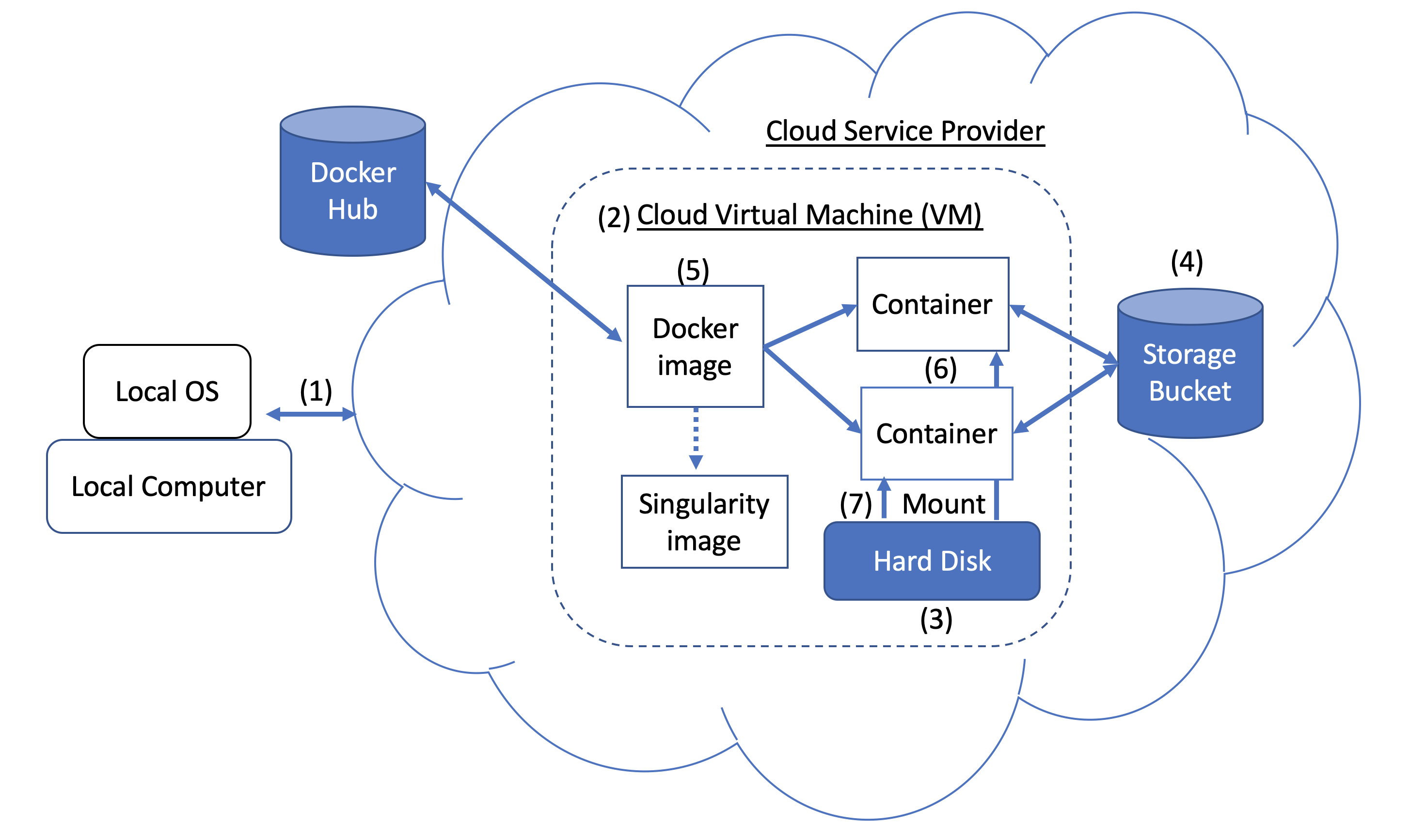 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.