يحتوي هذا المستودع على وثائق لتطبيقات سطر أوامر NCBI Blast+ في صورة Docker. سنوضح كيفية استخدام صورة Docker لتشغيل تحليل الانفجار على منصة Google Cloud (GCP) و Amazon Web Services (AWS) باستخدام مثال أساسي صغير ومثال أكثر تقدماً على مستوى الإنتاج. بعض المعرفة الأساسية لأوامر UNIX/Linux و BLAST+ مفيدة في إكمال هذا البرنامج التعليمي.
runrun
يجد المركز الوطني لمعلومات التكنولوجيا الحيوية (NCBI) أداة البحث المحلية الأساسية (Blast) مناطق من التشابه المحلي بين التسلسلات. يقارن البرنامج تسلسل النيوكليوتيدات أو البروتين بقواعد بيانات التسلسل ويحسب الأهمية الإحصائية للمطابقات. يمكن استخدام الانفجار لاستنتاج العلاقات الوظيفية والتطورية بين التسلسلات وكذلك المساعدة في تحديد أفراد عائلات الجينات.
تم تقديمه في عام 2009 ، Blast+ هو نسخة محسنة من تطبيقات سطر أوامر Blast. للحصول على وصف كامل لميزات وقدرات Blast+، يرجى الرجوع إلى دليل مستخدم تطبيقات سطر أوامر Blast.
توفر الحوسبة السحابية وفورات محتملة للتكاليف باستخدام الموارد الحسابية عند الطلب والقابلة للتطوير والمرنة. في حين أن الوصف التفصيلي للتقنيات والفوائد السحابية المختلفة خارج نطاق هذا المستودع ، فإن الأقسام التالية تحتوي على معلومات مطلوبة لبدء تشغيل صورة Blast+ Docker على منصة Google Cloud (GCP).
Docker هي أداة لتنفيذ المحاكاة الافتراضية مستوى نظام التشغيل باستخدام حاويات البرامج. في تكنولوجيا الحاويات * ، الصورة هي لقطة لبيئة تحليلية تغلف التطبيقات (التطبيقات) والتبعيات. يمكن حفظ صورة ، وهي ملف تم تصميمه من قائمة الإرشادات بشكل أساسي ومشاركته بسهولة للآخرين لإعادة إنشاء البيئة التحليلية الدقيقة عبر المنصات وأنظمة التشغيل. الحاوية هي مثيل وقت تشغيل الصورة. باستخدام الحاوية ، يمكن للمستخدمين تجاوز الخطوات المعقدة في كثير من الأحيان في تجميع وتكوين وتثبيت أداة تعتمد على UNIX مثل Blast+. بالإضافة إلى قابلية الحمل ، يعد تحفيز الحاوية نهجًا خفيف الوزن لجعل التحليل أكثر قابلية للاكتشاف ، ويمكن الوصول إليه ، قابل للتشغيل ، قابل لإعادة الاستخدام (عادلة) ، وفي نهاية المطاف ، قابل للتكرار.
*هناك العديد من أدوات ومعايير الحاويات ، مثل Docker و Singularity. سوف نركز فقط على Docker ، والذي يعتبر المعيار الفعلي من قبل الكثيرين في هذا المجال.
تتضمن الأقسام التالية إرشادات لإنشاء جهاز Google Virtual ، وتثبيت Docker ، وقم بتشغيل أوامر Blast+ باستخدام صورة Docker.
يوفر هذا القسم مجموعة سريعة من تحليل الانفجار في بيئة Docker على مثيل Google. هذا المقصود بمثابة نظرة عامة على أولئك الذين يريدون فقط فهم مبادئ الحل. إذا كنت تعمل مع مثيلات Amazon ، فيرجى الانتقال إلى قسم إعداد خدمات الويب Amazon في هذه الوثائق. سيتم استخدام Google Cloud Shell ، وهي بيئة SHELL التفاعلية ، في هذا المثال ، مما يجعل من الممكن تشغيل المثال الصغير التالي دون الحاجة إلى إجراء إعداد إضافي ، مثل إنشاء حساب فواتير أو مثيل حساب. تتم تغطية أوصاف أكثر تفصيلاً لخطوات التحليل والأوامر البديلة والموضوعات المتقدمة في الأقسام اللاحقة من هذه الوثائق.
المتطلبات: حساب Google
تدفق المهمة: 
بيانات الإدخال:
أولاً ، في نافذة أو علامة تبويب منفصلة ، قم بتسجيل الدخول على https://console.cloud.google.com/
انقر فوق زر تنشيط Cloud Shell في الركن الأيمن العلوي من وحدة التحكم منصة Google Cloud. 
سترى الآن نافذة جلسة السحابة الخاصة بك: 
والخطوة التالية هي نسخ أوامر الأوامر أدناه في جلسة السحابة الخاصة بك.
يرجى ملاحظة: في github يمكنك استخدام الماوس الخاص بك لنسخ ؛ ومع ذلك ، في قذيفة الأوامر يجب عليك استخدام لوحة المفاتيح الخاصة بك. في Windows أو Unix/Linux ، استخدم Control+C للنسخ والتحكم Control+V للصق. على MacOS ، استخدم Command+C لنسخ و Command+V للصق.
للتمرير في قذيفة السحابة ، قم بتمكين شريط التمرير في Terminal settings باستخدام أيقونة وجع. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
في هذه المرحلة ، يجب أن ترى الإخراج على الشاشة. مع استعلامك ، حدد Blast تسلسل البروتين P80049.1 كطابق مع درجة 14.2 وقيمة إلكترونية من 0.96.
للتحليل الأكبر ، يوصى باستخدام علامة -out لحفظ الإخراج في ملف. على سبيل المثال ، قم بإلحاق -out /blast/results/blastp.out إلى الأمر الأخير في الخطوة 3 أعلاه وعرض محتوى ملف الإخراج هذا باستخدام more $HOME/results/blastp.out .
يمكنك أيضًا الاستعلام عن P01349.FSA مقابل PDB كما هو موضح في كتلة الكود التالية.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
لقد أكملت الآن مهمة بسيطة وشاهدت كيف يعمل Blast+ مع Docker. للتعرف على Docker و Blast+ على مقياس الإنتاج ، يرجى المتابعة إلى القسم التالي.
في القسم 2-دليل خطوة بخطوة باستخدام صورة BLAST+ Docker ، سنستخدم نفس المثال الصغير من القسم السابق ونناقش الأساليب البديلة ، وأوامر Docker و Blast+ الإضافية ، وخيارات وأمر Docker. في القسم 3 ، سوف نوضح كيفية تشغيل صورة Blast+ Docker على مقياس الإنتاج.
أولاً ، تحتاج إلى إعداد جهاز افتراضي لـ Google Cloud Platform (GCP) للتحليل.
تقدم GCP حاليًا رصيدًا بقيمة 300 دولار ، والذي ينتهي صلاحيته بعد 12 شهرًا من التنشيط ، لتحفيز مستخدمي السحابة الجدد. ستظهر لك الخطوات التالية كيفية تنشيط هذا الائتمان. سيُطلب منك الحصول على معلومات الفواتير ، لكن GCP لن يقوم بشحن تلقائي لك بمجرد انتهاء المحاكمة ؛ يجب أن تختار الترقية يدويًا إلى حساب مدفوع.
بعد تسجيل الدخول ، انقر فوق تنشيط لتفعيل الائتمان 300 دولار. 
أدخل بلدك ، على سبيل المثال ، الولايات المتحدة ، وتحديد المربع الذي يشير إلى أنك قد قرأت وقبول شروط الخدمة.
ضمن "نوع الحساب" ، حدد "فرد". (قد يتم تحديد هذا مسبقًا في حساب Google الخاص بك)
أدخل اسمك وعنوانك.
تحت "كيف تدفع" ، حدد "المدفوعات التلقائية". (قد يتم تحديد هذا مسبقًا في حساب Google الخاص بك) يشير هذا إلى أنك ستدفع تكاليف بعد استخدام الخدمة ، إما عندما تكون قد وصلت إلى عتبة الفواتير أو كل 30 يومًا ، أيهما يأتي أولاً.
ضمن "طريقة الدفع" ، حدد "إضافة بطاقة الائتمان أو الخصم" وأدخل معلومات بطاقة الائتمان الخاصة بك. لن يتم شحنك تلقائيًا بمجرد انتهاء التجربة. يجب أن تختار الترقية إلى حساب مدفوع قبل أن يتم فرض طريقة الدفع الخاصة بك.
انقر فوق "ابدأ التجريبي المجاني" لإنهاء التسجيل. عند اكتمال هذه العملية ، يجب أن ترى شاشة ترحيب GCP.
في هذه المرحلة ، يجب أن ترى تقدير التكلفة لهذه الحالة على الجانب الأيمن من نافذتك. 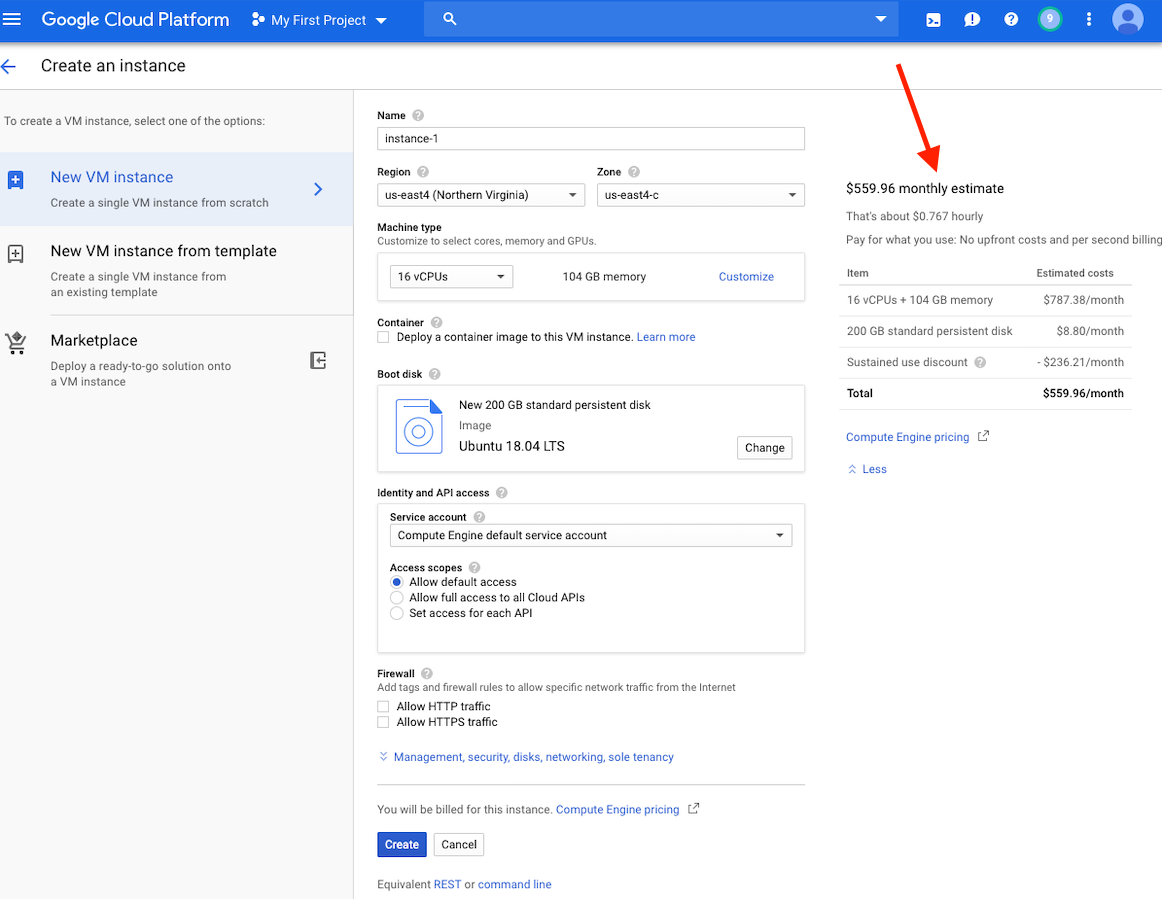
يرجى ملاحظة: إنشاء VM في نفس المنطقة مثل التخزين يمكن أن يوفر أداء أفضل. نوصي بإنشاء VM في منطقة الولايات المتحدة East4. إذا كان لديك وظيفة ستستغرق عدة ساعات ، ولكن أقل من 24 ساعة ، فيمكنك الاستفادة من VMs الاستباقية.
يمكن العثور على إرشادات مفصلة لإنشاء حساب GCP وإطلاق VM هنا.
بمجرد إنشاء VM الخاص بك ، يجب عليك الوصول إليه من جهاز الكمبيوتر المحلي. هناك العديد من الطرق للوصول إلى VM ، اعتمادًا على الطرق التي ترغب في استخدامها. على GCP ، الطريقة الأكثر وضوحًا هي SSH من المتصفح.
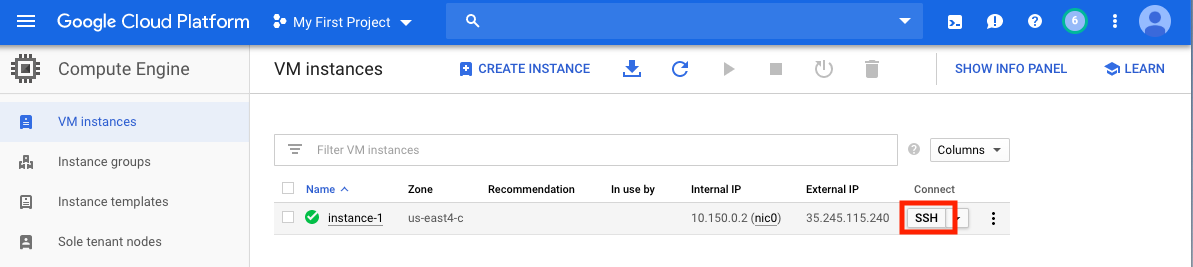
لديك الآن قذيفة القيادة تعمل وأنت مستعد للمضي قدما.
تذكر أن تتوقف أو حذف VM لمنع تكبد تكلفة إضافية.
في هذا القسم ، سنقوم بتغطية تثبيت Docker ، ونناقش العديد من خيارات docker run ، وندرس بنية أمر Docker. سوف نستخدم نفس المثال الصغير من القسم 1 ونستكشف الأساليب البديلة في تشغيل صورة BLAST+ Docker. ومع ذلك ، فإننا نستخدم مثيل VM حقيقي ، والذي يوفر أداءً ووظائف أكبر من Google Cloud Shell.
بيانات الإدخال
في نظام الإنتاج ، يجب تثبيت Docker كتطبيق.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
لتأكيد التثبيت الصحيح لـ Docker ، قم بتشغيل Command docker run hello-world . إذا تم تثبيتها بشكل صحيح ، فيجب أن ترى "Hello from Docker! ..." (https://docs.docker.com/samples/library/hello-world/)
هذا القسم اختياري.
فيما يلي قائمة بخيارات سطر أوامر docker run المستخدمة في هذا البرنامج التعليمي.
| الاسم ، قصير اليد (إذا كان ذلك متاحًا) | وصف |
|---|---|
--rm | قم بإزالة الحاوية تلقائيًا عند خروجها |
--volume ، -v | ربط وحدة التخزين |
--workdir ، -w | دليل العمل داخل الحاوية |
هذا القسم اختياري.
بالنسبة لهذا البرنامج التعليمي ، سيكون من المفيد فهم بنية أمر Docker. يتكون الأمر التالي من ثلاثة أجزاء.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
الجزء الأول من docker run --rm ncbi/blast هو تعليمات لتشغيل صورة Docker ncbi/blast وإزالة الحاوية عند اكتمال التشغيل.
يجعل الجزء الثاني من الأمر بيانات تسلسل الاستعلام متاحة في الحاوية. يستخدم Docker Bind Mounts -v لتركيب الدلائل المحلية إلى الدلائل داخل الحاوية وتوفير إذن الوصول RW (قراءة وكتابة) أو RO (اقرأ فقط). على سبيل المثال ، على افتراض تخزين تسلسلات الموضوع الخاصة بك في دليل $ home/fasta على المضيف المحلي ، يمكنك استخدام المعلمة التالية لجعل هذا الدليل متاحًا داخل الحاوية في/blast/fasta كدليل للقراءة فقط -v $HOME/fasta:/blast/fasta:ro . يضع علامة -w /blast/blastdb_custom دليل العمل داخل الحاوية.
الجزء الثالث من الأمر هو أمر Blast+. في هذه الحالة ، يتم تنفيذ MakeblastDB لإنشاء ملفات قاعدة بيانات الانفجار.
يمكنك بدء جلسة باش تفاعلية لهذه الصورة باستخدام docker run -it ncbi/blast /bin/bash . بالنسبة لصورة Blast+ Docker ، توجد التنفيذيون في المجلد /الانفجار /BIN و /ROOT /EDIRECT وإضافته إلى مسار $ المتغير.
للحصول على وثائق إضافية حول أمر docker run ، يرجى الرجوع إلى الوثائق.
هذا القسم اختياري.
| قيادة Docker | وصف |
|---|---|
docker ps -a | يعرض قائمة الحاويات |
docker rm $(docker ps -q -f status=exited) | يزيل جميع الحاويات المخرجات ، إذا كان لديك حاوية واحدة على الأقل |
docker rm <CONTAINER_ID> | يزيل حاوية |
docker images | يعرض قائمة بالصور |
docker rmi <REPOSITORY (IMAGE_NAME)> | يزيل صورة |
هذا القسم اختياري.
باستخدام صورة Docker هذه ، يمكنك تشغيل Blast+ في حاوية معزولة ، مما يسهل استنساخ نتائج الانفجار. بصفتك مستخدمًا لصورة Docker هذه ، من المتوقع أن توفر قواعد بيانات BLASL وتسلسل (تسلسل) الاستعلام لتشغيل الانفجار وكذلك موقع خارج الحاوية لحفظ النتائج. فيما يلي قائمة بالأدلة التي تستخدمها Blast+. سوف تنشئها في الخطوة 2.
| دليل | غاية | ملحوظات |
|---|---|---|
$HOME/blastdb | يخزن قواعد بيانات الانفجار المقدمة من NCBI | إذا تم تعيينه على مسار واحد مطلق ، يمكن استخدام متغير بيئة $BLASTDB بدلاً من ذلك (انظر تكوين الانفجار عبر متغيرات البيئة.) |
$HOME/queries | يخزن تسلسل الاستعلام المقدم من المستخدم (S) | |
$HOME/fasta | يخزن تسلسل fasta المقدم من المستخدم لإنشاء قاعدة بيانات (ق) الانفجار | |
$HOME/results | متاجر الانفجار نتائج | جبل مع أذونات rw |
$HOME/blastdb_custom | يخزن قواعد بيانات الانفجار المقدمة من المستخدم |
هذا القسم اختياري.
يعرض الأمر التالي أحدث إصدار للانفجار.
docker run --rm ncbi/blast blastn -version
تتيح لك إلحاق علامة باسم الصورة ( ncbi/blast ) استخدام إصدار مختلف من BLAST+ (انظر "العلامات المدعومة وملاحظات الإصدار المعنية" للإصدارات المدعومة).
توجد إصدارات مختلفة من Blast+ في صور Docker مختلفة. سيبدأ الأمر التالي تنزيل صورة BLAST+ الإصدار 2.9.0 Docker.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
على سبيل المثال ، لاستخدام صورة docker في الإصدار 2.9.0 بدلاً من أحدث إصدار ، استبدل الجزء الأول من الأمر
docker run --rm ncbi/blast مع docker run --rm ncbi/blast:2.9.0
هذا القسم اختياري.
في هذا المثال ، سنبدأ بجلب تسلسل الاستعلام وقواعد البيانات ثم إنشاء قاعدة بيانات Blast مخصصة.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
للتحقق من قاعدة بيانات BLAST التي تم إنشاؤها حديثًا أعلاه ، يمكنك تشغيل الأمر التالي لعرض الملحقات وطول التسلسل والاسم الشائع للتسلسل في قاعدة البيانات.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
كبديل ، يمكنك أيضًا تنزيل قواعد بيانات BLAST المسبقة من NCBI أو NCBI Google Storage Bucket.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
للحصول على وصف مفصل لـ update_blastdb.pl ، يرجى الرجوع إلى الوثائق. بشكل افتراضي ، سيتم تنزيل update_blastdb.pl من السحابة شريطة أن تكون متصلاً بـ NCBI ، أو من NCBI إذا كنت لا تستخدم مزودًا سحابيًا مدعومًا.
هذا القسم اختياري.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
هذا القسم اختياري.
يتصاعد الأمر أدناه لمسار $HOME/blastdb على الجهاز المحلي AS /blast/blastdb على الحاوية ، ويعرض blastdbcmd قواعد بيانات الانفجار المتاحة في هذا الموقع.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
يجب أن ترى /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
يجب أن ترى /blast/blastdb_custom/nurse-shark-proteins Protein .
عند تشغيل الانفجار في حاوية Docker ، لاحظ الحامل المحددة لأمر docker run لجعل الإدخال والمخرجات متاحة. في الأمثلة أدناه ، يوفر أول حواملان الوصول إلى قواعد بيانات الانفجار ، ويوفر الحامل الثالث إمكانية الوصول إلى تسلسل (تسلسل) الاستعلام ، ويوفر الجبل الرابع دليلًا لحفظ النتائج. (لاحظ الخيارات :ro و :rw ، والتي تُركض الدلائل على أنها قراءة فقط وقراءة على التوالي.)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
في هذه المرحلة ، يجب أن ترى ملف الإخراج $HOME/results/blastp.out . مع استعلامك ، حدد Blast تسلسل البروتين P80049.1 كطابق مع درجة 14.2 وقيمة إلكترونية من 0.96. لعرض محتوى ملف الإخراج هذا ، استخدم الأمر more $HOME/results/blastp.out .
تذكر أن تتوقف أو حذف VM لمنع تكبد تكلفة إضافية. يمكنك القيام بذلك في وحدة التحكم في GCP كما هو موضح أدناه. 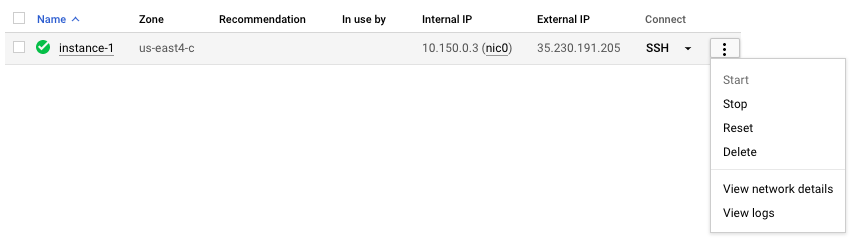
واحدة من وعود الحوسبة السحابية هي قابلية التوسع. في هذا القسم ، سوف نوضح كيفية استخدام صورة Blast+ Docker على مقياس الإنتاج على منصة Google Cloud. سنقوم بإجراء تحليل للانفجار على غرار النهج الموصوف في هذا المنشور لمقارنة de Novo المحاذاة contigs من تسلسل البكتيريا 16S-23S ضد قاعدة بيانات جمع النيوكليوتيدات (NT).
لاختبار قابلية التوسع ، سوف نستخدم مدخلات بأحجام مختلفة لتقدير مقدار الوقت لتنزيل قاعدة بيانات مجموعة Nucleotide وتشغيل البحث عن الانفجار باستخدام أحدث إصدار من صورة Blast+ Docker. يتم تلخيص النتائج المتوقعة في الجداول التالية.
ملفات الإدخال: 28 عينة (ملفات multi-fasta) التي تحتوي على contigs de novo محاذاة من المنشور.
(تعليمات لتنزيل وإنشاء ملفات الإدخال موصوفة في كتلة الكود أدناه.)
قاعدة البيانات: قاعدة بيانات مجموعة النوكليوتيدات المسبقة المسبقة ، الإصدار 5 (NT): 68.7217 غيغابايت (من مايو 2019)
| إدخال اسم الملف | محتوى الملف | حجم الملف | عدد التسلسلات | عدد النيوكليوتيدات | حجم الإخراج المتوقع | |
|---|---|---|---|---|---|---|
| تحليل 1 | Query1.fa | عينة فقط 1 | 59 كيلو بايت | 121 | 51،119 | 3.1 غيغابايت |
| تحليل 2 | Query5.fa | العينات فقط 1-5 | 422 كيلو بايت | 717 | 375154 | 10.4 غيغابايت |
| التحليل 3 | Query.FA | جميع العينات الـ 28 | 2.322 ميغابايت | 3798 | 2،069،892 | 47.8 جيجابايت |
| نوع VM/المنطقة | وحدة المعالجة المركزية | الذاكرة (GB) | تكلفة كل ساعة* | تنزيل NT (دقيقة) | التحليل 1 (دقيقة) | التحليل 2 (دقيقة) | التحليل 3 (دقيقة) | التكلفة الإجمالية ** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-East4c | 8 | 30 | 0.312 دولار | 9 | 22 | - | - | - |
| N1-Standard-16 US-East4c | 16 | 60 | 0.611 دولار | 9 | 14 | 53 | 205 | 2.86 دولار |
| N1-Highmem-16 US-East4c | 16 | 104 | 0.767 دولار | 9 | 9 | 30 | 143 | 2.44 دولار |
| N1-Highmem-16 US-West2a | 16 | 104 | 0.809 دولار | 11 | 9 | 30 | 147 | 2.60 دولار |
| N1-HIGHMEM-16 US-WEST1B | 16 | 104 | 0.674 دولار | 11 | 9 | 30 | 147 | 2.17 دولار |
| موقع Blast (Blastn) | - | - | - | - | يتجاوز عمليات البحث القيود الحالية على الاستخدام | يتجاوز عمليات البحث القيود الحالية على الاستخدام | يتجاوز عمليات البحث القيود الحالية على الاستخدام | - |
يتم تكوين جميع مثيلات GCP مع 200 غيغابايت من القرص القياسي المستمر.
*تم توفير التكاليف بالساعة بواسطة منصة Google Cloud (May 2019) عند إنشاء VMs وتخضع للتغيير.
** تم تقدير إجمالي التكاليف باستخدام التكلفة بالساعة وإجمالي الوقت لتنزيل NT و Run Rension 1 ، التحليل 2 ، والتحليل 3. يتم استخدام التقديرات للمقارنة فقط ؛ قد تختلف تكاليفك ومسؤوليتك في مراقبة وإدارة.
يرجى الرجوع إلى GCP لمزيد من المعلومات حول أنواع الآلات والمناطق والمناطق وحساب التكلفة.
يرجى ملاحظة أن تشغيل blastn BINARY دون تحديد معلمة -task الخاصة به يستدعي خوارزمية Megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
لقد أكملت البرنامج التعليمي بأكمله. في هذه المرحلة ، إذا لم تكن بحاجة إلى البيانات التي تم تنزيلها لمزيد من التحليل ، فيرجى حذف VM لمنع تكبد التكلفة الإضافية.
لحذف مثيل ، اتبع الإرشادات الواردة في القسم إيقاف مثيل GCP.
للحصول على معلومات إضافية ، يرجى الرجوع إلى وثائق Google Cloud Platform في دورة حياة المثيل.
لتشغيل هذه الأمثلة ، ستحتاج إلى حساب Amazon Web Services (AWS). إذا لم يكن لديك حساب بالفعل ، فيمكنك إنشاء حساب يوفر القدرة على استكشاف خدمات AWS وتجربة AWS مجانًا إلى حدود محددة لكل خدمة. للبدء في زيارة موقع الطبقة المجانية ، سيتطلب ذلك بطاقة ائتمان صالحة ولكن لن يتم فرض رسوم عليها إذا قمت بحساب داخل الطبقة المجانية. عند اختيار منتج الطبقة المجانية ، تأكد من أنه في حساب فئة المنتج.
تنشئ هذه التعليمات EC2 VM استنادًا إلى صورة آلة Amazon (AMI) التي تتضمن Docker وتبعياتها.
مع إنشاء VM ، يمكنك الوصول إليه من جهاز الكمبيوتر المحلي باستخدام SSH. يعمل ملف المفتاح / .PEM الخاص بك كبيان اعتمادك.
هناك عدة طرق لإنشاء اتصال SSH. من قائمة مثيل EC2 في وحدة التحكم AWS ، حدد Connect ، ثم اتبع الإرشادات لطريقة الاتصال عميل SSH مستقل .
يمكن العثور على الإرشادات التفصيلية للاتصال بـ Linux VM هنا.
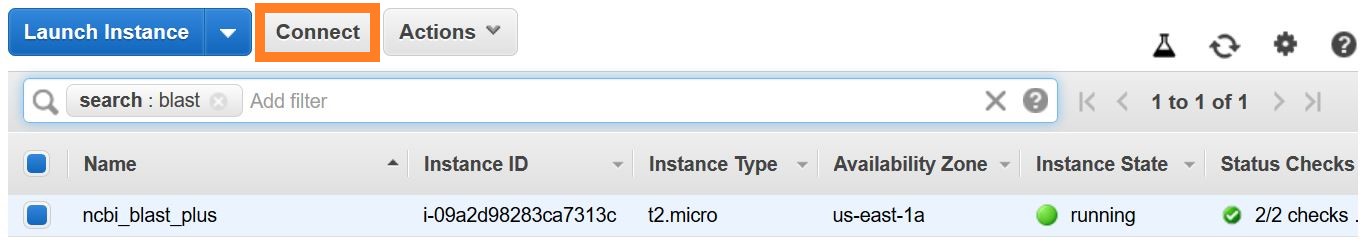
حدد EC2-User باعتباره اسم المستخدم ، بدلاً من الجذر في سطر أوامر SSH الخاص بك أو عند المطالبة بتسجيل الدخول ، حدد EC2-User كاسم مستخدم.
في هذا المثال ، سنبدأ بجلب تسلسل الاستعلام وقواعد البيانات ثم إنشاء قاعدة بيانات Blast مخصصة.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
للتحقق من قاعدة بيانات BLAST التي تم إنشاؤها حديثًا أعلاه ، يمكنك تشغيل الأمر التالي لعرض الملحقات وطول التسلسل والاسم الشائع للتسلسل في قاعدة البيانات.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
عند تشغيل الانفجار في حاوية Docker ، لاحظ أن Mounts (خيار -v ) محدد لأمر docker run لجعل الإدخال والمخرجات متاحة. في الأمثلة أدناه ، يوفر أول حواملان الوصول إلى قواعد بيانات الانفجار ، ويوفر الحامل الثالث إمكانية الوصول إلى تسلسل (تسلسل) الاستعلام ، ويوفر الجبل الرابع دليلًا لحفظ النتائج. (لاحظ الخيارات :ro و :rw ، والتي تُركض الدلائل على أنها قراءة فقط وقراءة على التوالي.)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
في هذه المرحلة ، يجب أن ترى ملف الإخراج $HOME/results/blastp.out . مع استعلامك ، حدد Blast تسلسل البروتين P80049.1 كطابق مع درجة 14.2 وقيمة إلكترونية من 0.96. لعرض محتوى ملف الإخراج هذا ، استخدم الأمر more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
الإخراج المتوقع هو قائمة من DBS Blast ، بما في ذلك اسمها ، وصفها ، الحجم ، وتاريخ آخر تحديث.
للحصول على وصف مفصل لـ update_blastdb.pl ، يرجى الرجوع إلى الوثائق. بشكل افتراضي ، سيتم تنزيل update_blastdb.pl من السحابة شريطة أن تكون متصلاً بـ NCBI ، أو من NCBI إذا كنت لا تستخدم مزودًا سحابيًا مدعومًا.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
الإخراج المتوقع هو قائمة بأسماء DBS Blast.
تذكر أن تتوقف أو إنهاء VM لمنع تكبد تكلفة إضافية. يمكنك القيام بذلك من قائمة مثيل EC2 في وحدة التحكم AWS كما هو موضح أدناه.
هذا المثال يتطلب مضيف متعدد النواة. على هذا النحو ، سيتم تحقيق رسوم حساب EC2 من خلال تنفيذ هذا المثال. المعدل الحالي لنوع المثيل المستخدم - t2.large - هو 0.093 دولار/ساعة.
تنشئ هذه التعليمات EC2 VM استنادًا إلى صورة آلة Amazon (AMI) التي تتضمن Docker وتبعياتها.
مع إنشاء VM ، يمكنك الوصول إليه من جهاز الكمبيوتر المحلي باستخدام SSH. يعمل ملف المفتاح / .PEM الخاص بك كبيان اعتمادك.
هناك عدة طرق لإنشاء اتصال SSH. من قائمة مثيل EC2 في وحدة التحكم AWS ، حدد Connect ، ثم اتبع الإرشادات لطريقة الاتصال عميل SSH مستقل .
يمكن العثور على الإرشادات التفصيلية للاتصال بـ Linux VM هنا.
حدد EC2-User باعتباره اسم المستخدم ، بدلاً من الجذر في سطر أوامر SSH الخاص بك أو عند المطالبة بتسجيل الدخول ، حدد EC2-User كاسم مستخدم.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
يتصاعد الأمر أدناه (باستخدام خيار -v ) مسار $HOME/blastdb على الجهاز المحلي AS /blast/blastdb على الحاوية ، ويظهر blastdbcmd قواعد بيانات الانفجار المتاحة في هذا الموقع.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
يجب أن ترى /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
في هذه المرحلة ، يجب أن ترى ملف الإخراج $HOME/results/blastp_pdbaa.out . لعرض محتوى ملف الإخراج هذا ، استخدم الأمر more $HOME/results/blastp_pdbaa.out .
تتمثل إحدى طرق نقل الملفات بين جهاز الكمبيوتر المحلي ومثيل Linux في استخدام بروتوكول النسخ الآمن (SCP).
يوفر القسم ينقل الملفات إلى مثيلات Linux من Linux باستخدام SCP من دليل مستخدم Amazon EC2 لحالات Linux تعليمات مفصلة لهذه العملية.
يستضيف NCBI نفس قواعد البيانات على AWS و GCP وموقع NCBI FTP. يحتوي الجدول أدناه على قائمة قواعد البيانات الحالية اعتبارًا من نوفمبر 2022.
من الممكن أيضًا الحصول على القائمة الحالية مع الأمر:
Docker Run -RM NCBI/Blast Update_blastdb.pl -Showall Pretty
أو
update_blastdb.pl -showall جميلة # بعد تنزيل حزمة Blast+.
كما هو موضح أعلاه ، يمكن أيضًا استخدام update_blastdb.pl لتنزيل قواعد البيانات هذه. سيتم تحديد المورد المناسب تلقائيًا (على سبيل المثال ، GCP إذا كنت داخل هذا المزود).
يمكن أيضًا البحث في قواعد البيانات هذه باستخدام مرونة على GCP و AWS.
من المحتمل أن يؤدي الوصول إلى قواعد البيانات على AWS أو GCP خارج مزود السحابة إلى رسوم الخروج إلى حسابك. إذا لم تكن على مزود السحابة ، فيجب عليك استخدام قواعد البيانات في موقع NCBI FTP.
| اسم | يكتب | عنوان |
|---|---|---|
| 16S_RIBOSOMAL_RNA | الحمض النووي | 16S RIBOSOMAL RNA (سلالات البكتيريا وسلالات نوع Archaea) |
| 18S_Fungal_Sequences | الحمض النووي | تسلسل RNA الريبوسوم 18S (SSU) من نوع الفطريات والمواد المرجعية |
| 28S_Fungal_Sequences | الحمض النووي | 28S RIBOSOMAL RNA Sequences (LSU) من نوع الفطريات والمواد المرجعية |
| فيروس بيتاكورون | الحمض النووي | فيروس بيتاكورون |
| GCF_000001405.38_TOP_LEVEL | الحمض النووي | homo sapiens grch38.p12 [gcf_000001405.38] الكروموسومات بالإضافة |
| GCF_000001635.26_TOP_LEVEL | الحمض النووي | Mus Musculus GRCM38.P6 [GCF_000001635.26] الكروموسومات بالإضافة |
| its_refseq_fungi | الحمض النووي | منطقة الفاصل المكتوبة الداخلية (ITS) من نوع الفطريات والمواد المرجعية |
| its_eukaryote_sequences | الحمض النووي | انفجار حقيقيات النواة |
| LSU_EUKARYOTE_RRNA | الحمض النووي | الحمض النووي الريبوسوم الفرعي الكبير لحقيقي حقيقيات |
| LSU_PROKARYOTE_RRNA | الحمض النووي | الحمض النووي الريبوسوم الفرعي الكبير للذاتية للذاتية |
| ssu_eukaryote_rrna | الحمض النووي | وحمض نيو نوكليكي فرعي صغير لحقيقي حقيقيات |
| env_nt | الحمض النووي | العينات البيئية |
| nt | الحمض النووي | مجموعة النوكليوتيدات (NT) |
| باتنت | الحمض النووي | تسلسلات النوكليوتيدات المستمدة من قسم براءات الاختراع في GenBank |
| PDBNT | الحمض النووي | قاعدة بيانات النوكليوتيد PDB |
| ref_euk_rep_genomes | الحمض النووي | قاعدة بيانات الجينوم التمثيلية حقوق النواة refseq |
| ref_prok_rep_genomes | الحمض النووي | الجينوم التمثيلي لممثل الدفاع عن النفس REFSEQ (يحتوي على مجموعة REFSEQ) |
| ref_viroids_rep_genomes | الحمض النووي | refseq viroids الجينومات التمثيلية |
| ref_viruses_rep_genomes | الحمض النووي | فيروسات Refseq الجينومات التمثيلية |
| RefSeq_RNA | الحمض النووي | تسلسلات مرجعية لنسخة NCBI |
| RefSeq_Select_RNA | الحمض النووي | Refseq حدد تسلسل الحمض النووي الريبي |
| TSA_NT | الحمض النووي | تسلسل تجميع بندقية النصوص (TSA) |
| env_nr | بروتين | البروتينات من مشاريع Metagenomic WGS |
| معلم | بروتين | قاعدة بيانات تاريخية لـ SmartBlast |
| ن | بروتين | جميع ترجمات CDS غير الممتاز+PDB+Swissprot+PIR+PRF باستثناء العينات البيئية من مشاريع WGS |
| PDBAA | بروتين | قاعدة بيانات البروتين PDB |
| باتا | بروتين | تسلسل البروتين المستمدة من تقسيم براءات الاختراع في GenBank |
| RefSeq_protein | بروتين | التسلسلات المرجعية البروتين NCBI |
| refseq_select_prot | بروتين | مرجع اختيار البروتينات |
| Swissprot | بروتين | تسلسل غير زائفة uniprotkb/swissprot |
| TSA_NR | بروتين | تسلسل تجميع بندقية النصوص (TSA) |
| CDD | بروتين | قاعدة بيانات المجال المحفوظة (CDD) عبارة |
يوفر NCBI البيانات الوصفية لقواعد بيانات الانفجار المتاحة في AWS و GCP وموقع NCBI FTP.
من المحتمل أن يؤدي الوصول إلى قواعد البيانات على AWS أو GCP خارج مزود السحابة إلى رسوم الخروج إلى حسابك. إذا لم تكن على مزود السحابة ، فيجب عليك استخدام قواعد البيانات في موقع NCBI FTP.
على AWS و GCP ، يكون الملف في دليل فرعي يعتمد على التاريخ مع قواعد البيانات. للعثور على أحدث دليل فرعي صالح ، اقرأ أولاً s3://ncbi-blast-databases/latest-dir (على AWS) أو gs://blast-db/latest-dir (على GCP). latest-dir هو ملف نصي مع ختم التاريخ (على سبيل المثال ، 2020-09-29-01-05-01) يحدد أحدث دليل. سيكون الدليل المناسب هو URI AWS أو GCP لقواعد بيانات الانفجار (على سبيل المثال ، s3://ncbi-blast-databases/ for AWS) بالإضافة إلى النص في latest-dir . مثال URI ، في AWS ، سيكون s3://ncbi-blast-databases/2020-09-29-01-05-01 . سيكون GCP URI متشابهًا.
يظهر أدناه مقتطف من ملف بيانات التعريف. معظم الحقول لها معاني واضحة. تشتمل الملفات على قاعدة بيانات Blast. يمثل الحقل bytes-total حجم قاعدة بيانات الانفجار الكلي بالبايت ويهدف إلى تحديد مقدار مساحة القرص المطلوبة.
المثال أدناه هو من AWS ، ولكن ملفات البيانات الوصفية على GCP لها نفس التنسيق. Databases on the FTP site are in gzipped tarfiles, one per volume of the BLAST database, so those are listed rather than the individual files.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
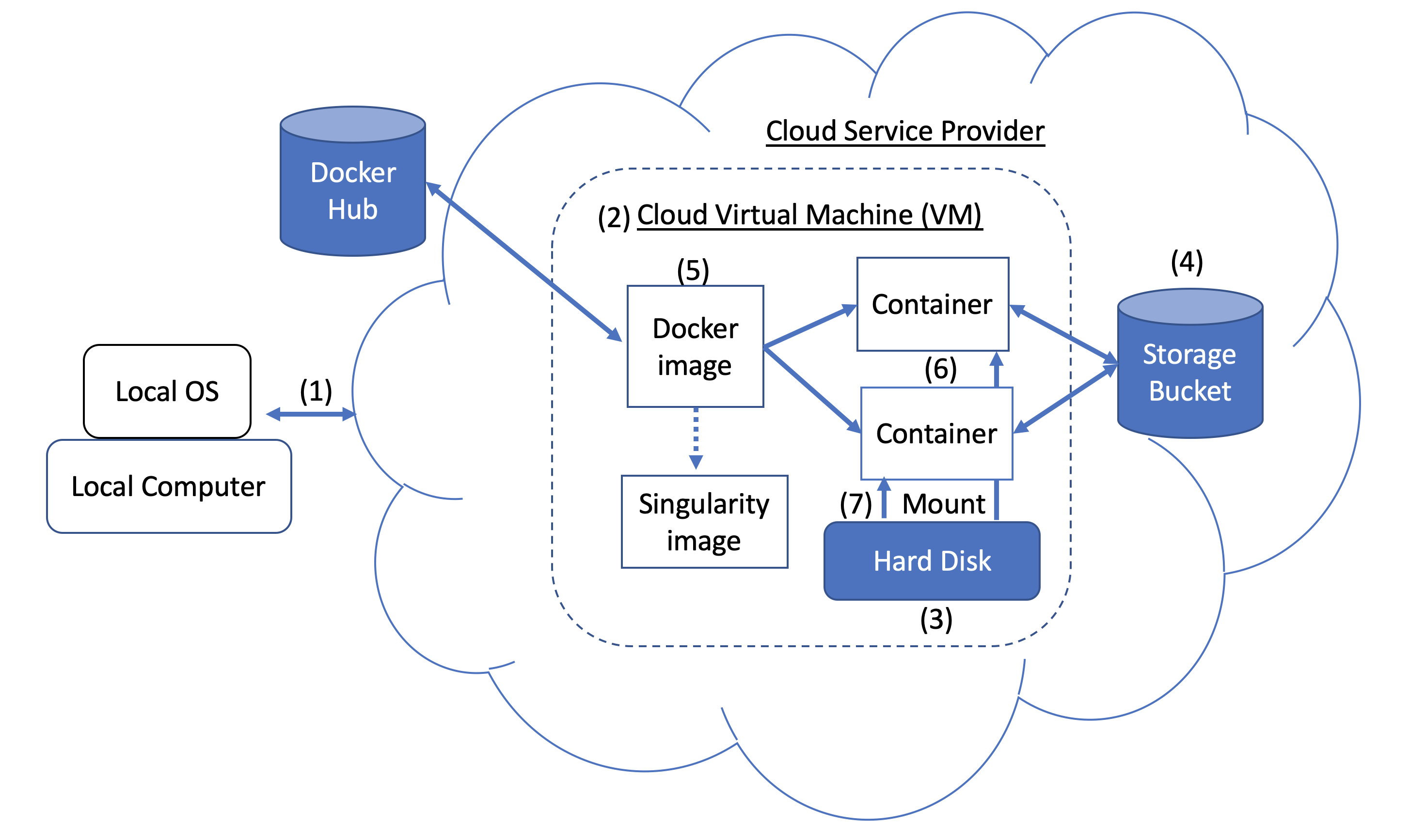 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.