Этот репозиторий содержит документацию для приложений командной строки NCBI BLAST+ на изображении Docker. Мы продемонстрируем, как использовать изображение Docker для запуска анализа взрыва на платформе Google Cloud (GCP) и Amazon Web Services (AWS), используя небольшой базовый пример и более продвинутый пример уровня производства. Некоторые базовые знания команд Unix/Linux и Blast+ полезны для завершения этого учебника.
run Параметры командыrun Структура команд
Национальный центр биотехнологической информации (NCBI) Базовый инструмент поиска локального выравнивания (BLAST) находит районы локального сходства между последовательностями. Программа сравнивает нуклеотидные или белковые последовательности с последовательными базами данных и вычисляет статистическую значимость совпадений. Взрыв может быть использован для вывода функциональных и эволюционных отношений между последовательностями, а также помочь идентифицировать членов семейств генов.
Введенный в 2009 году, Blast+ - это улучшенная версия приложений командной строки BLAST. Для полного описания функций и возможностей Blast+, пожалуйста, обратитесь к руководству пользователя Command Command Line.
Cloud Computing предлагает потенциальную экономию затрат, используя ресурсы по требованию, масштабируемым и эластичным вычислительным ресурсам. В то время как подробное описание различных облачных технологий и преимуществ выходит за рамки этого репозитория, в следующих разделах содержатся информация, необходимая для начала запуска изображения Blast+ Docker на платформе Google Cloud (GCP).
Docker-это инструмент для выполнения виртуализации уровня операционной системы с использованием программных контейнеров. В технологии контейнеризации * изображение представляет собой снимок аналитической среды, инкапсулирующей приложения (ы) и зависимости. Изображение, которое, по сути, представляет собой файл, построенный из списка инструкций, может быть сохранено и легко обмениваться для других, чтобы воссоздать точную аналитическую среду между платформами и операционными системами. Контейнер - это экземпляр времени выполнения изображения. Используя контейнеризацию, пользователи могут обойти часто соответствующие шаги в компиляции, настройке и установке инструмента на основе UNIX, такого как Blast+. В дополнение к портативности, контейнеризация является легким подходом для того, чтобы сделать анализ более доступным, доступным, совместимым, многократным (справедливым) и, в конечном счете, воспроизводимым.
*Существует много инструментов и стандартов контейнеризации, таких как Docker и Singularity. Мы сосредоточимся исключительно на Docker, который считается де -факто стандартом многими в этой области.
Следующие разделы включают инструкции по созданию виртуальной машины Google, установки Docker и выполнения команд Blast+ с помощью изображения Docker.
В этом разделе представлен быстрый проход анализа взрыва в среде Docker в экземпляре Google. Это предназначено в качестве обзора для тех, кто просто хочет понять принципы решения. Если вы работаете с экземплярами Amazon, пожалуйста, перейдите в раздел настройки веб -сервисов Amazon в этой документации. Google Cloud Shell, интерактивная среда оболочки, будет использоваться для этого примера, что позволяет запустить следующий небольшой пример без необходимости выполнения дополнительной настройки, такой как создание биллинговой учетной записи или вычислительный экземпляр. Более подробные описания этапов анализа, альтернативных команд и более продвинутых тем рассматриваются в более поздних разделах этой документации.
Требования: учетная запись Google
Поток задачи: 
Входные данные:
Во -первых, в отдельном окне или вкладке браузера войдите по адресу https://console.cloud.google.com/
Нажмите кнопку Activate Cloud Shell в правом верхнем углу консоли Google Cloud Platform. 
Теперь вы увидите окно сеанса облачной оболочки: 
Следующим шагом является скопирование и вставить команды ниже в вашем сеансе облачной оболочки.
Обратите внимание: в GitHub вы можете использовать свою мышь для копирования; Однако в командной оболочке вы должны использовать свою клавиатуру. В Windows или Unix/Linux используйте сочетание Control+C для копирования и Control+V для вставки. На macOS используйте Command+C для копирования и Command+V для вставки.
Чтобы прокрутить облачную оболочку, включите полос прокрутки в Terminal settings со значком гаечного ключа. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
На этом этапе вы должны увидеть выход на экране. С вашим запросом Blast идентифицировал белковую последовательность p80049.1 как матч с оценкой 14,2 и E-значением 0,96.
Для более широкого анализа рекомендуется использовать флаг -out для сохранения вывода в файл. Например, приложение -out /blast/results/blastp.out к последней команде на шаге 3 выше и просмотрите содержимое этого выходного файла, используя more $HOME/results/blastp.out .
Вы также можете запросить P01349.FSA против PDB, как показано в следующем блоке кода.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
Теперь вы выполнили простую задачу и увидели, как работает Blast+ с Docker. Чтобы узнать о Docker и Blast+ в производственной шкале, перейдите к следующему разделу.
В разделе 2-пошаговое руководство с использованием изображения Blast+ Docker мы будем использовать тот же небольшой пример из предыдущего раздела и обсудим альтернативные подходы, дополнительные полезные команды Docker и Blast+, а также параметры команды Docker. В разделе 3 мы продемонстрируем, как запустить изображение Blast+ Docker в производственной шкале.
Во -первых, вам нужно настроить виртуальную машину Google Cloud Platform (GCP) для анализа.
В настоящее время GCP предлагает кредит в размере 300 долларов США, который истекает 12 месяцев от активации, чтобы стимулировать новых пользователей облаков. Следующие шаги покажут вам, как активировать этот кредит. Вам будет предложено получить информацию о выставлении счетов, но GCP не будет автозарядным вам после завершения испытания; Вы должны выбрать вручную перейти на платную учетную запись.
После входа нажмите «Активировать» , чтобы активировать кредит в размере 300 долларов. 
Введите свою страну, например, Соединенные Штаты, и установите флажок, указывающий, что вы прочитали и принимаете условия обслуживания.
В разделе «Тип учетной записи» выберите «Индивидуальный». (Это может быть предварительно выбрано в вашей учетной записи Google)
Введите свое имя и адрес.
В разделе «Как вы платите», выберите «Автоматические платежи». (Это может быть предварительно выбрано в вашей учетной записи Google) Это указывает на то, что вы будете оплачивать расходы после того, как вы использовали услугу, либо при достижении порога выставления счетов, либо каждые 30 дней, в зависимости от того, что наступает раньше.
В соответствии с «Методом оплаты» выберите «Добавить кредитную или дебетовую карту» и введите информацию о вашей кредитной карте. Вы не будете автоматически заряжаться после завершения испытания. Вы должны выбрать обновление до оплачиваемой учетной записи, прежде чем ваш метод оплаты будет взиматься.
Нажмите «Начни мою бесплатную пробную версию», чтобы закончить регистрацию. Когда этот процесс будет завершен, вы должны увидеть экран приветствия GCP.
На этом этапе вы должны увидеть оценку стоимости для этого экземпляра в правой стороне вашего окна. 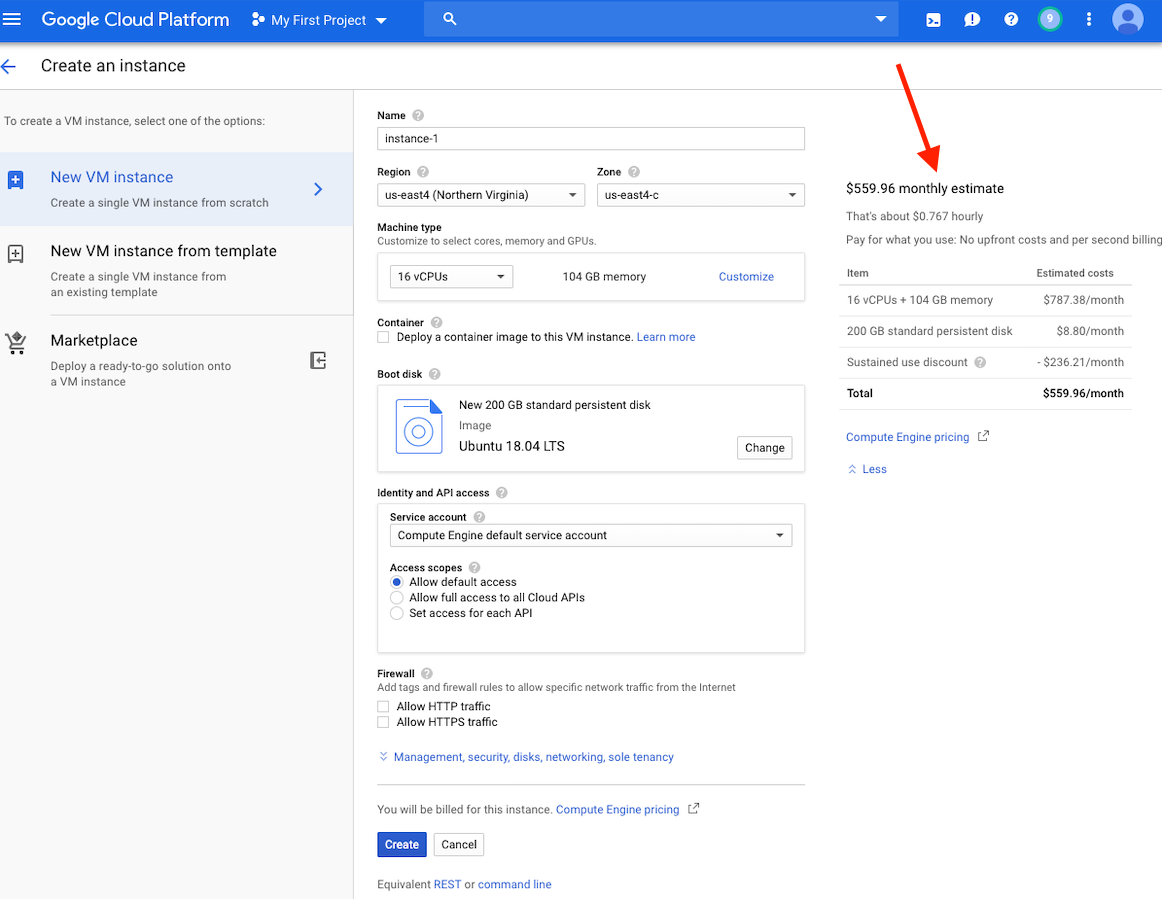
Обратите внимание: создание виртуальной машины в том же регионе, что и хранение, может обеспечить лучшую производительность. Мы рекомендуем создать виртуальную машину в регионе US-East4. Если у вас есть работа, которая займет несколько часов, но менее 24 часов, вы можете воспользоваться преимуществами упреждающих виртуальных машин.
Подробные инструкции по созданию учетной записи GCP и запуска виртуальной машины можно найти здесь.
После того, как вы создали виртуальную машину, вы должны получить к нему доступ к местному компьютеру. Есть много методов для доступа к вашей виртуальной машине, в зависимости от способов, которыми вы хотели бы использовать его. На GCP наиболее простым способом является SSH из браузера.
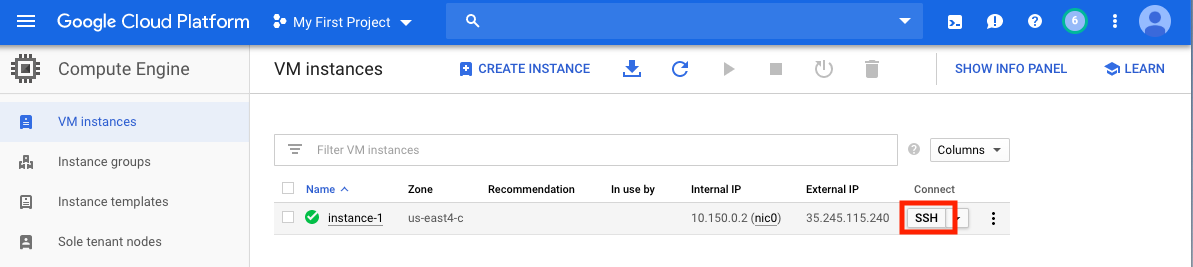
Теперь у вас работает командная оболочка, и вы готовы продолжить.
Не забудьте остановить или удалить виртуальную машину, чтобы предотвратить дополнительные расходы.
В этом разделе мы рассмотрим установку Docker, обсудим различные параметры команды docker run и рассмотрим структуру команды Docker. Мы будем использовать тот же небольшой пример из раздела 1 и исследуем альтернативные подходы при запуске изображения Blast+ Docker. Тем не менее, мы используем реальный экземпляр VM, который обеспечивает большую производительность и функциональность, чем Google Cloud Shell.
Входные данные
В производственной системе Docker должен быть установлен в качестве приложения.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
Чтобы подтвердить правильную установку Docker, запустите команду docker run hello-world . При правильном установке вы должны увидеть «Привет из Docker! ...» (https://docs.docker.com/samples/library/hello-world/)
Этот раздел необязательно.
Ниже приведен список параметров командной строки docker run используемых в этом уроке.
| Имя, короткая рука (если доступно) | Описание |
|---|---|
--rm | Автоматически удалить контейнер, когда он выходит |
--volume , -v | Привязать объем |
--workdir , -w | Рабочий каталог внутри контейнера |
Этот раздел необязательно.
Для этого урока было бы полезно понять структуру команды Docker. Следующая команда состоит из трех частей.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Первая часть команды docker run --rm ncbi/blast -это инструкция по запуску изображения Docker ncbi/blast и удаления контейнера при завершении выполнения.
Вторая часть команды делает данные последовательности запросов доступными в контейнере. Docker Bind Mounts использует -v для установки локальных каталогов в каталогах внутри контейнера и предоставления разрешения на доступ RW (чтение и запись) или RO (только для чтения). Например, предполагая, что ваши предметные последовательности хранятся в каталоге $ home/fasta на локальном хосте, вы можете использовать следующий параметр, чтобы сделать этот каталог доступным внутри контейнера в/blast/fasta в качестве каталога только для чтения -v $HOME/fasta:/blast/fasta:ro . Флаг -w /blast/blastdb_custom устанавливает рабочий каталог внутри контейнера.
Третья часть команды - команда BLAST+. В этом случае он выполняет MakeBlastDB для создания файлов базы данных BLAST.
Вы можете запустить интерактивный сеанс Bash для этого изображения, используя docker run -it ncbi/blast /bin/bash . Для изображения Blast+ Docker исполняемые файлы находятся в папке /Blast /Bin и /Root /edirect и добавлены в путь $.
Для получения дополнительной документации по команде docker run , пожалуйста, обратитесь к документации.
Этот раздел необязательно.
| Команда Docker | Описание |
|---|---|
docker ps -a | Отображает список контейнеров |
docker rm $(docker ps -q -f status=exited) | Удаляет все выходящие контейнеры, если у вас есть не менее 1 выхода контейнера |
docker rm <CONTAINER_ID> | Удаляет контейнер |
docker images | Отображает список изображений |
docker rmi <REPOSITORY (IMAGE_NAME)> | Удаляет изображение |
Этот раздел необязательно.
С помощью этого изображения Docker вы можете запустить Blast+ в изолированном контейнере, способствуя воспроизводимости результатов взрыва. Как пользователь этого изображения Docker, вы ожидаете, что базы данных BLAST и последовательности запросов для запуска взрыва, а также местоположения вне контейнера, чтобы сохранить результаты. Ниже приведен список каталогов, используемых BLAST+. Вы создадите их на шаге 2.
| Каталог | Цель | Примечания |
|---|---|---|
$HOME/blastdb | Хранят базы данных BLAST, предоставленных NCBI | Если вместо этого может быть использована переменная среды $BLASTDB , если установлен на один абсолютный путь (см. Настройку взрыва с помощью переменных среды.) |
$HOME/queries | Хранит предоставленные пользователем последовательности запросов (ы) | |
$HOME/fasta | Хранит предоставленные пользователем последовательности FASTA для создания базы данных BLAST | |
$HOME/results | Результаты взрыва магазинов | rw |
$HOME/blastdb_custom | Хранит предоставленные пользователями базы данных BLAST |
Этот раздел необязательно.
Следующая команда отображает последнюю версию взрыва.
docker run --rm ncbi/blast blastn -version
Добавление тега к имени изображения ( ncbi/blast ) позволяет использовать другую версию BLAST+ (см. В разделе «Поддерживаемые теги и соответствующие заметки о выпуске» для поддерживаемых версий).
Различные версии Blast+ существуют на разных изображениях Docker. Следующая команда будет инициировать загрузку изображения Docker Docker Blast+ версии 2.9.0.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
Например, для использования изображения Docker Blast+ версия 2.9.0 вместо последней версии замените первую часть команды
docker run --rm ncbi/blast с docker run --rm ncbi/blast:2.9.0
Этот раздел необязательно.
В этом примере мы начнем с извлечения последовательностей запросов и баз данных, а затем создаем пользовательскую базу данных BLAST.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Чтобы проверить недавно созданную базу данных BLAST выше, вы можете запустить следующую команду для отображения образцов, длины последовательности и общего имени последовательностей в базе данных.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
В качестве альтернативы вы также можете загрузить предварительные базы данных BLAST из NCBI или ведро NCBI Google.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
Для получения подробного описания update_blastdb.pl , пожалуйста, обратитесь к документации. По умолчанию update_blastdb.pl будет загружать из облака при условии, что вы подключены или из NCBI, если вы не используете поддерживаемый облачный провайдер.
Этот раздел необязательно.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Этот раздел необязательно.
Команда ниже приводит к пути $HOME/blastdb на локальной машине AS /blast/blastdb на контейнере, а blastdbcmd показывает доступные базы данных BLAST в этом месте.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Вы должны увидеть выходной /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
Вы должны увидеть белок выходной /blast/blastdb_custom/nurse-shark-proteins Protein .
При запуске взрыва в контейнере Docker обратите внимание на установки креплений, указанных в команде docker run чтобы сделать вход и выходы доступными. В приведенных ниже примерах первые две крепления обеспечивают доступ к базам данных BLAST, третье крепление обеспечивает доступ к последовательности запросов (ы), а четвертое крепление обеспечивает каталог для сохранения результатов. (Обратите внимание на опции :ro и :rw , которые устанавливают каталоги как только для чтения и чтения соответственно.)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
На этом этапе вы должны увидеть выходной файл $HOME/results/blastp.out . С вашим запросом Blast идентифицировал белковую последовательность p80049.1 как матч с оценкой 14,2 и E-значением 0,96. Чтобы просмотреть содержимое этого выходного файла, используйте команду more $HOME/results/blastp.out .
Не забудьте остановить или удалить виртуальную машину, чтобы предотвратить дополнительные расходы. Вы можете сделать это на консоли GCP, как показано ниже. 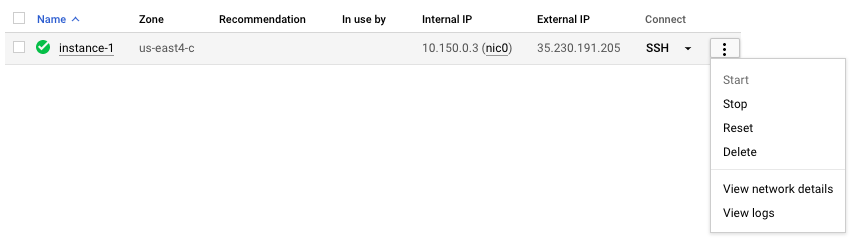
Одним из обещаний облачных вычислений является масштабируемость. В этом разделе мы продемонстрируем, как использовать изображение Blast+ Docker в производственной шкале на платформе Google Cloud. Мы проведем анализ взрыва, аналогичный подходу, описанному в этой публикации, для сравнения выровненных контигов DE novo из бактериального секвенирования 16S-23S с базой данных сбора нуклеотидов (NT).
Чтобы проверить масштабируемость, мы будем использовать входные данные разных размеров, чтобы оценить количество времени для загрузки базы данных сбора нуклеотидов и выполнения поиска взрыва, используя последнюю версию изображения Blast+ Docker. Ожидаемые результаты суммированы в следующих таблицах.
Входные файлы: 28 образцов (мульти-фанаты), содержащие выравнивающие контиги DE novo из публикации.
(Инструкции по загрузке и созданию входных файлов описаны в блоке кода ниже.)
База данных: предварительно форматированная база данных сбора нуклеотидов взрыва, версия 5 (NT): 68,7217 ГБ (с мая 2019 года)
| Имя ввода файла | Содержание файла | Размер файла | Количество последовательностей | Количество нуклеотидов | Ожидаемый выходной размер | |
|---|---|---|---|---|---|---|
| Анализ 1 | Query1.fa | только образец 1 | 59 КБ | 121 | 51,119 | 3,1 ГБ |
| Анализ 2 | Query5.fa | Только образцы 1-5 | 422 КБ | 717 | 375,154 | 10,4 ГБ |
| Анализ 3 | Query.fa | Все 28 образцов | 2,322 МБ | 3798 | 2 069 892 | 47,8 ГБ |
| Тип виртуальной машины/зона | Процессор | Память (ГБ) | Почасовая стоимость* | Скачать NT (мин) | Анализ 1 (мин) | Анализ 2 (мин) | Анализ 3 (мин) | Общая стоимость ** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-EAST4C | 8 | 30 | $ 0,312 | 9 | 22 | - | - | - |
| N1-Standard-16 US-EAST4C | 16 | 60 | $ 0,611 | 9 | 14 | 53 | 205 | $ 2,86 |
| N1-Highmem-16 US-EAST4C | 16 | 104 | $ 0,767 | 9 | 9 | 30 | 143 | $ 2,44 |
| N1-Highmem-16 US-WEST2A | 16 | 104 | $ 0,809 | 11 | 9 | 30 | 147 | $ 2,60 |
| N1-Highmem-16 US-West1b | 16 | 104 | $ 0,674 | 11 | 9 | 30 | 147 | $ 2,17 |
| Blast Warte (Blastn) | - | - | - | - | Поиски превышают текущие ограничения на использование | Поиски превышают текущие ограничения на использование | Поиски превышают текущие ограничения на использование | - |
Все экземпляры GCP настроены с 200 ГБ постоянного стандартного диска.
*Почасовые затраты были предоставлены Google Cloud Platform (май 2019 г.), когда виртуальные машины были созданы и могут быть изменены.
** Общие затраты оценивались с использованием почасовой стоимости и общего времени для загрузки NT и прогона анализа 1, анализа 2 и анализа 3. Оценки используются только для сравнения; Ваши затраты могут варьироваться и несут вашу ответственность за контроль и управление.
Пожалуйста, обратитесь к GCP для получения дополнительной информации о типах машин, регионах и зонах, а также вычислите стоимость.
Обратите внимание, что запуск blastn Binary без указания его параметра -task вызывает алгоритм Megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
Вы завершили весь учебник. На этом этапе, если вам не нужны загруженные данные для дальнейшего анализа, удалите виртуальную машину, чтобы предотвратить дополнительные затраты.
Чтобы удалить экземпляр, следуйте инструкциям в разделе «Остановить экземпляр GCP».
Для получения дополнительной информации, пожалуйста, обратитесь к документации Google Cloud Platform о жизненном цикле.
Чтобы запустить эти примеры, вам понадобится учетная запись Amazon Web Services (AWS). Если у вас его еще нет, вы можете создать учетную запись, которая предоставляет возможность изучать и пробовать услуги AWS бесплатно до указанных пределов для каждой службы. Чтобы начать посещать сайт бесплатного уровня, это потребует действительной кредитной карты, однако она не будет взиматься, если вы вычислите на свободном уровне. При выборе бесплатного уровня продукта, убедитесь, что он находится в категории продукта .
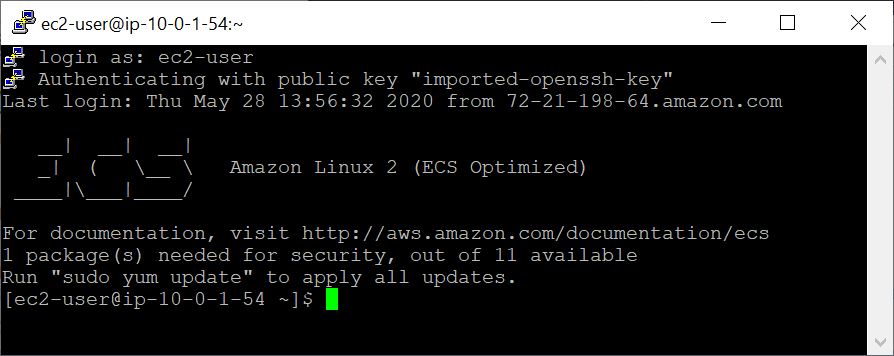
Эти инструкции создают виртуальную машину EC2 на основе изображения машины Amazon (AMI), которое включает Docker и его зависимости.
С созданным виртуальной машиной вы получаете доступ к нему с локального компьютера с помощью SSH. Ваш файл пары ключей / .pem служит вашим учетным данных.
Есть несколько способов установить связь SSH. В списке экземпляров EC2 в консоли AWS выберите Connect , затем следуйте инструкциям для метода соединения, автономного клиента SSH .
Подробные инструкции для подключения к виртуальной реальности Linux можно найти здесь.
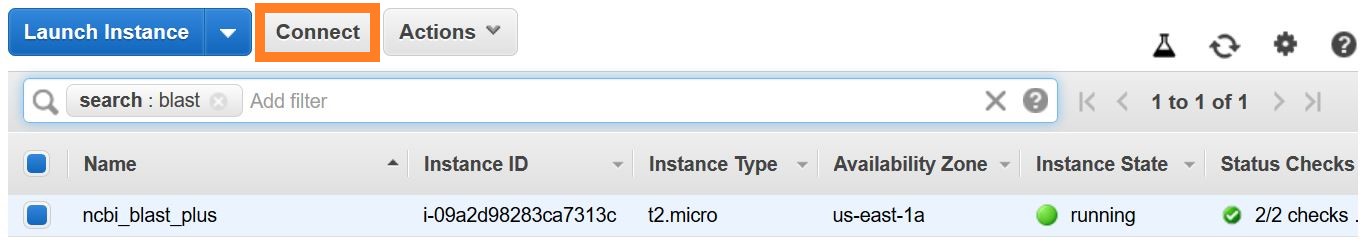
Укажите EC2-USER как имя пользователя, а не root в вашей командной строке SSH или при предложении для входа, укажите EC2-USER в качестве имени пользователя.
В этом примере мы начнем с извлечения последовательностей запросов и баз данных, а затем создаем пользовательскую базу данных BLAST.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Чтобы проверить недавно созданную базу данных BLAST выше, вы можете запустить следующую команду для отображения образцов, длины последовательности и общего имени последовательностей в базе данных.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
При запуске взрыва в контейнере Docker обратите внимание на Mounts ( -v опция), указанную в команде docker run чтобы сделать входные и выходные данные доступными. В приведенных ниже примерах первые две крепления обеспечивают доступ к базам данных BLAST, третье крепление обеспечивает доступ к последовательности запросов (ы), а четвертое крепление обеспечивает каталог для сохранения результатов. (Обратите внимание на опции :ro и :rw , которые устанавливают каталоги как только для чтения и чтения соответственно.)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
На этом этапе вы должны увидеть выходной файл $HOME/results/blastp.out . С вашим запросом Blast идентифицировал белковую последовательность p80049.1 как матч с оценкой 14,2 и E-значением 0,96. Чтобы просмотреть содержимое этого выходного файла, используйте команду more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
Ожидаемый вывод - это список взрывных DBS, включая их имя, описание, размер и последнюю обновленную дату.
Для получения подробного описания update_blastdb.pl , пожалуйста, обратитесь к документации. По умолчанию update_blastdb.pl будет загружать из облака при условии, что вы подключены или из NCBI, если вы не используете поддерживаемый облачный провайдер.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Ожидаемый вывод - это список названий Blast DBS.
Не забудьте остановить или прекратить виртуальную машину, чтобы предотвратить дополнительные расходы. Вы можете сделать это из списка экземпляров EC2 в консоли AWS, как показано ниже.
Этот пример требует многоядерного хоста. Таким образом, вычисления EC2 будут реализованы путем выполнения этого примера. Текущая ставка для используемого типа экземпляра - t2.large - составляет 0,093 долл. США/час.
Эти инструкции создают виртуальную машину EC2 на основе изображения машины Amazon (AMI), которое включает Docker и его зависимости.
С созданным виртуальной машиной вы получаете доступ к нему с локального компьютера с помощью SSH. Ваш файл пары ключей / .pem служит вашим учетным данных.
Есть несколько способов установить связь SSH. В списке экземпляров EC2 в консоли AWS выберите Connect , затем следуйте инструкциям для метода соединения, автономного клиента SSH .
Подробные инструкции для подключения к виртуальной реальности Linux можно найти здесь.
Укажите EC2-USER как имя пользователя, а не root в вашей командной строке SSH или при предложении для входа, укажите EC2-USER в качестве имени пользователя.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
Команда ниже приводит к монтатам (с использованием опции -v ) путь $HOME/blastdb на локальной машине как /blast/blastdb на контейнере, а blastdbcmd показывает доступные базы данных BLAST в этом месте.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Вы должны увидеть выходной /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
На этом этапе вы должны увидеть выходной файл $HOME/results/blastp_pdbaa.out . Чтобы просмотреть содержимое этого выходного файла, используйте команду more $HOME/results/blastp_pdbaa.out .
Одним из способов передачи файлов между вашим локальным компьютером и экземпляром Linux является использование протокола Secure Copy (SCP).
Раздел , передающий файлы на экземпляры Linux из Linux с использованием SCP Руководства пользователя Amazon EC2 для экземпляров Linux, предоставляет подробные инструкции для этого процесса.
NCBI проводит те же базы данных на AWS, GCP и NCBI FTP. В таблице ниже есть список баз данных, текущий по состоянию на ноябрь 2022 года.
Также возможно получить текущий список с командой:
Docker Run -RM NCBI/BLAST UPDATE_BLASTDB.PL -SHOWALL Pretty
или
Update_blastdb.pl -Showall Pretty # после загрузки пакета Blast+.
Как показано выше, Update_blastdb.pl также можно использовать для загрузки этих баз данных. Он автоматически выберет соответствующий ресурс (например, GCP, если вы находитесь в этом поставщике).
Эти базы данных также можно искать с помощью Elasticblast на GCP и AWS.
Доступ к базам данных на AWS или GCP за пределами облачного провайдера, вероятно, приведет к выходу в вашу учетную запись. Если вы не находитесь в облачном провайдере, вам следует использовать базы данных на сайте NCBI FTP.
| Имя | Тип | Заголовок |
|---|---|---|
| 16S_RIBOSOMAL_RNA | ДНК | 16S Рибосомальная РНК (штаммы бактерий и архей) |
| 18s_fungal_ sequences | ДНК | 18S рибосомальные последовательности РНК (SSU) из типа грибов и эталонного материала |
| 28S_FUNGAL_SESECENCES | ДНК | 28s рибосомные РНК -последовательности (LSU) из типа грибов и эталонного материала |
| Бетакоронавирус | ДНК | Бетакоронавирус |
| Gcf_000001405.38_top_level | ДНК | Homo sapiens grch38.p12 [gcf_000001405.38] хромосомы плюс непредвиденные и нереализованные каркасы |
| Gcf_000001635.26_top_level | ДНК | Mus musculus grcm38.p6 [gcf_000001635.26] хромосомы плюс непредвиденные и нереализованные каркасы |
| Ith_refseq_fungi | ДНК | Внутренняя транскрибированная область проставки (ITS) из типа грибов и справочного материала |
| Its_eukaryote_ sequences | ДНК | Его эукариот взрыв |
| Lsu_eukaryote_rrna | ДНК | Большая субъединица рибосомная нуклеиновая кислота для эукариот |
| Lsu_prokaryote_rrna | ДНК | Большая субъединица рибосомная нуклеиновая кислота для прокариот |
| Ssu_eukaryote_rrna | ДНК | Небольшая субъединица рибосомная нуклеиновая кислота для эукариот |
| env_nt | ДНК | Образцы окружающей среды |
| н.д. | ДНК | Коллекция нуклеотидов (NT) |
| по -прежнему | ДНК | Нуклеотидные последовательности, полученные из патентного деления GenBank |
| pdbnt | ДНК | База данных нуклеотидов PDB |
| ref_euk_rep_genomes | ДНК | Refseq Eukaryotic Rebritiate Genome Database Genome |
| ref_prok_rep_genomes | ДНК | Refseq Prokaryote Prestrimative Genomes (содержит сборку Refseq) |
| ref_viroids_rep_genomes | ДНК | Refseq viroids |
| ref_viruses_rep_genomes | ДНК | Refseq Viruses Представительные геномы |
| refseq_rna | ДНК | Справочные последовательности транскриптов NCBI |
| refseq_select_rna | ДНК | Refseq выбирать последовательности РНК |
| tsa_nt | ДНК | Последовательности сборочных сборов транскриптома (TSA) |
| env_nr | Белок | Белки из метагеномных проектов WGS |
| достопримечательность | Белок | Знаковая база данных для SmartBlast |
| нр | Белок | Все не Redendant Genbank CDS Translations+PDB+Swissprot+PIR+PRF, исключая образцы окружающей среды из проектов WGS |
| Pdbaa | Белок | База данных белков PDB |
| Патаа | Белок | Белковые последовательности, полученные из патентного деления GenBank |
| Refseq_protein | Белок | Справочные последовательности белка NCBI |
| refseq_select_prot | Белок | Refseq Select Белки |
| Swissprot | Белок | Неограниченные последовательности Uniprotkb/Swissprot |
| TSA_NR | Белок | Последовательности сборочных сборов транскриптома (TSA) |
| CDD | Белок | База данных консервативной области (CDD) представляет собой набор хорошо аннотированных моделей выравнивания нескольких последовательностей, представленных в качестве специфичных для положения матриц баллов |
NCBI предоставляет метаданные для доступных баз данных BLAST на AWS, GCP и FTP NCBI.
Доступ к базам данных на AWS или GCP за пределами облачного провайдера, вероятно, приведет к выходу в вашу учетную запись. Если вы не находитесь в облачном провайдере, вам следует использовать базы данных на сайте NCBI FTP.
На AWS и GCP файл находится в зависимой от даты подкаталерии с базами данных. Чтобы найти последнюю достоверную подкаталог, сначала прочитайте s3://ncbi-blast-databases/latest-dir (on aws) или gs://blast-db/latest-dir (на GCP). latest-dir -это текстовый файл с маркой даты (например, 2020-09-29-01-05-01) с указанием самого последнего каталога. Правильным каталогом будет базовый URI AWS или GCP для баз данных BLAST (например, s3://ncbi-blast-databases/ для AWS) плюс текст в файле latest-dir . Примером URI в AWS будет s3://ncbi-blast-databases/2020-09-29-01-05-01 . GCP URI будет похожим.
Выдержка из файла метаданных показана ниже. Большинство полей имеют очевидные значения. Файлы составляют базу данных BLAST. Поле bytes-total представляет общий размер базы данных BLAST в байтах и предназначено для указания того, сколько дискового пространства требуется.
Пример ниже - от AWS, но файлы метаданных на GCP имеют одинаковый формат. Databases on the FTP site are in gzipped tarfiles, one per volume of the BLAST database, so those are listed rather than the individual files.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
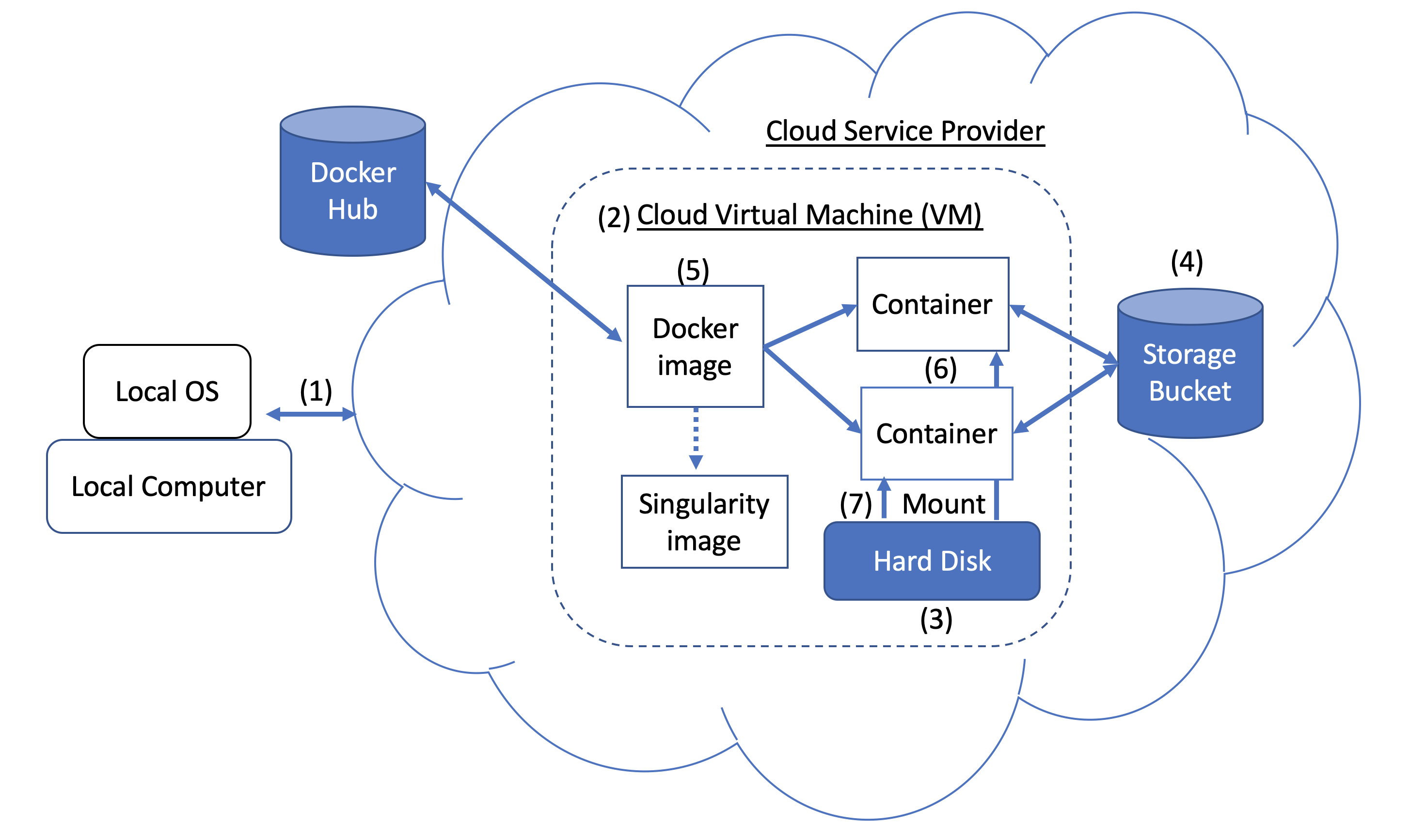 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.