Este repositorio contiene documentación para las aplicaciones de línea de comandos NCBI BLAST+ en una imagen Docker. Demostraremos cómo usar la imagen Docker para ejecutar el análisis de explosión en la plataforma de Google Cloud (GCP) y Amazon Web Services (AWS) utilizando un pequeño ejemplo básico y un ejemplo de nivel de producción más avanzado. Algunos conocimientos básicos de los comandos UNIX/Linux y Blast+ son útiles para completar este tutorial.
run de Dockerrun de Docker
La herramienta básica de búsqueda de alineación local del Centro Nacional de Biotecnología (NCBI) encuentra regiones de similitud local entre secuencias. El programa compara secuencias de nucleótidos o proteínas con bases de datos de secuencia y calcula la importancia estadística de las coincidencias. La explosión puede usarse para inferir relaciones funcionales y evolutivas entre secuencias, así como para ayudar a identificar miembros de familias de genes.
Introducido en 2009, BLAST+ es una versión mejorada de las aplicaciones de la línea de comandos de Blast. Para obtener una descripción completa de las características y capacidades de BLAST+, consulte el Manual del usuario de las aplicaciones de la línea de comandos de BLAST.
La computación en la nube ofrece ahorros potenciales de costos mediante el uso de recursos computacionales a pedido, escalables y elásticos. Si bien una descripción detallada de varias tecnologías y beneficios en la nube está fuera del alcance de este repositorio, las siguientes secciones contienen información necesaria para comenzar a ejecutar la imagen BLAST+ Docker en la plataforma de Google Cloud (GCP).
Docker es una herramienta para realizar la virtualización de nivel de sistema operativo utilizando contenedores de software. En la tecnología de contenedores * , una imagen es una instantánea de un entorno analítico que encapsula la (s) aplicación (s) y dependencias. Una imagen, que es esencialmente un archivo creado a partir de una lista de instrucciones, se puede guardar y compartir fácilmente para que otros recrearan el entorno analítico exacto en plataformas y sistemas operativos. Un contenedor es una instancia de tiempo de ejecución de una imagen. Mediante el uso de contenedores, los usuarios pueden evitar los pasos a menudo complicados para compilar, configurar e instalar una herramienta basada en UNIX como BLAST+. Además de la portabilidad, la contenedorización es un enfoque liviano para hacer que el análisis sea más búsqueda, accesible, interoperable, reutilizable (justo) y, en última instancia, reproducible.
*Hay muchas herramientas y estándares de contenedores, como Docker y Singularity. Nos centraremos únicamente en Docker, que es considerado el estándar de facto por muchos en el campo.
Las siguientes secciones incluyen instrucciones para crear una máquina virtual de Google, instalar Docker y ejecutar comandos BLAST+ usando la imagen Docker.
Esta sección proporciona una ejecución rápida de un análisis de explosión en el entorno Docker en una instancia de Google. Esto se pretende como una descripción general para aquellos que solo quieren comprender los principios de la solución. Si trabaja con instancias de Amazon, realice la sección Configuración de servicios web de Amazon de esta documentación. Google Cloud Shell, un entorno de shell interactivo, se utilizará para este ejemplo, lo que permite ejecutar el siguiente pequeño ejemplo sin tener que realizar una configuración adicional, como crear una cuenta de facturación o una instancia de cálculo. Las descripciones más detalladas de los pasos de análisis, los comandos alternativos y los temas más avanzados se cubren en las secciones posteriores de esta documentación.
Requisitos: una cuenta de Google
Flujo de la tarea:
Datos de entrada:
Primero, en una ventana o pestaña de navegador separada, inicie sesión en https://console.cloud.google.com/
Haga clic en el botón Activar Shell Cloud en la esquina superior derecha de la consola de la plataforma de Google Cloud. 
Ahora verá su ventana de sesión de shell de nube: 
El siguiente paso es copiar y pegar los comandos a continuación en su sesión de shell de nube.
Tenga en cuenta: en GitHub puede usar su mouse para copiar; Sin embargo, en el shell de comando debe usar su teclado. En Windows o Unix/Linux, use el Control+C para copiar y Control+V para pegar. En macOS, use Command+C para copiar y Command+V para pegar.
Para desplazarse en la carcasa de la nube, habilite la barra de desplazamiento en Terminal settings con el icono de la llave. 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
En este punto, debería ver la salida en la pantalla. Con su consulta, BLAST identificó la secuencia de proteínas P80049.1 como una coincidencia con una puntuación de 14.2 y un valor electrónico de 0.96.
Para un análisis más grande, se recomienda usar el indicador -out para guardar la salida en un archivo. Por ejemplo, append -out /blast/results/blastp.out al último comando en el paso 3 anterior y vea el contenido de este archivo de salida usando more $HOME/results/blastp.out .
También puede consultar P01349.FSA contra el PDB como se muestra en el siguiente bloque de código.
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
Ahora ha completado una tarea simple y ha visto cómo funciona Blast+ con Docker. Para aprender sobre Docker y BLAST+ a escala de producción, proceda a la siguiente sección.
En la Sección 2: una guía paso a paso utilizando la imagen BLAST+ Docker, utilizaremos el mismo pequeño ejemplo de la sección anterior y discutiremos enfoques alternativos, comandos adicionales de Docker y BLAST+, y opciones y estructuras de comando Docker. En la Sección 3, demostraremos cómo ejecutar la imagen BLAST+ Docker a escala de producción.
Primero, debe configurar una máquina virtual de Google Cloud Platform (GCP) para el análisis.
GCP actualmente ofrece un crédito de $ 300, que expira 12 meses desde la activación, para incentivar a los nuevos usuarios de la nube. Los siguientes pasos le mostrarán cómo activar este crédito. Se le solicitará información de facturación, pero GCP no le cobrará automáticamente una vez que finalice la prueba; Debe optar por actualizar manualmente a una cuenta pagada.
Después de iniciar sesión, haga clic en activar para activar el crédito de $ 300.
Ingrese a su país, por ejemplo, Estados Unidos, y marque la casilla que indique que ha leído y acepta los términos de servicio.
En "Tipo de cuenta", seleccione "Individual". (Esto puede ser preseleccionado en su cuenta de Google)
Ingrese su nombre y dirección.
En "Cómo paga", seleccione "Pagos automáticos". (Esto puede ser revelado en su cuenta de Google) Esto indica que pagará los costos después de haber usado el servicio, ya sea cuando haya alcanzado su umbral de facturación o cada 30 días, lo que ocurra primero.
Según el "Método de pago", seleccione "Agregar una tarjeta de crédito o débito" e ingrese la información de su tarjeta de crédito. No se le cobrará automáticamente una vez que finalice la prueba. Debe optar por actualizar a una cuenta paga antes de que se le cobre su método de pago.
Haga clic en "Iniciar mi prueba gratuita" para finalizar el registro. Cuando se complete este proceso, debería ver una pantalla de bienvenida GCP.
En este punto, debería ver una estimación de costos para esta instancia en el lado derecho de su ventana. 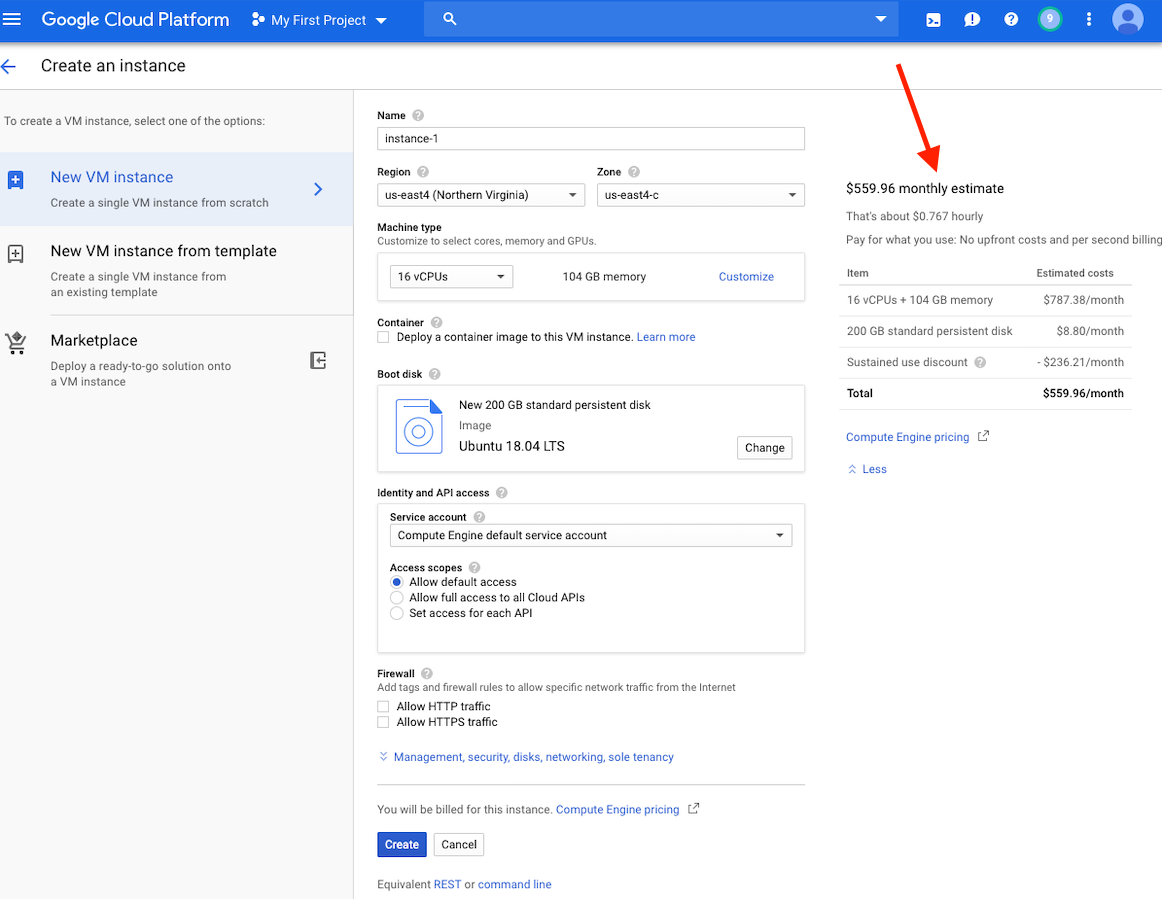
Tenga en cuenta: crear una VM en la misma región que el almacenamiento puede proporcionar un mejor rendimiento. Recomendamos crear una VM en la región US-East4. Si tiene un trabajo que llevará varias horas, pero menos de 24 horas, puede aprovechar las máquinas virtuales preventibles.
Las instrucciones detalladas para crear una cuenta GCP y el lanzamiento de una VM pueden encontrar aquí.
Una vez que haya creado su VM, debe acceder a ella desde su computadora local. Hay muchos métodos para acceder a su VM, dependiendo de las formas en que le gustaría usarlo. En el GCP, la forma más directa es SSH desde el navegador.
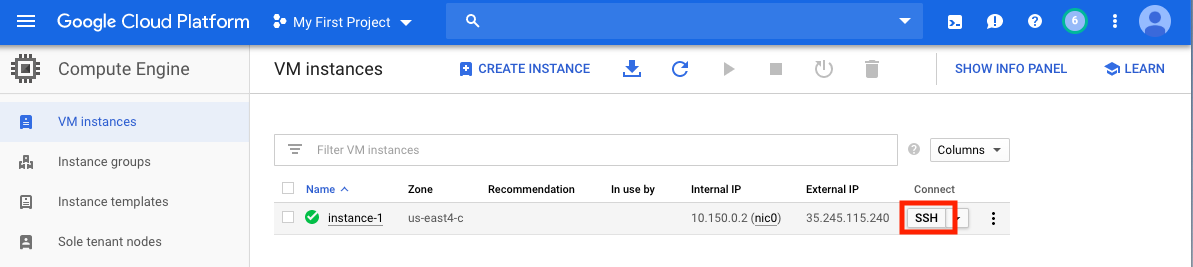
Ahora tiene un shell de comando en ejecución y está listo para continuar.
Recuerde detener o eliminar la VM para evitar incurrir en costos adicionales.
En esta sección, cubriremos la instalación de Docker, discutiremos varias opciones de comando docker run y examinaremos la estructura de un comando Docker. Usaremos el mismo pequeño ejemplo de la Sección 1 y exploraremos enfoques alternativos en la ejecución de la imagen BLAST+ Docker. Sin embargo, estamos utilizando una instancia de VM real, que proporciona un mayor rendimiento y funcionalidad que el Shell de Google Cloud.
Datos de entrada
En un sistema de producción, Docker debe instalarse como una aplicación.
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
Para confirmar la instalación correcta de Docker, ejecute el comando docker run hello-world . Si se instala correctamente, debería ver "¡Hola desde Docker! ..." (https://docs.docker.com/samples/library/hello-world/)
Esta sección es opcional.
A continuación se muestra una lista de las opciones de línea de comandos docker run en este tutorial.
| Nombre, corta (si está disponible) | Descripción |
|---|---|
--rm | Eliminar automáticamente el contenedor cuando salga |
--volume , -v | Atar montura un volumen |
--workdir , -w | Directorio de trabajo dentro del contenedor |
Esta sección es opcional.
Para este tutorial, sería útil comprender la estructura de un comando Docker. El siguiente comando consta de tres partes.
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
La primera parte del comando docker run --rm ncbi/blast es una instrucción para ejecutar la imagen Docker ncbi/blast y eliminar el contenedor cuando se complete la ejecución.
La segunda parte del comando hace que los datos de la secuencia de consulta sean accesibles en el contenedor. Docker Bind Mounts usa -v para montar los directorios locales a los directorios dentro del contenedor y proporcionar permiso de acceso RW (leer y escribir) o RO (solo leer). Por ejemplo, suponiendo que las secuencias de su sujeto se almacenen en el directorio $ Home/FASTA en el host local, puede usar el siguiente parámetro para que ese directorio sea accesible dentro del contenedor en/Blast/FASTA como directorio de solo lectura -v $HOME/fasta:/blast/fasta:ro . La bandera -w /blast/blastdb_custom establece el directorio de trabajo dentro del contenedor.
La tercera parte del comando es el comando BLAST+. En este caso, está ejecutando MakebLastDB para crear archivos de base de datos BLAST.
Puede iniciar una sesión de Bash interactiva para esta imagen usando docker run -it ncbi/blast /bin/bash . Para la imagen BLAST+ Docker, los ejecutables están en la carpeta /Blast /Bin y /Root /Edirect y se agregan a la ruta variable $.
Para obtener documentación adicional en el comando docker run , consulte la documentación.
Esta sección es opcional.
| Comando Docker | Descripción |
|---|---|
docker ps -a | Muestra una lista de contenedores |
docker rm $(docker ps -q -f status=exited) | Elimina todos los contenedores excitados, si tiene al menos 1 contenedor salido |
docker rm <CONTAINER_ID> | Elimina un contenedor |
docker images | Muestra una lista de imágenes |
docker rmi <REPOSITORY (IMAGE_NAME)> | Elimina una imagen |
Esta sección es opcional.
Con esta imagen de Docker, puede ejecutar Blast+ en un contenedor aislado, facilitando la reproducibilidad de los resultados de la explosión. Como usuario de esta imagen de Docker, se espera que proporcione bases de datos de explosión y secuencias de consultas para ejecutar BLAST, así como una ubicación fuera del contenedor para guardar los resultados. La siguiente es una lista de directorios utilizados por BLAST+. Los creará en el paso 2.
| Directorio | Objetivo | Notas |
|---|---|---|
$HOME/blastdb | Almacena bases de datos de explosión proporcionadas por NCBI | Si se establece en una sola ruta absoluta , la variable de entorno $BLASTDB podría usarse en su lugar (consulte Configuración de la explosión a través de las variables de entorno). |
$HOME/queries | Almacena secuencia (s) de consultas proporcionadas por el usuario | |
$HOME/fasta | Almacena secuencias FASTA proporcionadas por el usuario para crear una base de datos de explosión de explosión | |
$HOME/results | Resultados de la explosión de las tiendas | Montar con permisos de rw |
$HOME/blastdb_custom | Almacena bases de datos de explosión proporcionadas por el usuario |
Esta sección es opcional.
El siguiente comando muestra la última versión de explosión.
docker run --rm ncbi/blast blastn -version
Agregar una etiqueta al nombre de la imagen ( ncbi/blast ) le permite usar una versión diferente de la sección BLAST+ (consulte "Etiquetas compatibles y notas de versión respectivas" para versiones compatibles).
Existen diferentes versiones de Blast+ en diferentes imágenes de Docker. El siguiente comando iniciará la descarga de la imagen de Docker BLAST+ Versión 2.9.0.
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
Por ejemplo, para usar la imagen de Docker BLAST+ Versión 2.9.0 en lugar de la última versión, reemplace la primera parte del comando
docker run --rm ncbi/blast con docker run --rm ncbi/blast:2.9.0
Esta sección es opcional.
En este ejemplo, comenzaremos obteniendo secuencias de consultas y bases de datos y luego crearemos una base de datos de explosión personalizada.
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Para verificar la recién creada base de datos de explosión anterior, puede ejecutar el siguiente comando para mostrar las accesiones, la longitud de la secuencia y el nombre común de las secuencias en la base de datos.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Como alternativa, también puede descargar bases de datos BLAST preformatadas de NCBI o el Bucket de almacenamiento de Google NCBI.
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
Para obtener una descripción detallada de update_blastdb.pl , consulte la documentación. De forma predeterminada, update_blastdb.pl se descargará desde la nube siempre que esté conectado, o desde NCBI si no está utilizando un proveedor de nube compatible.
Esta sección es opcional.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
Esta sección es opcional.
El comando a continuación monta la ruta $HOME/blastdb en la máquina local como /blast/blastdb en el contenedor, y blastdbcmd muestra las bases de datos de explosión disponibles en esta ubicación.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Debería ver una /blast/blastdb/pdbaa Protein .
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
Debería ver una /blast/blastdb_custom/nurse-shark-proteins Protein .
Cuando se ejecute BLAST en un contenedor Docker, tenga en cuenta las monturas especificadas en el comando docker run para que la entrada y las salidas sean accesibles. En los ejemplos a continuación, las dos primeras monturas proporcionan acceso a las bases de datos de explosión, el tercer soporte proporciona acceso a la (s) secuencia (s) de consulta y el cuarto soporte proporciona un directorio para guardar los resultados. (Tenga en cuenta las opciones :ro y :rw , que montan los directorios como solo lectura y de lectura respectivamente).
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
En este punto, debería ver el archivo de salida $HOME/results/blastp.out . Con su consulta, BLAST identificó la secuencia de proteínas P80049.1 como una coincidencia con una puntuación de 14.2 y un valor electrónico de 0.96. Para ver el contenido de este archivo de salida, use el comando more $HOME/results/blastp.out .
Recuerde detener o eliminar la VM para evitar incurrir en costos adicionales. Puede hacer esto en la consola GCP como se muestra a continuación. 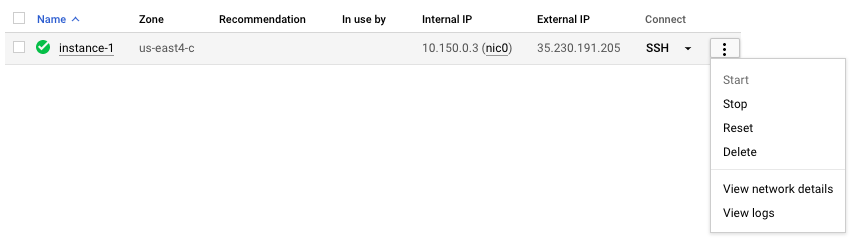
Una de las promesas de la computación en la nube es la escalabilidad. En esta sección, demostraremos cómo usar la imagen BLAST+ Docker a escala de producción en la plataforma de Google Cloud. Realizaremos un análisis de explosión similar al enfoque descrito en esta publicación para comparar contigs alineados de novo de la secuenciación bacteriana 16S-23S contra la base de datos de la recolección de nucleótidos (NT).
Para probar la escalabilidad, utilizaremos entradas de diferentes tamaños para estimar la cantidad de tiempo para descargar la base de datos de la colección de nucleótidos y ejecutar la búsqueda de explosión utilizando la última versión de la imagen BLAST+ Docker. Los resultados esperados se resumen en las siguientes tablas.
Archivos de entrada: 28 muestras (archivos multifasta) que contienen contigs alineados de novo de la publicación.
(Las instrucciones para descargar y crear los archivos de entrada se describen en el bloque de código a continuación).
Base de datos: Base de datos de colección de nucleótidos de explosión previamente formateada, versión 5 (NT): 68.7217 GB (de mayo de 2019)
| Nombre del archivo de entrada | File de contenido | Tamaño de archivo | Número de secuencias | Número de nucleótidos | Tamaño de salida esperado | |
|---|---|---|---|---|---|---|
| Análisis 1 | consulta1.fa | Solo muestra 1 | 59 kb | 121 | 51,119 | 3.1 GB |
| Análisis 2 | consulta 5.fa | Solo muestras 1-5 | 422 kb | 717 | 375,154 | 10.4 GB |
| Análisis 3 | consulta.fa | las 28 muestras | 2.322 MB | 3798 | 2,069,892 | 47.8 GB |
| Tipo/zona de VM | UPC | Memoria (GB) | Costo por hora* | Descargar NT (min) | Análisis 1 (min) | Análisis 2 (min) | Análisis 3 (min) | Costo total ** |
|---|---|---|---|---|---|---|---|---|
| N1-Estandard-8 US-East4c | 8 | 30 | $ 0.312 | 9 | 22 | - | - | - |
| N1-Estandard-16 US-East4c | 16 | 60 | $ 0.611 | 9 | 14 | 53 | 205 | $ 2.86 |
| N1-Highmem-16 US-East4c | 16 | 104 | $ 0.767 | 9 | 9 | 30 | 143 | $ 2.44 |
| N1-Highmem-16 US-West2a | 16 | 104 | $ 0.809 | 11 | 9 | 30 | 147 | $ 2.60 |
| N1-Highmem-16 US-West1b | 16 | 104 | $ 0.674 | 11 | 9 | 30 | 147 | $ 2.17 |
| Sitio web de BLAST (BLASTN) | - | - | - | - | Las búsquedas exceden las restricciones actuales sobre el uso | Las búsquedas exceden las restricciones actuales sobre el uso | Las búsquedas exceden las restricciones actuales sobre el uso | - |
Todas las instancias de GCP están configuradas con 200 GB de disco estándar persistente.
*Los costos por hora fueron proporcionados por Google Cloud Platform (mayo de 2019) cuando se crearon máquinas virtuales y están sujetas a cambios.
** Los costos totales se estimaron utilizando el costo por hora y el tiempo total para descargar el análisis NT y Ejecutar 1, el análisis 2 y el análisis 3. Las estimaciones se utilizan solo para comparación; Sus costos pueden variar y es su responsabilidad de monitorear y administrar.
Consulte GCP para obtener más información sobre tipos de máquinas, regiones y zonas, y calcular el costo.
Tenga en cuenta que ejecutar el binario blastn sin especificar su parámetro -task invoca el algoritmo Megablast.
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
Has completado todo el tutorial. En este punto, si no necesita los datos descargados para un análisis posterior, elimine la VM para evitar incurrir en costos adicionales.
Para eliminar una instancia, siga las instrucciones en la sección Detener la instancia GCP.
Para obtener información adicional, consulte la documentación de Google Cloud Platform en el ciclo de vida de las instancias.
Para ejecutar estos ejemplos, necesitará una cuenta de Amazon Web Services (AWS). Si aún no tiene uno, puede crear una cuenta que brinde la capacidad de explorar y probar los servicios de AWS de forma gratuita hasta los límites especificados para cada servicio. Para comenzar, visite el sitio de nivel gratuito, esto requerirá una tarjeta de crédito válida, sin embargo, no se le cobrará si calcula dentro del nivel gratuito. Al elegir un producto de nivel gratuito, asegúrese de que esté en la categoría de productos .
Estas instrucciones crean una VM EC2 basada en una imagen de Amazon Machine (AMI) que incluye Docker y sus dependencias.
Con la VM creada, accede desde su computadora local usando SSH. Su archivo de pares de clave / .pem sirve como su credencial.
Hay varias formas de establecer una conexión SSH. Desde la lista de instancias de EC2 en la consola AWS, seleccione Connect , luego siga las instrucciones para el método de conexión de un cliente SSH independiente .
Las instrucciones detalladas para conectarse a una VM Linux se pueden encontrar aquí.
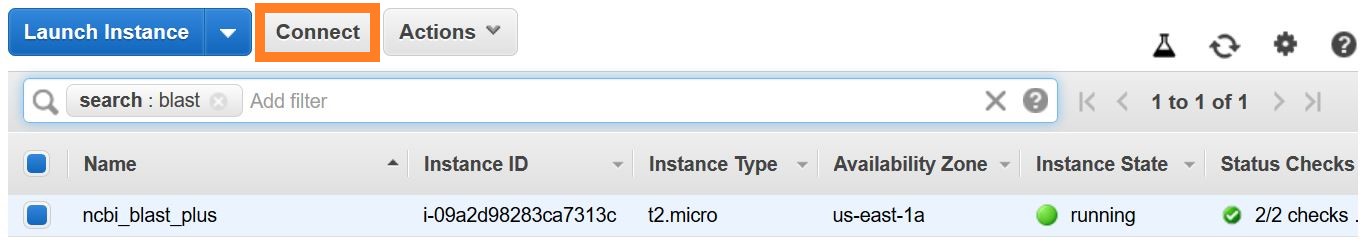
Especifique el usuario de EC2 como el nombre de usuario, en lugar de root en su línea de comando SSH o cuando se le solicite iniciar sesión, especifique el usuario de EC2 como el nombre de usuario.
En este ejemplo, comenzaremos obteniendo secuencias de consultas y bases de datos y luego crearemos una base de datos de explosión personalizada.
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
Para verificar la recién creada base de datos de explosión anterior, puede ejecutar el siguiente comando para mostrar las accesiones, la longitud de la secuencia y el nombre común de las secuencias en la base de datos.
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
Al ejecutar BLAST en un contenedor Docker, tenga en cuenta las monturas ( -v opción) especificadas en el comando docker run para que las entradas y las salidas sean accesibles. En los ejemplos a continuación, las dos primeras monturas proporcionan acceso a las bases de datos de explosión, el tercer soporte proporciona acceso a la (s) secuencia (s) de consulta y el cuarto soporte proporciona un directorio para guardar los resultados. (Tenga en cuenta las opciones :ro y :rw , que montan los directorios como solo lectura y de lectura respectivamente).
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
En este punto, debería ver el archivo de salida $HOME/results/blastp.out . Con su consulta, BLAST identificó la secuencia de proteínas P80049.1 como una coincidencia con una puntuación de 14.2 y un valor electrónico de 0.96. Para ver el contenido de este archivo de salida, use el comando more $HOME/results/blastp.out .
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
La salida esperada es una lista de DBS BLAST, que incluye su nombre, descripción, tamaño y última fecha actualizada.
Para obtener una descripción detallada de update_blastdb.pl , consulte la documentación. De forma predeterminada, update_blastdb.pl se descargará desde la nube siempre que esté conectado, o desde NCBI si no está utilizando un proveedor de nube compatible.
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
El resultado esperado es una lista de los nombres de BLAST DBS.
Recuerde detener o terminar la VM para evitar incurrir en costos adicionales. Puede hacer esto desde la lista de instancias de EC2 en la consola AWS como se muestra a continuación.
Este ejemplo requiere un host de múltiples núcleos. Como tal, los cargos de cómputo de EC2 se realizarán ejecutando este ejemplo. La tarifa actual para el tipo de instancia utilizado - T2.Large - es $ 0.093/h.
Estas instrucciones crean una VM EC2 basada en una imagen de Amazon Machine (AMI) que incluye Docker y sus dependencias.
Con la VM creada, accede desde su computadora local usando SSH. Su archivo de pares de clave / .pem sirve como su credencial.
Hay varias formas de establecer una conexión SSH. Desde la lista de instancias de EC2 en la consola AWS, seleccione Connect , luego siga las instrucciones para el método de conexión de un cliente SSH independiente .
Las instrucciones detalladas para conectarse a una VM Linux se pueden encontrar aquí.
Especifique el usuario de EC2 como el nombre de usuario, en lugar de root en su línea de comando SSH o cuando se le solicite iniciar sesión, especifique el usuario de EC2 como el nombre de usuario.

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
El comando a continuación se monta (usando la opción -v ) la ruta $HOME/blastdb en la máquina local como /blast/blastdb en el contenedor, y blastdbcmd muestra las bases de datos de explosión disponibles en esta ubicación.
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
Debería ver una /blast/blastdb/pdbaa Protein .
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
En este punto, debería ver el archivo de salida $HOME/results/blastp_pdbaa.out . Para ver el contenido de este archivo de salida, use el comando more $HOME/results/blastp_pdbaa.out .
Una forma de transferir archivos entre su computadora local y una instancia de Linux es usar el Protocolo de copia segura (SCP).
La sección Transferencia de archivos a instancias de Linux desde Linux utilizando SCP de la Guía del usuario de Amazon EC2 para instancias de Linux proporciona instrucciones detalladas para este proceso.
El NCBI aloja las mismas bases de datos en AWS, GCP y el sitio NCBI FTP. La siguiente tabla tiene la lista de bases de datos actuales a noviembre de 2022.
También es posible obtener la lista actual con el comando:
Docker Run - -RM NCBI/Blast Update_blastdb.pl --showall Pretty
o
update_blastdb.pl --showall Pretty # Después de descargar el paquete BLAST+.
Como se muestra arriba, Update_blastdb.pl también se puede usar para descargar estas bases de datos. Seleccionará automáticamente el recurso apropiado (por ejemplo, GCP si está dentro de ese proveedor).
Estas bases de datos también se pueden buscar con Elasticblast en GCP y AWS.
Acceder a las bases de datos en AWS o GCP fuera del proveedor de la nube probablemente dará como resultado cargos de salida a su cuenta. Si no está en el proveedor de la nube, debe usar las bases de datos en el sitio NCBI FTP.
| Nombre | Tipo | Título |
|---|---|---|
| 16S_RIBOSOMAL_RNA | ADN | ARN ribosómico 16S (bacterias y cepas tipo arquea) |
| 18S_Fungal_Sequences | ADN | 18S secuencias de ARN ribosómico (SSU) del tipo de hongos y material de referencia |
| 28S_Fungal_Sequences | ADN | 28S secuencias de ARN ribosómico (LSU) del tipo de hongos y material de referencia |
| Betacoronavirus | ADN | Betacoronavirus |
| GCF_000001405.38_TOP_LEVEL | ADN | HOMO SAPIENS GRCH38.P12 [GCF_000001405.38] Cromosomas más andamios no colocados y no ubalizados |
| GCF_000001635.26_TOP_LEVEL | ADN | Mus Musculus GRCM38.P6 [GCF_000001635.26] Cromosomas más andamios no colocados y no ubalizados |
| Its_refseq_fungi | ADN | Región espaciadora transcrita interna (ITS) del tipo de hongos y material de referencia |
| Its_eukaryote_sequences | ADN | Su explosión de eucariota |
| LSU_EUKARYOTE_RRRNA | ADN | Ácido nucleico ribosómico de subunidad grande para eucariotas |
| LSU_PROCARYOTE_RRRNA | ADN | Ácido nucleico ribosómico de subunidad grande para procariotas |
| SSU_EUKARYOTE_RRRNA | ADN | Ácido nucleico ribosómico de subunidad pequeña para eucariotas |
| env_nt | ADN | muestras ambientales |
| Nuevo Testamento | ADN | Recolección de nucleótidos (NT) |
| patente | ADN | Secuencias de nucleótidos derivadas de la división de patentes de GenBank |
| PDBNT | ADN | Base de datos de nucleótidos PDB |
| ref_euk_rep_genomes | ADN | REFSEQ Representante Eucariota Base de datos del genoma |
| ref_prok_rep_Genomes | ADN | Refseq Procariota Genomas representativos (contiene el ensamblaje de Refseq) |
| ref_viroids_rep_Genomes | ADN | Refseq Viroids Representante Genomas |
| ref_viruses_rep_Genomes | ADN | Refseq Virus Genomas representativos |
| refseq_rna | ADN | Secuencias de referencia de transcripción de NCBI |
| refseq_select_rna | ADN | Refseq Seleccionar secuencias de ARN |
| tsa_nt | ADN | Secuencias de ensamblaje de escopeta de transcriptome (TSA) |
| env_nr | Proteína | Proteínas de proyectos metagenómicos de WGS |
| marca | Proteína | Base de datos de referencia para Smartblast |
| nr | Proteína | Todas las traducciones de CDS de GenBank no redundantes+PDB+SwissProt+PIR+PRF excluyendo muestras ambientales de proyectos WGS |
| PDBAA | Proteína | Base de datos de proteínas PDB |
| pataa | Proteína | Secuencias de proteínas derivadas de la división de patentes de GenBank |
| refseq_protein | Proteína | Secuencias de referencia de proteínas NCBI |
| refseq_select_prot | Proteína | Refseq seleccionar proteínas |
| swissprot | Proteína | Secuencias uniprotkb/swissprot no redundantes |
| tsa_nr | Proteína | Secuencias de ensamblaje de escopeta de transcriptome (TSA) |
| CDD | Proteína | Base de datos de dominio conservada (CDD) es una colección de modelos de alineación de secuencias múltiples bien anotados representados como matrices de puntuación específicas de posición |
El NCBI proporciona metadatos para las bases de datos BLAST disponibles en AWS, GCP y el sitio NCBI FTP.
Acceder a las bases de datos en AWS o GCP fuera del proveedor de la nube probablemente dará como resultado cargos de salida a su cuenta. Si no está en el proveedor de la nube, debe usar las bases de datos en el sitio NCBI FTP.
En AWS y GCP, el archivo está en un subdirectorio dependiente de la fecha con las bases de datos. Para encontrar el último subdirectorio válido, lea por primera vez s3://ncbi-blast-databases/latest-dir (en AWS) o gs://blast-db/latest-dir (en GCP). latest-dir es un archivo de texto con un sello de fecha (por ejemplo, 2020-09-29-01-05-01) que especifica el directorio más reciente. El directorio adecuado será el URI de la base AWS o GCP para las bases de datos BLAST (por ejemplo, s3://ncbi-blast-databases/ FOR AWS) más el texto en el latest-dir . Un ejemplo URI, en AWS, sería s3://ncbi-blast-databases/2020-09-29-01-05-01 . El URI GCP sería similar.
A continuación se muestra un extracto de un archivo de metadatos. La mayoría de los campos tienen significados obvios. Los archivos comprenden la base de datos BLAST. El campo bytes-total representa el tamaño total de la base de datos de explosión en bytes y está destinado a especificar cuánto espacio en disco se requiere.
El siguiente ejemplo es de AWS, pero los archivos de metadatos en GCP tienen el mismo formato. Las bases de datos en el sitio FTP se encuentran en tarfiles de tarro GZippip, una por volumen de la base de datos de explosión, por lo que se enumeran en lugar de los archivos individuales.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
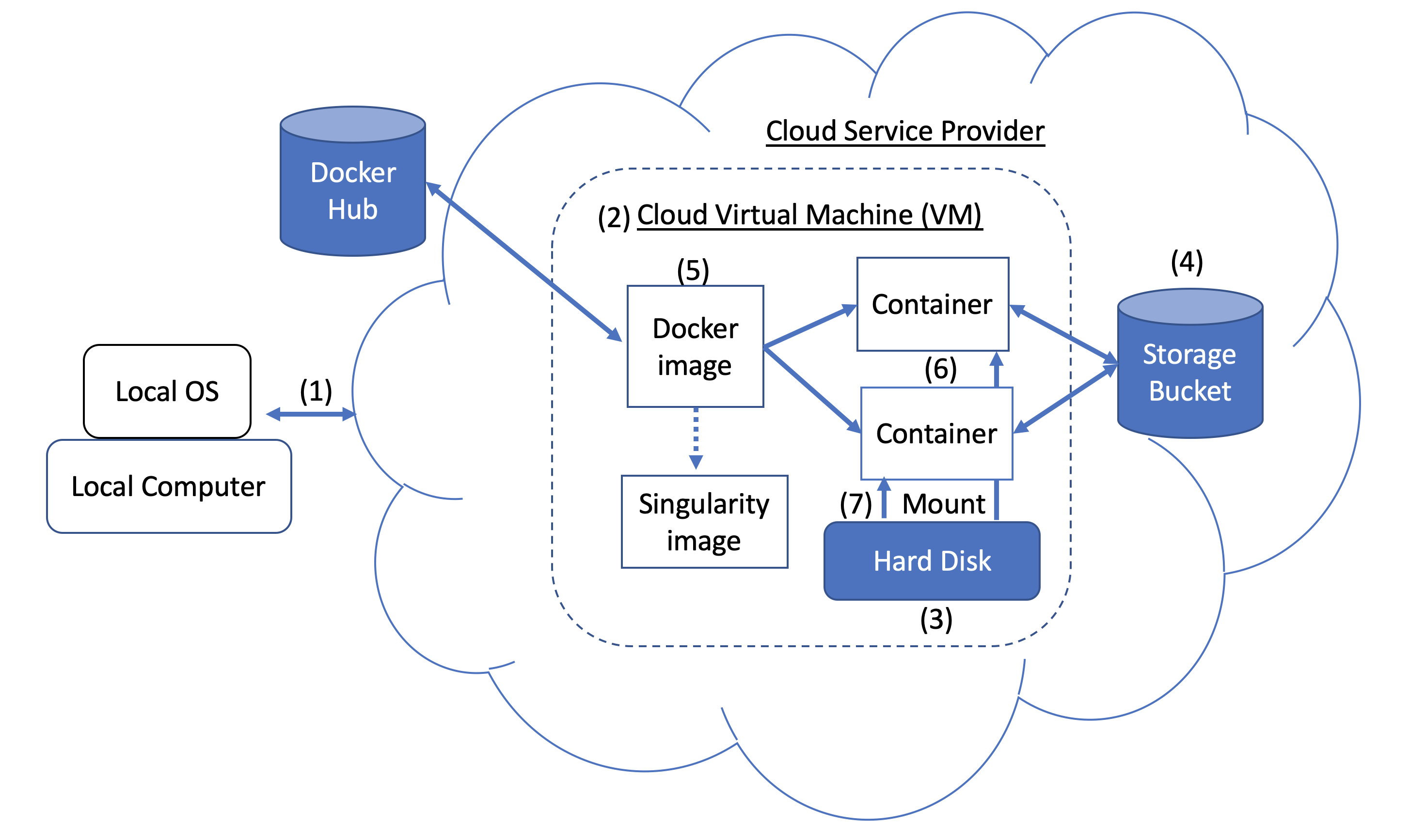 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.