ที่เก็บนี้มีเอกสารประกอบสำหรับแอปพลิเคชันบรรทัดคำสั่ง NCBI BLAST+ ในอิมเมจนักเทียบท่า เราจะสาธิตวิธีการใช้อิมเมจ Docker เพื่อเรียกใช้การวิเคราะห์ BLAST บนแพลตฟอร์ม Google Cloud (GCP) และ Amazon Web Services (AWS) โดยใช้ตัวอย่างพื้นฐานขนาดเล็กและตัวอย่างระดับการผลิตขั้นสูงมากขึ้น ความรู้พื้นฐานบางประการเกี่ยวกับคำสั่ง UNIX/LINUX และ BLAST+ มีประโยชน์ในการทำแบบฝึกหัดนี้ให้สำเร็จ
runrun
ศูนย์ข้อมูลเทคโนโลยีชีวภาพแห่งชาติ (NCBI) เครื่องมือค้นหาการจัดตำแหน่งท้องถิ่นขั้นพื้นฐาน (BLAST) ค้นหาภูมิภาคที่มีความคล้ายคลึงกันในท้องถิ่นระหว่างลำดับ โปรแกรมเปรียบเทียบลำดับนิวคลีโอไทด์หรือโปรตีนกับฐานข้อมูลลำดับและคำนวณนัยสำคัญทางสถิติของการจับคู่ BLAST สามารถใช้เพื่ออนุมานความสัมพันธ์การทำงานและวิวัฒนาการระหว่างลำดับรวมถึงช่วยระบุสมาชิกของตระกูลยีน
เปิดตัวในปี 2009 BLAST+ เป็นแอปพลิเคชันสายคำสั่ง BLAST ที่ได้รับการปรับปรุง สำหรับคำอธิบายทั้งหมดของคุณสมบัติและความสามารถของ BLAST+โปรดดูที่คู่มือผู้ใช้ Line คำสั่ง Blast
คลาวด์คอมพิวติ้งเสนอการประหยัดต้นทุนที่อาจเกิดขึ้นโดยใช้ทรัพยากรการคำนวณตามความต้องการปรับขนาดได้และยืดหยุ่น ในขณะที่คำอธิบายโดยละเอียดเกี่ยวกับเทคโนโลยีคลาวด์และผลประโยชน์ต่าง ๆ อยู่นอกขอบเขตสำหรับที่เก็บนี้ส่วนต่อไปนี้มีข้อมูลที่จำเป็นในการเริ่มต้นการเรียกใช้อิมเมจ BLAST+ Docker บนแพลตฟอร์ม Google Cloud (GCP)
Docker เป็นเครื่องมือในการดำเนินการเสมือนจริงระดับระบบปฏิบัติการโดยใช้คอนเทนเนอร์ซอฟต์แวร์ ในเทคโนโลยีคอนเทนเนอร์ * ภาพเป็นภาพรวมของแอพพลิเคชั่นการห่อหุ้มสภาพแวดล้อมการวิเคราะห์และการพึ่งพา ภาพซึ่งเป็นไฟล์ที่สร้างขึ้นจากรายการคำแนะนำสามารถบันทึกและแบ่งปันได้อย่างง่ายดายเพื่อให้ผู้อื่นสร้างสภาพแวดล้อมการวิเคราะห์ที่แน่นอนในแพลตฟอร์มและระบบปฏิบัติการ คอนเทนเนอร์เป็นอินสแตนซ์รันไทม์ของภาพ ด้วยการใช้ containerization ผู้ใช้สามารถข้ามขั้นตอนที่ซับซ้อนบ่อยครั้งในการรวบรวมการกำหนดค่าและการติดตั้งเครื่องมือที่ใช้ UNIX เช่น BLAST+ นอกเหนือจากความสามารถในการพกพาแล้วคอนเทนเนอร์เป็นวิธีที่มีน้ำหนักเบาในการวิเคราะห์ที่ค้นพบได้มากขึ้นเข้าถึงได้ทำงานร่วมกันได้ง่าย (ยุติธรรม) และในที่สุดก็สามารถทำซ้ำได้
*มีเครื่องมือและมาตรฐานคอนเทนเนอร์มากมายเช่นนักเทียบท่าและความเป็นเอกเทศ เราจะมุ่งเน้นไปที่ Docker เพียงอย่างเดียวซึ่งถือว่าเป็นมาตรฐานตามพฤตินัยของหลาย ๆ คนในสนาม
ส่วนต่อไปนี้รวมถึงคำแนะนำในการสร้าง Google Virtual Machine ติดตั้ง Docker และเรียกใช้คำสั่ง BLAST+ โดยใช้อิมเมจ Docker
ส่วนนี้ให้การวิเคราะห์การระเบิดอย่างรวดเร็วในสภาพแวดล้อม Docker ในอินสแตนซ์ของ Google นี่เป็นภาพรวมสำหรับผู้ที่ต้องการความเข้าใจในหลักการของการแก้ปัญหา หากคุณทำงานกับอินสแตนซ์ของ Amazon โปรดไปที่ส่วนการตั้งค่าบริการเว็บ Amazon ของเอกสารนี้ Google Cloud Shell ซึ่งเป็นสภาพแวดล้อมของเชลล์แบบโต้ตอบจะถูกใช้สำหรับตัวอย่างนี้ซึ่งทำให้สามารถเรียกใช้ตัวอย่างเล็ก ๆ ต่อไปนี้โดยไม่ต้องทำการตั้งค่าเพิ่มเติมเช่นการสร้างบัญชีการเรียกเก็บเงินหรือการคำนวณอินสแตนซ์ คำอธิบายโดยละเอียดเพิ่มเติมของขั้นตอนการวิเคราะห์คำสั่งทางเลือกและหัวข้อขั้นสูงเพิ่มเติมจะครอบคลุมในส่วนต่อมาของเอกสารนี้
ข้อกำหนด: บัญชี Google
การไหลของงาน: 
ข้อมูลอินพุต:
ขั้นแรกในหน้าต่างหรือแท็บเบราว์เซอร์แยกต่างหากลงชื่อเข้าใช้ที่ https://console.cloud.google.com/
คลิกปุ่ม เปิดใช้งานเชลล์เชลล์ ที่มุมขวาบนของคอนโซลแพลตฟอร์ม Google Cloud 
ตอนนี้คุณจะเห็นหน้าต่างเซสชันเชลล์เมฆของคุณ: 
ขั้นตอนต่อไปคือการคัดลอกและวางคำสั่งด้านล่างในเซสชัน Cloud Shell ของคุณ
โปรดทราบ: ใน GitHub คุณสามารถใช้เมาส์เพื่อคัดลอก; อย่างไรก็ตามในเชลล์คำสั่งคุณต้องใช้แป้นพิมพ์ของคุณ ใน Windows หรือ Unix/Linux ให้ใช้ Control+C เพื่อคัดลอกและ Control+V เพื่อวาง บน MacOS ให้ใช้ Command+C เพื่อคัดลอกและ Command+V เพื่อวาง
ในการเลื่อนในเชลล์เมฆให้เปิดใช้งานแถบเลื่อนใน Terminal settings ด้วยไอคอนประแจ 
# Time needed to complete this section: <10 minutes
# Step 1. Retrieve sequences
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Step 2. Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
## Step 3. Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
## Output on screen
## Scroll up to see the entire output
## Type "exit" to leave the Cloud Shell or continue to the next section
ณ จุดนี้คุณควรเห็นผลลัพธ์บนหน้าจอ ด้วยการสืบค้นของคุณ BLAST ระบุลำดับโปรตีน P80049.1 เป็นการจับคู่ที่มีคะแนน 14.2 และค่า e-value 0.96
สำหรับการวิเคราะห์ที่ใหญ่ขึ้นขอแนะนำให้ใช้ธง -out เพื่อบันทึกเอาต์พุตไปยังไฟล์ ตัวอย่างเช่นผนวก -out /blast/results/blastp.out results/blastp.out ไปที่คำสั่งสุดท้ายในขั้นตอนที่ 3 ด้านบนและดูเนื้อหาของไฟล์เอาต์พุตนี้โดยใช้ more $HOME/results/blastp.out
นอกจากนี้คุณยังสามารถสืบค้น P01349.FSA กับ PDB ดังแสดงในบล็อกรหัสต่อไปนี้
## Extend the example to query against the Protein Data Bank
## Time needed to complete this section: <10 minutes
## Confirm query
ls queries/P01349.fsa
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp pdbaa
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
## Output on screen
## Scroll up to see the entire output
## Leave the Cloud Shell
exit
ตอนนี้คุณได้ทำภารกิจง่าย ๆ แล้วและเห็นว่า Blast+ กับ Docker ทำงานได้อย่างไร หากต้องการเรียนรู้เกี่ยวกับ Docker และ BLAST+ ในระดับการผลิตโปรดดำเนินการต่อไปยังส่วนถัดไป
ในส่วนที่ 2-คู่มือทีละขั้นตอนโดยใช้อิมเมจ BLAST+ Docker เราจะใช้ตัวอย่างเล็ก ๆ เดียวกันจากส่วนก่อนหน้าและหารือเกี่ยวกับวิธีการทางเลือก Docker ที่มีประโยชน์เพิ่มเติมและคำสั่ง BLAST+ และตัวเลือกและโครงสร้างคำสั่ง Docker ในส่วนที่ 3 เราจะสาธิตวิธีการเรียกใช้ภาพ Blast+ Docker ในระดับการผลิต
ก่อนอื่นคุณต้องตั้งค่าเครื่องเสมือนของ Google Cloud Platform (GCP) (VM) สำหรับการวิเคราะห์
GCP กำลังเสนอเครดิต $ 300 ซึ่งหมดอายุ 12 เดือนจากการเปิดใช้งานเพื่อสร้างแรงจูงใจให้ผู้ใช้คลาวด์ใหม่ ขั้นตอนต่อไปนี้จะแสดงวิธีการเปิดใช้งานเครดิตนี้ คุณจะถูกขอข้อมูลการเรียกเก็บเงิน แต่ GCP จะไม่เรียกเก็บเงินอัตโนมัติเมื่อการทดลองสิ้นสุดลง คุณต้องเลือกที่จะอัพเกรดด้วยตนเองเป็นบัญชีที่ชำระเงิน
หลังจากลงชื่อเข้าใช้แล้วคลิก เปิดใช้งาน เพื่อเปิดใช้งานเครดิต $ 300 
ป้อนประเทศของคุณตัวอย่างเช่น สหรัฐอเมริกา และทำเครื่องหมายในช่องที่ระบุว่าคุณได้อ่านและยอมรับข้อกำหนดในการให้บริการ
ภายใต้“ ประเภทบัญชี” เลือก“ บุคคล” (อาจถูกเลือกล่วงหน้าในบัญชี Google ของคุณ)
ป้อนชื่อและที่อยู่ของคุณ
ภายใต้“ วิธีที่คุณจ่าย” เลือก“ การชำระเงินอัตโนมัติ” (สิ่งนี้อาจถูกเลือกล่วงหน้าในบัญชี Google ของคุณ) สิ่งนี้บ่งชี้ว่าคุณจะจ่ายค่าใช้จ่ายหลังจากที่คุณใช้บริการไม่ว่าจะมาถึงเกณฑ์การเรียกเก็บเงินหรือทุก ๆ 30 วัน
ภายใต้“ วิธีการชำระเงิน” เลือก“ เพิ่มบัตรเครดิตหรือบัตรเดบิต” และป้อนข้อมูลบัตรเครดิตของคุณ คุณจะไม่ถูกเรียกเก็บเงินโดยอัตโนมัติเมื่อการทดลองสิ้นสุดลง คุณต้องเลือกอัพเกรดเป็นบัญชีที่ชำระเงินก่อนที่จะมีการเรียกเก็บเงินการชำระเงินของคุณ
คลิก“ เริ่มทดลองใช้ฟรี” เพื่อลงทะเบียนให้เสร็จสิ้น เมื่อกระบวนการนี้เสร็จสมบูรณ์คุณควรเห็นหน้าจอต้อนรับ GCP
ณ จุดนี้คุณควรเห็นการประมาณค่าใช้จ่ายสำหรับอินสแตนซ์นี้ทางด้านขวาของหน้าต่าง 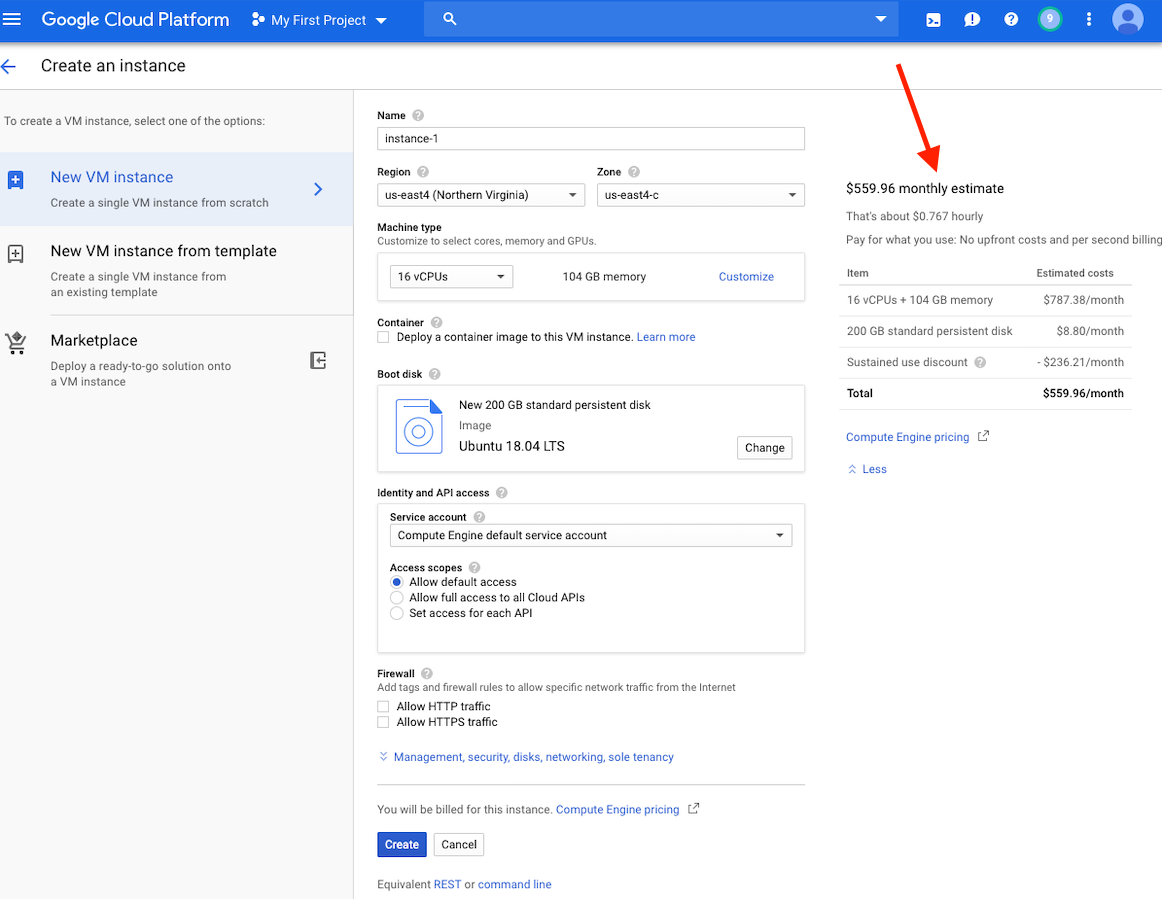
โปรดทราบ: การสร้าง VM ในภูมิภาคเดียวกันกับที่เก็บข้อมูลสามารถให้ประสิทธิภาพที่ดีขึ้น เราขอแนะนำให้สร้าง VM ในภูมิภาค US-East4 หากคุณมีงานที่ต้องใช้เวลาหลายชั่วโมง แต่น้อยกว่า 24 ชั่วโมงคุณอาจใช้ประโยชน์จาก VMs ที่ได้รับการยกเว้น
คำแนะนำโดยละเอียดสำหรับการสร้างบัญชี GCP และเปิดตัว VM สามารถพบได้ที่นี่
เมื่อคุณสร้าง VM แล้วคุณต้องเข้าถึงจากคอมพิวเตอร์ในพื้นที่ของคุณ มีหลายวิธีในการเข้าถึง VM ของคุณขึ้นอยู่กับวิธีที่คุณต้องการใช้ บน GCP วิธีที่ตรงไปตรงมาที่สุดคือ SSH จากเบราว์เซอร์
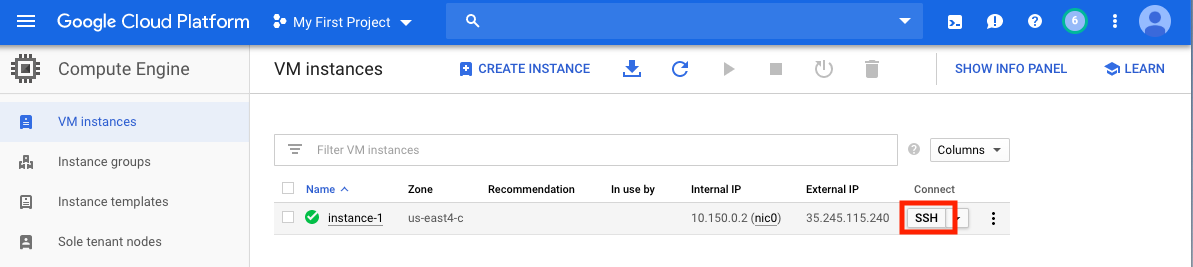
ตอนนี้คุณมีเชลล์คำสั่งที่ทำงานอยู่และคุณพร้อมที่จะดำเนินการต่อ
อย่าลืมหยุดหรือลบ VM เพื่อป้องกันค่าใช้จ่ายเพิ่มเติม
ในส่วนนี้เราจะครอบคลุมการติดตั้ง Docker หารือเกี่ยวกับตัวเลือกคำสั่ง docker run ต่างๆและตรวจสอบโครงสร้างของคำสั่ง Docker เราจะใช้ตัวอย่างเล็ก ๆ เดียวกันจากส่วนที่ 1 และสำรวจวิธีการอื่นในการเรียกใช้อิมเมจ BLAST+ Docker อย่างไรก็ตามเรากำลังใช้อินสแตนซ์ VM จริงซึ่งให้ประสิทธิภาพและฟังก์ชั่นที่ดีกว่า Google Cloud Shell
ข้อมูลอินพุต
ในระบบการผลิตจะต้องติดตั้ง Docker เป็นแอปพลิเคชัน
## Run these commands to install Docker and add non-root users to run Docker
sudo snap install docker
sudo apt update
sudo apt install -y docker.io
sudo usermod -aG docker $USER
exit
# exit and SSH back in for changes to take effect
เพื่อยืนยันการติดตั้ง Docker ที่ถูกต้องให้เรียกใช้คำสั่ง docker run hello-world หากติดตั้งอย่างถูกต้องคุณควรเห็น "สวัสดีจาก Docker! ... " (https://docs.docker.com/samples/library/hello-world/)
ส่วนนี้เป็นทางเลือก
ด้านล่างเป็นรายการตัวเลือกบรรทัดคำสั่ง docker run ที่ใช้ในบทช่วยสอนนี้
| ชื่อ, มือสั้น (ถ้ามี) | คำอธิบาย |
|---|---|
--rm | ลบคอนเทนเนอร์โดยอัตโนมัติเมื่อออก |
--volume , -v | ผูกติดตั้งระดับเสียง |
--workdir , -w | ไดเรกทอรีการทำงานภายในคอนเทนเนอร์ |
ส่วนนี้เป็นทางเลือก
สำหรับบทช่วยสอนนี้จะมีประโยชน์ในการทำความเข้าใจโครงสร้างของคำสั่ง Docker คำสั่งต่อไปนี้ประกอบด้วยสามส่วน
docker run --rm ncbi/blast
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
ส่วนแรกของคำสั่ง docker run --rm ncbi/blast เป็นคำสั่งให้เรียกใช้อิมเมจ Docker ncbi/blast และลบคอนเทนเนอร์เมื่อการรันเสร็จสมบูรณ์
ส่วนที่สองของคำสั่งทำให้ข้อมูลลำดับการสืบค้นสามารถเข้าถึงได้ในคอนเทนเนอร์ Docker Bind Mounts ใช้ -v เพื่อติดตั้งไดเรกทอรีท้องถิ่นไปยังไดเรกทอรีภายในคอนเทนเนอร์และให้สิทธิ์การเข้าถึง RW (อ่านและเขียน) หรือ RO (อ่านเท่านั้น) ตัวอย่างเช่นสมมติว่าลำดับหัวเรื่องของคุณจะถูกเก็บไว้ในไดเรกทอรี $ home/fasta บนโฮสต์ท้องถิ่นคุณสามารถใช้พารามิเตอร์ต่อไปนี้เพื่อให้ไดเรกทอรีนั้นสามารถเข้าถึงได้ภายในคอนเทนเนอร์ใน/blast/fasta เป็นไดเรกทอรีอ่านอย่างเดียว -v $HOME/fasta:/blast/fasta:ro ธง -w /blast/blastdb_custom ตั้งค่าไดเรกทอรีการทำงานภายในคอนเทนเนอร์
ส่วนที่สามของคำสั่งคือคำสั่ง BLAST+ ในกรณีนี้มันกำลังดำเนินการ makeBlastDB เพื่อสร้างไฟล์ฐานข้อมูล BLAST
คุณสามารถเริ่มเซสชันการทุบตีแบบโต้ตอบสำหรับภาพนี้โดยใช้ docker run -it ncbi/blast /bin/bash สำหรับอิมเมจ Blast+ Docker, Executables อยู่ในโฟลเดอร์ /blast /bin และ /root /edirect และเพิ่มลงในเส้นทาง $ ตัวแปร
สำหรับเอกสารเพิ่มเติมเกี่ยวกับคำสั่ง docker run โปรดดูเอกสารประกอบ
ส่วนนี้เป็นทางเลือก
| คำสั่ง Docker | คำอธิบาย |
|---|---|
docker ps -a | แสดงรายการคอนเทนเนอร์ |
docker rm $(docker ps -q -f status=exited) | ลบคอนเทนเนอร์ที่ออกทั้งหมดหากคุณมีคอนเทนเนอร์ที่ออกอย่างน้อย 1 รายการ |
docker rm <CONTAINER_ID> | ลบคอนเทนเนอร์ |
docker images | แสดงรายการรูปภาพ |
docker rmi <REPOSITORY (IMAGE_NAME)> | ลบภาพ |
ส่วนนี้เป็นทางเลือก
ด้วยภาพนักเทียบท่านี้คุณสามารถเรียกใช้ BLAST+ ในภาชนะที่แยกได้ซึ่งอำนวยความสะดวกในการทำซ้ำของผลลัพธ์การระเบิด ในฐานะผู้ใช้ของอิมเมจนักเทียบท่านี้คุณคาดว่าจะให้ฐานข้อมูล BLAST และลำดับการสืบค้นเพื่อเรียกใช้ BLAST รวมถึงตำแหน่งนอกคอนเทนเนอร์เพื่อบันทึกผลลัพธ์ ต่อไปนี้เป็นรายการของไดเรกทอรีที่ใช้โดย BLAST+ คุณจะสร้างพวกเขาในขั้นตอนที่ 2
| ไดเรกทอรี | วัตถุประสงค์ | หมายเหตุ |
|---|---|---|
$HOME/blastdb | จัดเก็บฐานข้อมูล Blast NCBI | หากตั้งค่าเป็นเส้นทาง เดียวที่แน่นอน ตัวแปรสภาพแวดล้อม $BLASTDB สามารถใช้แทนได้ (ดูการกำหนดค่า BLAST ผ่านตัวแปรสภาพแวดล้อม) |
$HOME/queries | จัดเก็บลำดับการสืบค้นที่ผู้ใช้ให้ | |
$HOME/fasta | จัดเก็บลำดับ Fasta ที่ผู้ใช้ให้ไว้เพื่อสร้างฐานข้อมูล Blast (S) | |
$HOME/results | เก็บผลการระเบิด | ติดตั้งด้วยสิทธิ์ rw |
$HOME/blastdb_custom | จัดเก็บฐานข้อมูล BLAST ที่ผู้ใช้ให้ |
ส่วนนี้เป็นทางเลือก
คำสั่งต่อไปนี้แสดงเวอร์ชัน BLAST ล่าสุด
docker run --rm ncbi/blast blastn -version
การต่อท้ายแท็กเข้ากับชื่อภาพ ( ncbi/blast ) ช่วยให้คุณสามารถใช้ BLAST+ เวอร์ชันอื่น (ดู“ แท็กที่รองรับและบันทึกย่อที่เกี่ยวข้อง” สำหรับเวอร์ชันที่รองรับ)
BLAST+ รุ่นที่แตกต่างกันมีอยู่ในภาพ Docker ที่แตกต่างกัน คำสั่งต่อไปนี้จะเริ่มดาวน์โหลดภาพ Docker BLAST+ เวอร์ชัน 2.9.0
docker run --rm ncbi/blast:2.9.0 blastn -version
## Display a list of images
docker images
ตัวอย่างเช่นในการใช้อิมเมจ BLAST+ เวอร์ชัน 2.9.0 Docker แทนเวอร์ชันล่าสุดให้แทนที่ส่วนแรกของคำสั่ง
docker run --rm ncbi/blast พร้อม docker run --rm ncbi/blast:2.9.0
ส่วนนี้เป็นทางเลือก
ในตัวอย่างนี้เราจะเริ่มต้นด้วยการดึงลำดับและลำดับฐานข้อมูลจากนั้นสร้างฐานข้อมูล BLAST ที่กำหนดเอง
# Start in a directory where you want to perform the analysis
## Create directories for analysis
cd ; mkdir blastdb queries fasta results blastdb_custom
## Retrieve query sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
ในการตรวจสอบฐานข้อมูล BLAST ที่สร้างขึ้นใหม่ด้านบนคุณสามารถเรียกใช้คำสั่งต่อไปนี้เพื่อแสดงการเข้าถึงความยาวลำดับและชื่อสามัญของลำดับในฐานข้อมูล
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
อีกทางเลือกหนึ่งคุณยังสามารถดาวน์โหลดฐานข้อมูล Blast Preformatted จาก NCBI หรือ Bucket Storage NCBI Google
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
สำหรับคำอธิบายโดยละเอียดของ update_blastdb.pl โปรดดูเอกสารประกอบ โดยค่าเริ่มต้น update_blastdb.pl จะดาวน์โหลดจากคลาวด์หากคุณเชื่อมต่อหรือจาก NCBI หากคุณไม่ได้ใช้ผู้ให้บริการคลาวด์ที่รองรับ
ส่วนนี้เป็นทางเลือก
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
ส่วนนี้เป็นทางเลือก
คำสั่งด้านล่างติดตั้งเส้นทาง $HOME/blastdb บนเครื่องท้องถิ่น AS /blast/blastdb บนคอนเทนเนอร์และ blastdbcmd แสดงฐานข้อมูล BLAST ที่มีอยู่ในตำแหน่งนี้
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
คุณควรเห็น /blast/blastdb/pdbaa Protein
## For the custom BLAST database used in this example -
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -list /blast/blastdb_custom -remove_redundant_dbs
คุณควรเห็นเอาต์พุต /blast/blastdb_custom/nurse-shark-proteins Protein
เมื่อเรียกใช้ BLAST ในคอนเทนเนอร์ Docker ให้สังเกตการเมาท์ที่ระบุไว้ในคำสั่ง docker run เพื่อให้สามารถเข้าถึงอินพุตและเอาต์พุตได้ ในตัวอย่างด้านล่างการเมานต์สองตัวแรกให้การเข้าถึงฐานข้อมูล BLAST ตัวยึดที่สามให้การเข้าถึงลำดับการสืบค้นและตัวยึดที่สี่ให้ไดเรกทอรีเพื่อบันทึกผลลัพธ์ (หมายเหตุ :ro และ :rw ซึ่งติดตั้งไดเรกทอรีเป็นอ่านอย่างเดียวและอ่านตามลำดับ)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
ณ จุดนี้คุณควรเห็นไฟล์เอาต์พุต $HOME/results/blastp.out ด้วยการสืบค้นของคุณ BLAST ระบุลำดับโปรตีน P80049.1 เป็นการจับคู่ที่มีคะแนน 14.2 และค่า e-value 0.96 หากต้องการดูเนื้อหาของไฟล์เอาต์พุตนี้ให้ใช้คำสั่ง more $HOME/results/blastp.out
อย่าลืมหยุดหรือลบ VM เพื่อป้องกันค่าใช้จ่ายเพิ่มเติม คุณสามารถทำได้ที่คอนโซล GCP ดังที่แสดงด้านล่าง 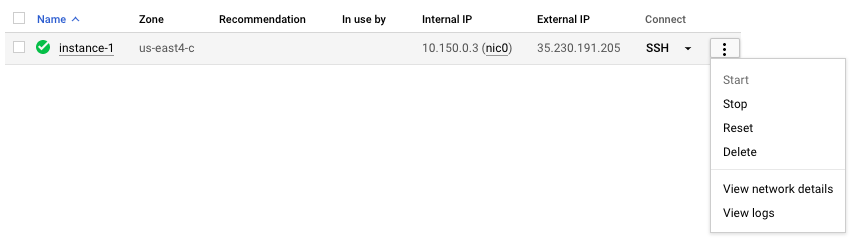
หนึ่งในสัญญาของคลาวด์คอมพิวติ้งคือความยืดหยุ่น ในส่วนนี้เราจะสาธิตวิธีการใช้ภาพ Blast+ Docker ในระดับการผลิตบนแพลตฟอร์ม Google Cloud เราจะทำการวิเคราะห์การระเบิดคล้ายกับวิธีการที่อธิบายไว้ในสิ่งพิมพ์นี้เพื่อเปรียบเทียบ de novo จัดเรียง contigs จากการเรียงลำดับของแบคทีเรีย 16S-23S กับฐานข้อมูล Nucleotide Collection (NT)
ในการทดสอบความสามารถในการปรับขนาดเราจะใช้อินพุตที่มีขนาดต่างกันเพื่อประเมินระยะเวลาในการดาวน์โหลดฐานข้อมูลคอลเลกชันนิวคลีโอไทด์และเรียกใช้การค้นหา BLAST โดยใช้ภาพ BLAST+ Docker เวอร์ชันล่าสุด ผลลัพธ์ที่คาดหวังจะสรุปไว้ในตารางต่อไปนี้
ไฟล์อินพุต: 28 ตัวอย่าง (ไฟล์มัลติ-ฟาสต้า) ที่มี de novo จัดเรียง contigs จากสิ่งพิมพ์
(คำแนะนำในการดาวน์โหลดและสร้างไฟล์อินพุตอธิบายไว้ในบล็อกรหัสด้านล่าง)
ฐานข้อมูล: ฐานข้อมูลการรวบรวมนิวคลีโอไทด์ BLAST ที่จัดรูปแบบล่วงหน้ารุ่น 5 (NT): 68.7217 GB (ตั้งแต่เดือนพฤษภาคม 2019)
| ชื่อไฟล์อินพุต | เนื้อหาไฟล์ | ขนาดไฟล์ | จำนวนลำดับ | จำนวนนิวคลีโอไทด์ | ขนาดเอาต์พุตที่คาดไว้ | |
|---|---|---|---|---|---|---|
| การวิเคราะห์ 1 | Query1.fa | ตัวอย่าง 1 เท่านั้น | 59 kb | 121 | 51,119 | 3.1 GB |
| การวิเคราะห์ 2 | query5.fa | ตัวอย่าง 1-5 เท่านั้น | 422 kb | 717 | 375,154 | 10.4 GB |
| การวิเคราะห์ 3 | query.fa | ตัวอย่างทั้งหมด 28 ตัวอย่าง | 2.322 MB | 3798 | 2,069,892 | 47.8 GB |
| ประเภท/โซน VM | ซีพียู | หน่วยความจำ (GB) | ค่าใช้จ่ายรายชั่วโมง* | ดาวน์โหลด NT (ขั้นต่ำ) | การวิเคราะห์ 1 (นาที) | การวิเคราะห์ 2 (นาที) | การวิเคราะห์ 3 (นาที) | ค่าใช้จ่ายทั้งหมด ** |
|---|---|---|---|---|---|---|---|---|
| N1-Standard-8 US-East4c | 8 | 30 | $ 0.312 | 9 | 22 | - | - | - |
| N1-Standard-16 US-East4c | 16 | 60 | $ 0.611 | 9 | 14 | 53 | 205 | $ 2.86 |
| N1-HIGHMEM-16 US-East4C | 16 | 104 | $ 0.767 | 9 | 9 | 30 | 143 | $ 2.44 |
| N1-HIGHMEM-16 US-WEST2A | 16 | 104 | $ 0.809 | 11 | 9 | 30 | 147 | $ 2.60 |
| N1-HIGHMEM-16 US-WEST1B | 16 | 104 | $ 0.674 | 11 | 9 | 30 | 147 | $ 2.17 |
| เว็บไซต์ Blast (Blastn) | - | - | - | - | การค้นหาเกินข้อ จำกัด ในปัจจุบันเกี่ยวกับการใช้งาน | การค้นหาเกินข้อ จำกัด ในปัจจุบันเกี่ยวกับการใช้งาน | การค้นหาเกินข้อ จำกัด ในปัจจุบันเกี่ยวกับการใช้งาน | - |
อินสแตนซ์ GCP ทั้งหมดได้รับการกำหนดค่าด้วยดิสก์มาตรฐานถาวร 200 GB
*ค่าใช้จ่ายรายชั่วโมงจัดทำโดย Google Cloud Platform (พฤษภาคม 2019) เมื่อ VM ถูกสร้างขึ้นและอาจมีการเปลี่ยนแปลง
** ประมาณค่าใช้จ่ายทั้งหมดโดยใช้ค่าใช้จ่ายรายชั่วโมงและเวลาทั้งหมดในการดาวน์โหลด NT และ RUN การวิเคราะห์ 1, การวิเคราะห์ 2 และการวิเคราะห์ 3. ประมาณการใช้สำหรับการเปรียบเทียบเท่านั้น ค่าใช้จ่ายของคุณอาจแตกต่างกันไปและเป็นความรับผิดชอบในการตรวจสอบและจัดการ
โปรดดูที่ GCP สำหรับข้อมูลเพิ่มเติมเกี่ยวกับประเภทของเครื่องภูมิภาคและโซนและคำนวณค่าใช้จ่าย
โปรดทราบว่าการเรียกใช้ blastn binary โดยไม่ระบุพารามิเตอร์ -task ของมันจะเรียกใช้อัลกอริทึม megablast
## Install Docker if not already done
## This section assumes using recommended hardware requirements below
## 16 CPUs, 104 GB memory and 200 GB persistent hard disk
## Modify the number of CPUs (-num_threads) in Step 3 if another type of VM is used.
## Step 1. Prepare for analysis
## Create directories
cd ; mkdir -p blastdb queries fasta results blastdb_custom
## Import and process input sequences
sudo apt install unzip
wget https://ndownloader.figshare.com/articles/6865397?private_link=729b346eda670e9daba4 -O fa.zip
unzip fa.zip -d fa
### Create three input query files
### All 28 samples
cat fa/*.fa > query.fa
### Sample 1
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa' > query1.fa
### Sample 1 to Sample 5
cat fa/'Sample_1 (paired) trimmed (paired) assembly.fa'
fa/'Sample_2 (paired) trimmed (paired) assembly.fa'
fa/'Sample_3 (paired) trimmed (paired) assembly.fa'
fa/'Sample_4 (paired) trimmed (paired) assembly.fa'
fa/'Sample_5 (paired) trimmed (paired) assembly.fa' > query5.fa
### Copy query sequences to $HOME/queries folder
cp query* $HOME/queries/.
## Step 2. Display BLAST databases on the GCP
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source gcp
## Download nt (nucleotide collection version 5) database
## This step takes approximately 10 min. The following command runs in the background.
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl --source gcp nt &
## At this point, confirm query/database have been properly provisioned before proceeding
## Check the size of the directory containing the BLAST database
## nt should be around 68 GB (this was in May 2019)
du -sk $HOME/blastdb
## Check for queries, there should be three files - query.fa, query1.fa and query5.fa
ls -al $HOME/queries
## From this point forward, it may be easier if you run these steps in a script.
## Simply copy and paste all the commands below into a file named script.sh
## Then run the script in the background `nohup bash script.sh > script.out &`
## Step 3. Run BLAST
## Run BLAST using query1.fa (Sample 1)
## This command will take approximately 9 minutes to complete.
## Expected output size: 3.1 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query1.fa -db nt -num_threads 16
-out /blast/results/blastn.query1.denovo16s.out
## Run BLAST using query5.fa (Samples 1-5)
## This command will take approximately 30 minutes to complete.
## Expected output size: 10.4 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query5.fa -db nt -num_threads 16
-out /blast/results/blastn.query5.denovo16s.out
## Run BLAST using query.fa (All 28 samples)
## This command will take approximately 147 minutes to complete.
## Expected output size: 47.8 GB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastn -query /blast/queries/query.fa -db nt -num_threads 16
-out /blast/results/blastn.query.denovo16s.out
## Stdout and stderr will be in script.out
## BLAST output will be in $HOME/results
คุณได้จบการสอนทั้งหมดแล้ว ณ จุดนี้หากคุณไม่ต้องการข้อมูลที่ดาวน์โหลดมาเพื่อการวิเคราะห์เพิ่มเติมโปรดลบ VM เพื่อป้องกันค่าใช้จ่ายเพิ่มเติมที่เกิดขึ้น
หากต้องการลบอินสแตนซ์ให้ทำตามคำแนะนำในส่วนหยุดอินสแตนซ์ GCP
สำหรับข้อมูลเพิ่มเติมโปรดดูเอกสารของ Google Cloud Platform เกี่ยวกับวงจรชีวิตอินสแตนซ์
ในการเรียกใช้ตัวอย่างเหล่านี้คุณจะต้องมีบัญชี Amazon Web Services (AWS) หากคุณยังไม่มีอยู่แล้วคุณสามารถสร้างบัญชีที่ให้ความสามารถในการสำรวจและลองใช้บริการ AWS ฟรีตามขีด จำกัด ที่ระบุสำหรับแต่ละบริการ ในการเริ่มต้นเยี่ยมชมเว็บไซต์ระดับฟรีซึ่งจะต้องใช้บัตรเครดิตที่ถูกต้อง แต่จะไม่ถูกเรียกเก็บเงินหากคุณคำนวณภายในระดับฟรี เมื่อเลือกผลิตภัณฑ์ระดับฟรีให้แน่ใจว่าอยู่ใน การคำนวณ หมวดหมู่ผลิตภัณฑ์
คำแนะนำเหล่านี้สร้าง EC2 VM ตามอิมเมจของ Amazon Machine (AMI) ที่มีนักเทียบท่าและการพึ่งพา
ด้วย VM ที่สร้างขึ้นคุณจะเข้าถึงได้จากคอมพิวเตอร์ในพื้นที่ของคุณโดยใช้ SSH ไฟล์คู่ / .pem ของคุณทำหน้าที่เป็นข้อมูลประจำตัวของคุณ
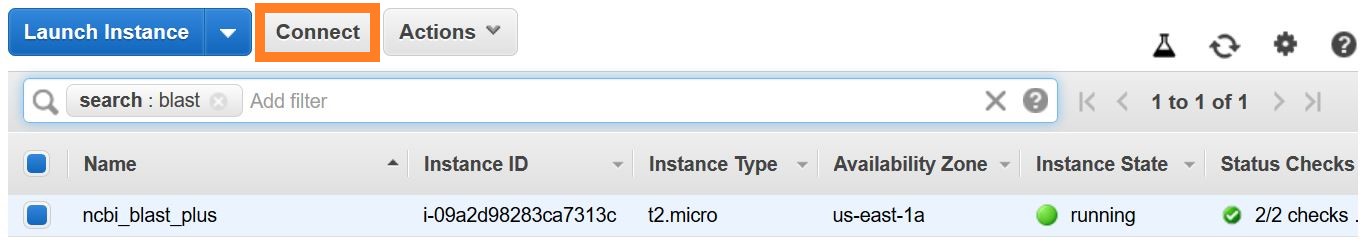
มีหลายวิธีในการสร้างการเชื่อมต่อ SSH จากรายการอินสแตนซ์ EC2 ในคอนโซล AWS เลือก เชื่อมต่อ จากนั้นทำตามคำแนะนำสำหรับวิธีการเชื่อมต่อ ไคลเอนต์ SSH แบบสแตนด์อโลน
คำแนะนำโดยละเอียดสำหรับการเชื่อมต่อกับ Linux VM สามารถพบได้ที่นี่
ระบุ ผู้ใช้ EC2 เป็นชื่อผู้ใช้แทนที่จะเป็นรูทในบรรทัดคำสั่ง SSH ของคุณหรือเมื่อได้รับแจ้งให้เข้าสู่ระบบระบุ ผู้ใช้ EC2 เป็นชื่อผู้ใช้
ในตัวอย่างนี้เราจะเริ่มต้นด้วยการดึงลำดับและลำดับฐานข้อมูลจากนั้นสร้างฐานข้อมูล BLAST ที่กำหนดเอง
## Retrieve sequences
## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
## Retrieve database sequences
docker run --rm ncbi/blast efetch -db protein -format fasta
-id Q90523,P80049,P83981,P83982,P83983,P83977,P83984,P83985,P27950
> fasta/nurse-shark-proteins.fsa
## Make BLAST database
docker run --rm
-v $HOME/blastdb_custom:/blast/blastdb_custom:rw
-v $HOME/fasta:/blast/fasta:ro
-w /blast/blastdb_custom
ncbi/blast
makeblastdb -in /blast/fasta/nurse-shark-proteins.fsa -dbtype prot
-parse_seqids -out nurse-shark-proteins -title "Nurse shark proteins"
-taxid 7801 -blastdb_version 5
ในการตรวจสอบฐานข้อมูล BLAST ที่สร้างขึ้นใหม่ด้านบนคุณสามารถเรียกใช้คำสั่งต่อไปนี้เพื่อแสดงการเข้าถึงความยาวลำดับและชื่อสามัญของลำดับในฐานข้อมูล
## Verify BLAST DB
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
ncbi/blast
blastdbcmd -entry all -db nurse-shark-proteins -outfmt "%a %l %T"
เมื่อเรียกใช้ BLAST ในคอนเทนเนอร์ Docker ให้สังเกตตัวเลือก Mounts ( -v ) ที่ระบุไว้ในคำสั่ง docker run เพื่อให้สามารถเข้าถึงอินพุตและเอาต์พุตได้ ในตัวอย่างด้านล่างการเมานต์สองตัวแรกให้การเข้าถึงฐานข้อมูล BLAST ตัวยึดที่สามให้การเข้าถึงลำดับการสืบค้นและตัวยึดที่สี่ให้ไดเรกทอรีเพื่อบันทึกผลลัพธ์ (หมายเหตุ :ro และ :rw ซึ่งติดตั้งไดเรกทอรีเป็นอ่านอย่างเดียวและอ่านตามลำดับ)
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
ณ จุดนี้คุณควรเห็นไฟล์เอาต์พุต $HOME/results/blastp.out ด้วยการสืบค้นของคุณ BLAST ระบุลำดับโปรตีน P80049.1 เป็นการจับคู่ที่มีคะแนน 14.2 และค่า e-value 0.96 หากต้องการดูเนื้อหาของไฟล์เอาต์พุตนี้ให้ใช้คำสั่ง more $HOME/results/blastp.out
docker run --rm ncbi/blast update_blastdb.pl --showall pretty --source aws
เอาต์พุตที่คาดหวังคือรายการของ Blast DBS รวมถึงชื่อคำอธิบายขนาดและวันที่อัปเดตล่าสุด
สำหรับคำอธิบายโดยละเอียดของ update_blastdb.pl โปรดดูเอกสารประกอบ โดยค่าเริ่มต้น update_blastdb.pl จะดาวน์โหลดจากคลาวด์หากคุณเชื่อมต่อหรือจาก NCBI หากคุณไม่ได้ใช้ผู้ให้บริการคลาวด์ที่รองรับ
docker run --rm ncbi/blast update_blastdb.pl --showall --source ncbi
เอาต์พุตที่คาดหวังคือรายการชื่อของ Blast DBS
อย่าลืมหยุดหรือยุติ VM เพื่อป้องกันค่าใช้จ่ายเพิ่มเติม คุณสามารถทำได้จากรายการอินสแตนซ์ EC2 ในคอนโซล AWS ดังที่แสดงด้านล่าง
ตัวอย่างนี้ต้องใช้โฮสต์มัลติคอร์ ดังนั้นค่าใช้จ่ายในการคำนวณ EC2 จะได้รับการรับรู้โดยการดำเนินการตัวอย่างนี้ อัตราปัจจุบันสำหรับประเภทอินสแตนซ์ที่ใช้ - T2.large - คือ $ 0.093/ชม.
คำแนะนำเหล่านี้สร้าง EC2 VM ตามอิมเมจของ Amazon Machine (AMI) ที่มีนักเทียบท่าและการพึ่งพา
ด้วย VM ที่สร้างขึ้นคุณจะเข้าถึงได้จากคอมพิวเตอร์ในพื้นที่ของคุณโดยใช้ SSH ไฟล์คู่ / .pem ของคุณทำหน้าที่เป็นข้อมูลประจำตัวของคุณ
มีหลายวิธีในการสร้างการเชื่อมต่อ SSH จากรายการอินสแตนซ์ EC2 ในคอนโซล AWS เลือก เชื่อมต่อ จากนั้นทำตามคำแนะนำสำหรับวิธีการเชื่อมต่อ ไคลเอนต์ SSH แบบสแตนด์อโลน
คำแนะนำโดยละเอียดสำหรับการเชื่อมต่อกับ Linux VM สามารถพบได้ที่นี่
ระบุ ผู้ใช้ EC2 เป็นชื่อผู้ใช้แทนที่จะเป็นรูทในบรรทัดคำสั่ง SSH ของคุณหรือเมื่อได้รับแจ้งให้เข้าสู่ระบบระบุ ผู้ใช้ EC2 เป็นชื่อผู้ใช้

## Create directories for analysis
cd $HOME; sudo mkdir bin blastdb queries fasta results blastdb_custom; sudo chown ec2-user:ec2-user *
## Retrieve query sequence
docker run --rm ncbi/blast efetch -db protein -format fasta
-id P01349 > queries/P01349.fsa
คำสั่งด้านล่างเมาท์ (ใช้ตัวเลือก -v ) เส้นทาง $HOME/blastdb บนเครื่องท้องถิ่น AS /blast/blastdb บนคอนเทนเนอร์และ blastdbcmd แสดงฐานข้อมูล BLAST ที่มีอยู่ในตำแหน่งนี้
## Download Protein Data Bank amino acid database (pdbaa)
docker run --rm
-v $HOME/blastdb:/blast/blastdb:rw
-w /blast/blastdb
ncbi/blast
update_blastdb.pl pdbaa
## Display database(s) in $HOME/blastdb
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
ncbi/blast
blastdbcmd -list /blast/blastdb -remove_redundant_dbs
คุณควรเห็น /blast/blastdb/pdbaa Protein
## Run BLAST+
docker run --rm
-v $HOME/blastdb:/blast/blastdb:ro
-v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
blastp -query /blast/queries/P01349.fsa -db pdbaa
-out /blast/results/blastp_pdbaa.out
ณ จุดนี้คุณควรเห็นไฟล์เอาต์พุต $HOME/results/blastp_pdbaa.out หากต้องการดูเนื้อหาของไฟล์เอาต์พุตนี้ให้ใช้คำสั่ง more $HOME/results/blastp_pdbaa.out
วิธีหนึ่งในการถ่ายโอนไฟล์ระหว่างคอมพิวเตอร์ในพื้นที่ของคุณและอินสแตนซ์ Linux คือการใช้ Secure Copy Protocol (SCP)
ส่วน การถ่ายโอนไฟล์ไปยังอินสแตนซ์ Linux จาก Linux โดยใช้ SCP ของคู่มือผู้ใช้ Amazon EC2 สำหรับอินสแตนซ์ Linux ให้คำแนะนำโดยละเอียดสำหรับกระบวนการนี้
NCBI โฮสต์ฐานข้อมูลเดียวกันใน AWS, GCP และเว็บไซต์ NCBI FTP ตารางด้านล่างมีรายการฐานข้อมูลปัจจุบัน ณ เดือนพฤศจิกายนปี 2022
นอกจากนี้ยังเป็นไปได้ที่จะได้รับรายการปัจจุบันด้วยคำสั่ง:
Docker Run -rm ncbi/blast update_blastdb.pl -Showall Pretty
หรือ
update_blastdb.pl -Showall สวย # หลังจากดาวน์โหลดแพ็คเกจ Blast+
ดังที่แสดงไว้ด้านบน update_blastdb.pl ยังสามารถใช้ในการดาวน์โหลดฐานข้อมูลเหล่านี้ มันจะเลือกทรัพยากรที่เหมาะสมโดยอัตโนมัติ (เช่น GCP หากคุณอยู่ในผู้ให้บริการรายนั้น)
ฐานข้อมูลเหล่านี้ยังสามารถค้นหาด้วยความยืดหยุ่นบน GCP และ AWS
การเข้าถึงฐานข้อมูลบน AWS หรือ GCP นอกผู้ให้บริการคลาวด์จะส่งผลให้ค่าใช้จ่ายออกไปยังบัญชีของคุณ หากคุณไม่ได้อยู่ในผู้ให้บริการคลาวด์คุณควรใช้ฐานข้อมูลที่ไซต์ NCBI FTP
| ชื่อ | พิมพ์ | ชื่อ |
|---|---|---|
| 16S_RIBOSOMAL_RNA | ดีเอ็นเอ | 16S ribosomal RNA (แบคทีเรียและสายพันธุ์ Archaea) |
| 18S_FUNGAL_SECTION | ดีเอ็นเอ | ลำดับ 18S ribosomal RNA (SSU) จากประเภทเชื้อราและวัสดุอ้างอิง |
| 28S_FUNGAL_SECTION | ดีเอ็นเอ | ลำดับ 28S ribosomal RNA (LSU) จากประเภทเชื้อราและวัสดุอ้างอิง |
| Betacoronavirus | ดีเอ็นเอ | Betacoronavirus |
| GCF_000001405.38_TOP_LEVEL | ดีเอ็นเอ | homo sapiens grch38.p12 [GCF_000001405.38] โครโมโซมรวมถึงโครงนั่งร้านที่ไม่ได้รับการออกแบบ |
| GCF_000001635.26_TOP_LEVEL | ดีเอ็นเอ | MUS Musculus GRCM38.P6 [GCF_000001635.26] โครโมโซมรวมถึงโครงนั่งร้านที่ไม่ได้รับการออกแบบ |
| its_refseq_fungi | ดีเอ็นเอ | พื้นที่เว้นวรรคที่ถอดความภายใน (ITS) จากประเภทเชื้อราและวัสดุอ้างอิง |
| ITS_EUKARYOTE_SECTION | ดีเอ็นเอ | ยูคาริโอตระเบิด |
| lsu_eukaryote_rrna | ดีเอ็นเอ | กรดนิวคลีอิก ribosomal subunit ขนาดใหญ่สำหรับยูคาริโอต |
| lsu_prokaryote_rrna | ดีเอ็นเอ | กรดนิวคลีอิก ribosomal subunit ขนาดใหญ่สำหรับ prokaryotes |
| SSU_EUKARYOTE_RRNA | ดีเอ็นเอ | กรดนิวคลีอิก ribosomal subunit ขนาดเล็กสำหรับยูคาริโอต |
| env_nt | ดีเอ็นเอ | ตัวอย่างสิ่งแวดล้อม |
| NT | ดีเอ็นเอ | การรวบรวมนิวคลีโอไทด์ (NT) |
| patnt | ดีเอ็นเอ | ลำดับนิวคลีโอไทด์ที่ได้มาจากการแบ่งสิทธิบัตรของ GenBank |
| pdbnt | ดีเอ็นเอ | ฐานข้อมูล PDB นิวคลีโอไทด์ |
| ref_euk_rep_genomes | ดีเอ็นเอ | ฐานข้อมูลจีโนมตัวแทนยูคาริโอต REFSEQ |
| ref_prok_rep_genomes | ดีเอ็นเอ | Refseq Prokaryote ตัวแทนจีโนม (มีชุดประกอบ REFSEQ) |
| ref_viroids_rep_genomes | ดีเอ็นเอ | จีโนมตัวแทนของ REFSEQ Viroids |
| ref_viruses_rep_genomes | ดีเอ็นเอ | จีโนมตัวแทนไวรัส REFSEQ |
| REFSEQ_RNA | ดีเอ็นเอ | ลำดับการอ้างอิงการถอดเสียง NCBI |
| REFSEQ_SELECT_RNA | ดีเอ็นเอ | REFSEQ เลือกลำดับ RNA |
| tsa_nt | ดีเอ็นเอ | ลำดับการประกอบปืนลูกซอง Transcriptome (TSA) |
| env_nr | โปรตีน | โปรตีนจากโครงการ Metagenomic WGS |
| สถานที่สำคัญ | โปรตีน | ฐานข้อมูลสถานที่สำคัญสำหรับ SmartBlast |
| NR | โปรตีน | การแปล GenBank CDS ที่ไม่ซ้ำซ้อนทั้งหมด+PDB+SWISSPROT+PIR+PRF ไม่รวมตัวอย่างสิ่งแวดล้อมจากโครงการ WGS |
| PDBAA | โปรตีน | ฐานข้อมูลโปรตีน PDB |
| Pataa | โปรตีน | ลำดับโปรตีนที่ได้มาจากการแบ่งสิทธิบัตรของ GenBank |
| REFSEQ_PROTEIN | โปรตีน | ลำดับการอ้างอิงโปรตีน NCBI |
| REFSEQ_SELECT_PROT | โปรตีน | REFSEQ เลือกโปรตีน |
| ชาวสวิสสปอต | โปรตีน | ลำดับ UNIPROTKB/SWISSPROT ที่ไม่ซ้ำซ้อน |
| tsa_nr | โปรตีน | ลำดับการประกอบปืนลูกซอง Transcriptome (TSA) |
| CDD | โปรตีน | อนุรักษ์ฐานข้อมูลโดเมน (CDD) เป็นคอลเลกชันของโมเดลการจัดตำแหน่งหลายลำดับที่มีคำอธิบายประกอบการจัดเรียงหลายลำดับเป็นเมทริกซ์คะแนนเฉพาะตำแหน่ง |
NCBI ให้ข้อมูลเมตาสำหรับฐานข้อมูล BLAST ที่มีอยู่ที่ AWS, GCP และเว็บไซต์ NCBI FTP
การเข้าถึงฐานข้อมูลบน AWS หรือ GCP นอกผู้ให้บริการคลาวด์จะส่งผลให้ค่าใช้จ่ายออกไปยังบัญชีของคุณ หากคุณไม่ได้อยู่ในผู้ให้บริการคลาวด์คุณควรใช้ฐานข้อมูลที่ไซต์ NCBI FTP
บน AWS และ GCP ไฟล์จะอยู่ในไดเรกทอรีย่อยที่ขึ้นกับวันที่ด้วยฐานข้อมูล หากต้องการค้นหาไดเรกทอรีย่อยที่ถูกต้องล่าสุดให้อ่าน s3://ncbi-blast-databases/latest-dir (บน AWS) หรือ gs://blast-db/latest-dir (บน GCP) latest-dir เป็นไฟล์ข้อความที่มีการประทับวันที่ (เช่น 2020-09-29-01-05-01) ระบุไดเรกทอรีล่าสุด ไดเรกทอรีที่เหมาะสมคือ AWS หรือ GCP Base URI สำหรับฐานข้อมูล BLAST (เช่น s3://ncbi-blast-databases/ สำหรับ AWS) บวกกับข้อความในไฟล์ latest-dir ตัวอย่าง URI ใน AWS จะเป็น s3://ncbi-blast-databases/2020-09-29-01-05-01 GCP URI จะคล้ายกัน
ข้อความที่ตัดตอนมาจากไฟล์ข้อมูลเมตาแสดงอยู่ด้านล่าง สาขาส่วนใหญ่มีความหมายที่ชัดเจน ไฟล์ประกอบด้วยฐานข้อมูล BLAST ฟิลด์ bytes-total แสดงขนาดฐานข้อมูล BLAST ทั้งหมดในไบต์และมีวัตถุประสงค์เพื่อระบุว่าจำเป็นต้องใช้พื้นที่ดิสก์มากแค่ไหน
ตัวอย่างด้านล่างมาจาก AWS แต่ไฟล์ข้อมูลเมตาบน GCP มีรูปแบบเดียวกัน Databases on the FTP site are in gzipped tarfiles, one per volume of the BLAST database, so those are listed rather than the individual files.
"16S_ribosomal_RNA": {
"version": "1.2",
"dbname": "16S_ribosomal_RNA",
"dbtype": "Nucleotide",
"db-version": 5,
"description": "16S ribosomal RNA (Bacteria and Archaea type strains)",
"number-of-letters": 32435109,
"number-of-sequences": 22311,
"last-updated": "2022-03-07T11:23:00",
"number-of-volumes": 1,
"bytes-total": 14917073,
"bytes-to-cache": 8495841,
"files": [
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ndb",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nog",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nni",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nnd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nsq",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nin",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.ntf",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.not",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nhr",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nos",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/16S_ribosomal_RNA.nto",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.btd",
"s3://ncbi-blast-databases/2020-09-26-01-05-01/taxdb.bti"
]
}
or email us.
National Center for Biotechnology Information (NCBI)
National Library of Medicine (NLM)
National Institutes of Health (NIH)
View refer to the license and copyright information for the software contained in this image.
As with all Docker images, these likely also contain other software which may be under other licenses (such as bash, etc., from the base distribution, along with any direct or indirect dependencies of the primary software being contained).
As with any pre-built image usage, it is the image user's responsibility to ensure that any use of this image complies with any relevant licenses for all software contained within.
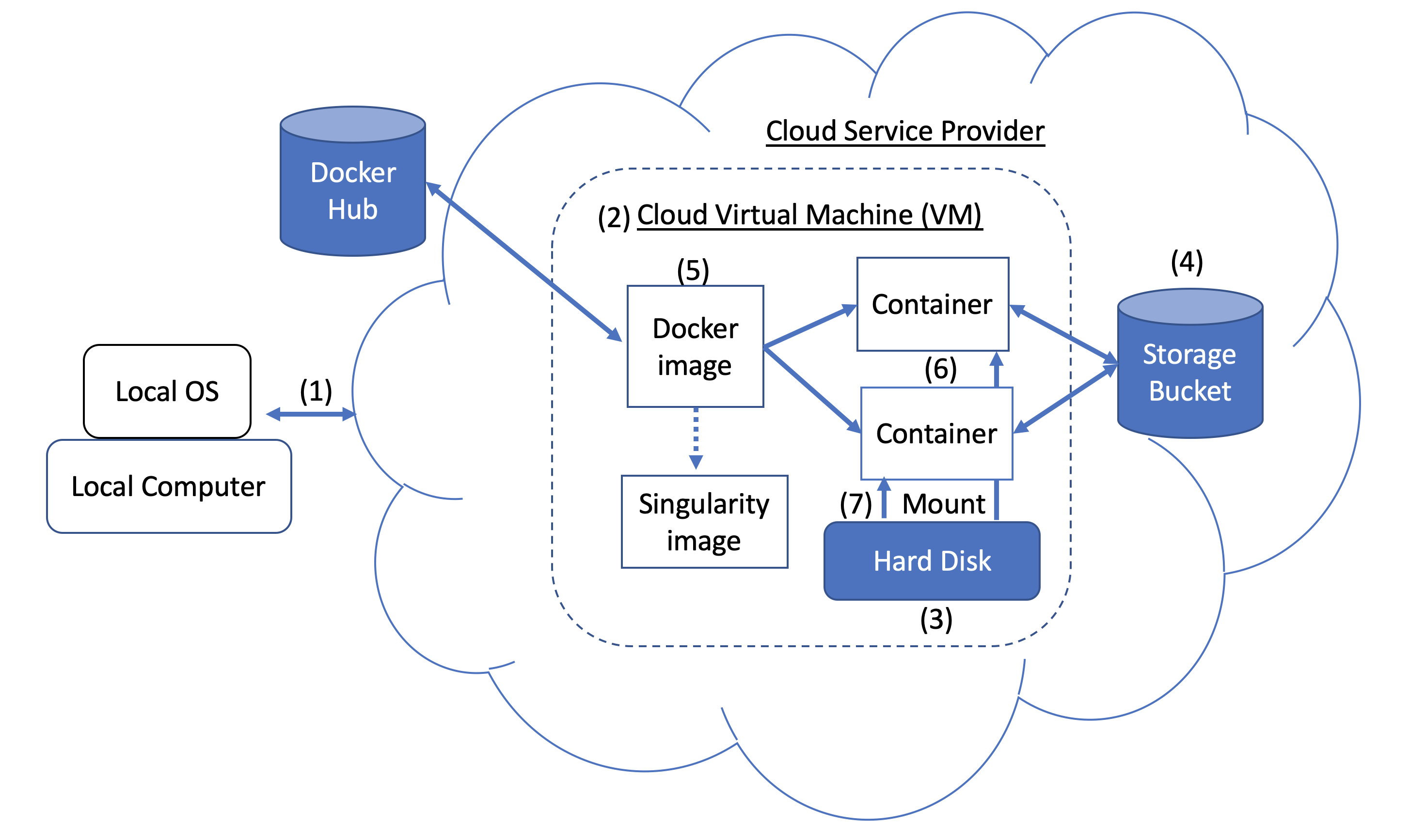 Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
Figure 1. Docker and Cloud Computing Concept. Users can access compute resources provided by cloud service providers (CSPs), such as the Google Cloud Platform, using SSH tunneling (1). When you create a VM (2), a hard disk (also called a boot/persistent disk) (3) is attached to that VM. With the right permissions, VMs can also access other storage buckets (4) or other data repositories in the public domain. Once inside a VM with Docker installed, you can run a Docker image (5), such as NCBI's BLAST image. An image can be used to create multiple running instances or containers (6). Each container is in an isolated environment. In order to make data accessible inside the container, you need to use Docker bind mounts (7) described in this tutorial.
A Docker image can be used to create a Singularity image. Please refer to Singularity's documentation for more detail.
As an alternative to what is described above, you can also run BLAST interactively inside a container.
When to use : This is useful for running a few (eg, fewer than 5-10) BLAST searches on small BLAST databases where you expect the search to complete in seconds/minutes.
docker run --rm -it
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
/bin/bash
# Once you are inside the container (note the root prompt), run the following BLAST commands.
blastp -query /blast/queries/P01349.fsa -db nurse-shark-proteins
-out /blast/results/blastp.out
# To view output, run the following command
more /blast/results/blastp.out
# Leave container
exit
In addition, you can run BLAST in detached mode by running a container in the background.
When to use : This is a more practical approach if you have many (eg, 10 or more) BLAST searches to run or you expect the search to take a long time to execute. In this case it may be better to start the BLAST container in detached mode and execute commands on it.
NOTE : Be sure to mount all required directories, as these need to be specified when the container is started.
# Start a container named 'blast' in detached mode
docker run --rm -dit --name blast
-v $HOME/blastdb:/blast/blastdb:ro -v $HOME/blastdb_custom:/blast/blastdb_custom:ro
-v $HOME/queries:/blast/queries:ro
-v $HOME/results:/blast/results:rw
ncbi/blast
sleep infinity
# Check the container is running in the background
docker ps -a
docker ps --filter "status=running"
Once the container is confirmed to be running in detached mode, run the following BLAST command.
docker exec blast blastp -query /blast/queries/P01349.fsa
-db nurse-shark-proteins -out /blast/results/blastp.out
# View output
more $HOME/results/blastp.out
# stop the container
docker stop blast
If you run into issues with docker stop blast command, reset the VM from the GCP Console or restart the SSH session.
To copy the file $HOME/script.out in the home directory on a local machine to the home directory on a GCP VM named instance-1 in project My First Project using GCP Cloud SDK.
GCP documentation
First install GCP Cloud SDK command line tools for your operating system.
# First, set up gcloud tools
# From local machine's terminal
gcloud init
# Enter a configuration name
# Select the sign-in email account
# Select a project, for example “my-first-project”
# Select a compute engine zone, for example, “us-east4-c”
# To copy the file $HOME/script.out to the home directory of GCP instance-1
# Instance name can be found in your Google Cloud Console -> Compute Engine -> VM instances
gcloud compute scp $HOME/script.out instance-1:~
# Optional - to transfer the file from the GCP instance to a local machine's home directory
gcloud compute scp instance-1:~/script.out $HOME/.