พื้นที่เก็บข้อมูลนี้มีวัสดุเสริมที่มาพร้อมกับกระดาษ "ชุดข้อมูลการโต้ตอบ SARS-COV-2 และคลังข้อมูลลำดับ VHH สำหรับแบบจำลองภาษาแอนติบอดี" ในบทความนี้เราแนะนำ Avida-Sars-Cov-2 ซึ่งเป็นชุดข้อมูลที่มีป้ายกำกับของการโต้ตอบ SARS-COV-2-VHH และ VHHCORPUS-2M ซึ่งมีลำดับ VHH มากกว่าสองล้านลำดับซึ่งจัดทำชุดข้อมูลใหม่สำหรับการประเมินและการฝึกอบรมภาษาแอนติบอดีก่อน ชุดข้อมูลมีอยู่ที่ https://datasets.cognanous.com ภายใต้ใบอนุญาต CC BY-NC 4.0
ในการเริ่มต้นใช้งานโคลนที่เก็บนี้และเรียกใช้คำสั่งต่อไปนี้เพื่อสร้างสภาพแวดล้อมเสมือนจริง
python -m venv ./venv
source ./venv/bin/activate
pip install -r requirements.txt| ชุดข้อมูล | ลิงค์ |
|---|---|
| VHHCORPUS-2M | หน้าโครงการ Hugging Face Hub |
| avida-sars-cov-2 | หน้าโครงการ Hugging Face Hub |
รหัสสำหรับการแปลงข้อมูล RAW (ไฟล์ FASTQ) ที่ได้รับจากการเรียงลำดับรุ่นถัดไป (NGS) เป็นชุดข้อมูลที่มีป้ายกำกับ, Avida-Sars-Cov-2 สามารถพบได้ภายใต้. ./dataset เราเปิดตัวไฟล์ FASTQ สำหรับประเภทแอนติเจน "OC43" ที่นี่เพื่อให้สามารถทำซ้ำการประมวลผลข้อมูลได้
ก่อนอื่นคุณต้องสร้างภาพนักเทียบท่า
docker build -t vhh_constructor:latest ./dataset/vhh_constructor หลังจากวางไฟล์ FASTQ ภายใต้ dataset/raw/fastq ให้ดำเนินการคำสั่งต่อไปนี้เพื่อส่งออกไฟล์ CSV ที่มีป้ายกำกับ
bash ./dataset/preprocess.shVHHBERT เป็นแบบจำลองที่ได้รับการฝึกฝนมาก่อนใน Roberta ในลำดับ VHH สองล้านครั้งใน VHHCORPUS-2M Vhhbert สามารถได้รับการฝึกอบรมล่วงหน้าด้วยคำสั่งต่อไปนี้
python benchmarks/pretrain.py --vocab-file " benchmarks/data/vocab_vhhbert.txt "
--epochs 20
--batch-size 128
--save-dir " outputs "ข้อโต้แย้ง:
| การโต้แย้ง | ที่จำเป็น | ค่าเริ่มต้น | คำอธิบาย |
|---|---|---|---|
| -vocab-file | ใช่ | เส้นทางของไฟล์คำศัพท์ | |
| -EPOCHS | เลขที่ | 20 | จำนวนยุค |
| -ขนาดแบทช์ | เลขที่ | 128 | ขนาดของมินิแบทช์ |
| -เมล็ด | เลขที่ | 123 | เมล็ดสุ่ม |
| ----ไดร์ | เลขที่ | ./saved | เส้นทางของไดเรกทอรีบันทึก |
Vhhbert ที่ผ่านการฝึกอบรมมาก่อนซึ่งได้รับการปล่อยตัวภายใต้ใบอนุญาต MIT นั้นมีอยู่ใน Hugging Face Hub
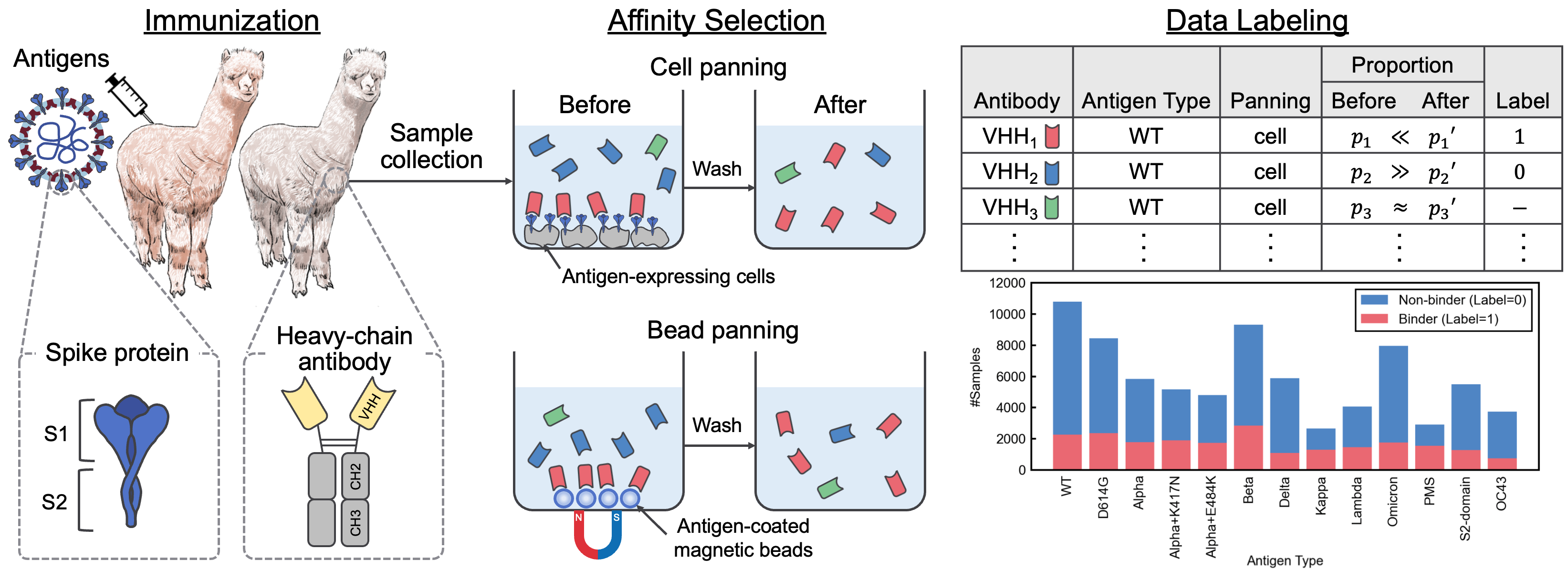
ในการประเมินประสิทธิภาพของแบบจำลองภาษาที่ผ่านการฝึกอบรมมาก่อนสำหรับการค้นพบแอนติบอดีเราได้กำหนดภารกิจการจำแนกแบบไบนารีเพื่อทำนายการผูกมัดหรือไม่ผูกพันของแอนติบอดีที่ไม่รู้จักกับแอนติเจน 13 แอนติเจนโดยใช้ avida-SARS-COV-2 สำหรับข้อมูลเพิ่มเติมเกี่ยวกับงานเปรียบเทียบดูกระดาษ
การปรับแต่งแบบจำลองภาษาสามารถทำได้โดยใช้คำสั่งต่อไปนี้
python benchmarks/finetune.py --palm-type " VHHBERT "
--epochs 30
--batch-size 32
--save-dir " outputs " palm-type ต้องเป็นหนึ่งในสิ่งต่อไปนี้:
VHHBERTVHHBERT-w/o-PTAbLangAntiBERTa2AntiBERTa2-CSSPIgBertProtBertESM-2-150MESM-2-650Mข้อโต้แย้ง:
| การโต้แย้ง | ที่จำเป็น | ค่าเริ่มต้น | คำอธิบาย |
|---|---|---|---|
| -ประเภทปาล์ม | เลขที่ | Vhhbert | ชื่อนางแบบ |
| -ไฟล์ embeddings | เลขที่ | ./benchmarks/data/antigen_embeddings.pkl | Path of Embeddings File สำหรับแอนติเจน |
| -EPOCHS | เลขที่ | 20 | จำนวนยุค |
| -ขนาดแบทช์ | เลขที่ | 128 | ขนาดของมินิแบทช์ |
| -เมล็ด | เลขที่ | 123 | เมล็ดสุ่ม |
| ----ไดร์ | เลขที่ | ./saved | เส้นทางของไดเรกทอรีบันทึก |
หากคุณใช้ avida-sars-cov-2, vhhcorpus-2m หรือ vhhbert ในการวิจัยของคุณโปรดใช้การอ้างอิงต่อไปนี้
@inproceedings { tsuruta2024sars ,
title = { A {SARS}-{C}o{V}-2 Interaction Dataset and {VHH} Sequence Corpus for Antibody Language Models } ,
author = { Hirofumi Tsuruta and Hiroyuki Yamazaki and Ryota Maeda and Ryotaro Tamura and Akihiro Imura } ,
booktitle = { Advances in Neural Information Processing Systems 37 } ,
year = { 2024 }
}