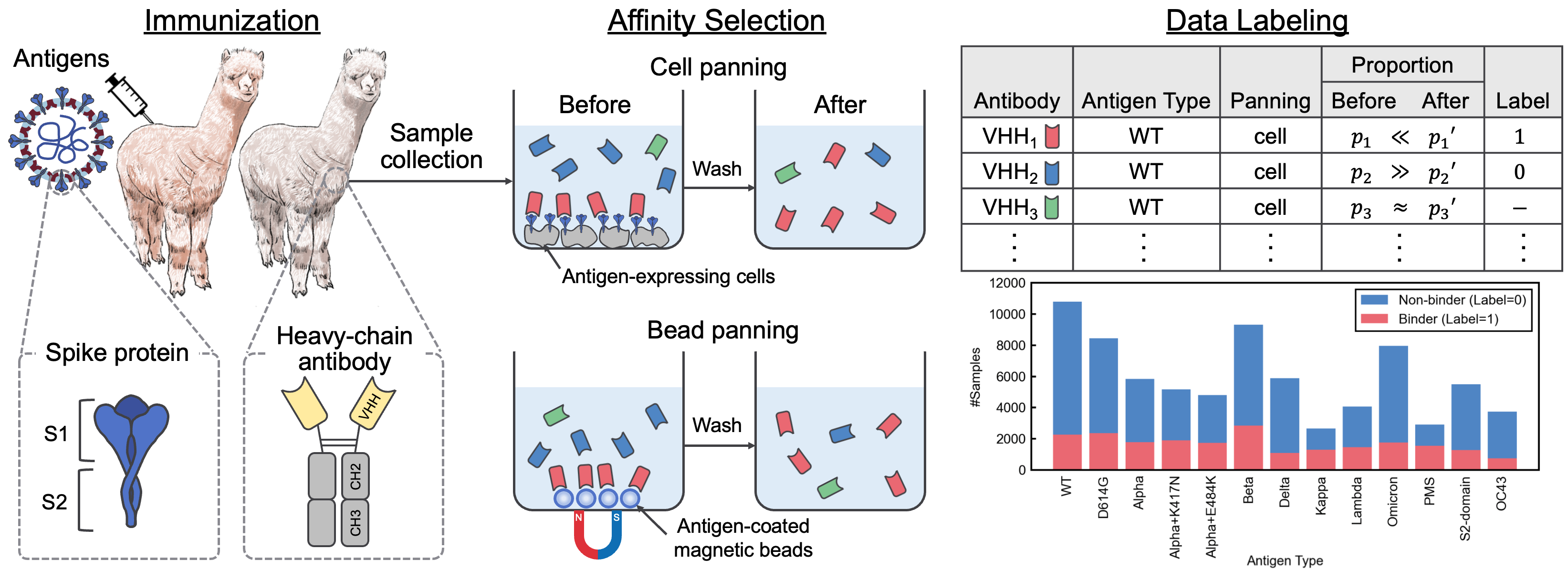
Dieses Repository enthält das ergänzende Material, das das Papier "A SARS-CoV-2-Interaktionsdatensatz und VHH-Sequenzkorpus für Antikörpersprachenmodelle" begleitet. In diesem Artikel haben wir Avida-SARS-CoV-2, einen gekennzeichneten Datensatz von SARS-CoV-2-VHH-Interaktionen, und VHHCorpus-2M eingeführt, das über zwei Millionen VHH-Sequenzen enthält, die neuartige Datensätze für die Bewertung und Vorabbildung von Antikörpersprachmodellen bereitstellen. Die Datensätze sind unter https://datasets.cognanous.com unter einer CC BY-NC 4.0-Lizenz verfügbar.
Klonen Sie dieses Repository, um zu beginnen, und führen Sie den folgenden Befehl aus, um eine virtuelle Umgebung zu erstellen.
python -m venv ./venv
source ./venv/bin/activate
pip install -r requirements.txt| Datensatz | Links |
|---|---|
| Vhhcorpus-2m | Umarmung der Gesichts -Hub -Projektseite |
| Avida-SARS-COV-2 | Umarmung der Gesichts -Hub -Projektseite |
Der Code zum Konvertieren der Rohdaten (FASTQ-Datei), die aus der Sequenzierung der nächsten Generation (NGS) in den gekennzeichneten Datensatz Avida-SARS-CoV-2 erhalten wurde, finden Sie unter ./dataset . Wir haben die Fastq -Dateien für den Antigentyp "OC43" hier veröffentlicht, damit die Datenverarbeitung reproduziert werden kann.
Zuerst müssen Sie ein Docker -Bild erstellen.
docker build -t vhh_constructor:latest ./dataset/vhh_constructor Führen Sie nach dem Platzieren der Fastq -Dateien unter dataset/raw/fastq den folgenden Befehl aus, um eine gekennzeichnete CSV -Datei auszugeben.
bash ./dataset/preprocess.shVHHBERT ist ein in Roberta ansässiges Modell, das auf zwei Millionen VHH-Sequenzen in VHHCorpus-2M vorgebracht ist. Vhhbert kann mit den folgenden Befehlen vorgeblendet werden.
python benchmarks/pretrain.py --vocab-file " benchmarks/data/vocab_vhhbert.txt "
--epochs 20
--batch-size 128
--save-dir " outputs "Argumente:
| Argument | Erforderlich | Standard | Beschreibung |
|---|---|---|---|
| -Vocab-Datei | Ja | Pfad der Wortschatzdatei | |
| --epochs | NEIN | 20 | Anzahl der Epochen |
| -Batchgröße | NEIN | 128 | Größe des Mini-Batch |
| --Samen | NEIN | 123 | Zufälliger Samen |
| -Save-Dir | NEIN | ./Save | Pfad des Save -Verzeichnisses |
Der vorgeborene Vhhbert, der unter der MIT-Lizenz veröffentlicht wurde, ist im Umarmungs-Face-Hub erhältlich.
Um die Leistung verschiedener vorgebildeter Sprachmodelle für die Entdeckung von Antikörpern zu bewerten, haben wir eine binäre Klassifizierungsaufgabe definiert, um die Bindung oder Nichtbindung unbekannter Antikörper gegen 13 Antigene mit Avida-SARS-CoV-2 vorherzusagen. Weitere Informationen zur Benchmarking -Aufgabe finden Sie in der Zeitung.
Die Feinabstimmung der Sprachmodelle kann mit dem folgenden Befehl durchgeführt werden.
python benchmarks/finetune.py --palm-type " VHHBERT "
--epochs 30
--batch-size 32
--save-dir " outputs " palm-type muss einer der folgenden sein:
VHHBERTVHHBERT-w/o-PTAbLangAntiBERTa2AntiBERTa2-CSSPIgBertProtBertESM-2-150MESM-2-650MArgumente:
| Argument | Erforderlich | Standard | Beschreibung |
|---|---|---|---|
| -Palm-Typ | NEIN | Vhhbert | Modellname |
| -Embeddings-Datei | NEIN | ./benchmarks/data/anigen_embeddings.pkl | Pfad der Einbettungsdatei für Antigene |
| --epochs | NEIN | 20 | Anzahl der Epochen |
| -Batchgröße | NEIN | 128 | Größe des Mini-Batch |
| --Samen | NEIN | 123 | Zufälliger Samen |
| -Save-Dir | NEIN | ./Save | Pfad des Save -Verzeichnisses |
Wenn Sie Avida-SARS-COV-2, VHHCORPUS-2M oder VHHBERT in Ihrer Forschung verwenden, verwenden Sie bitte das folgende Zitat.
@inproceedings { tsuruta2024sars ,
title = { A {SARS}-{C}o{V}-2 Interaction Dataset and {VHH} Sequence Corpus for Antibody Language Models } ,
author = { Hirofumi Tsuruta and Hiroyuki Yamazaki and Ryota Maeda and Ryotaro Tamura and Akihiro Imura } ,
booktitle = { Advances in Neural Information Processing Systems 37 } ,
year = { 2024 }
}