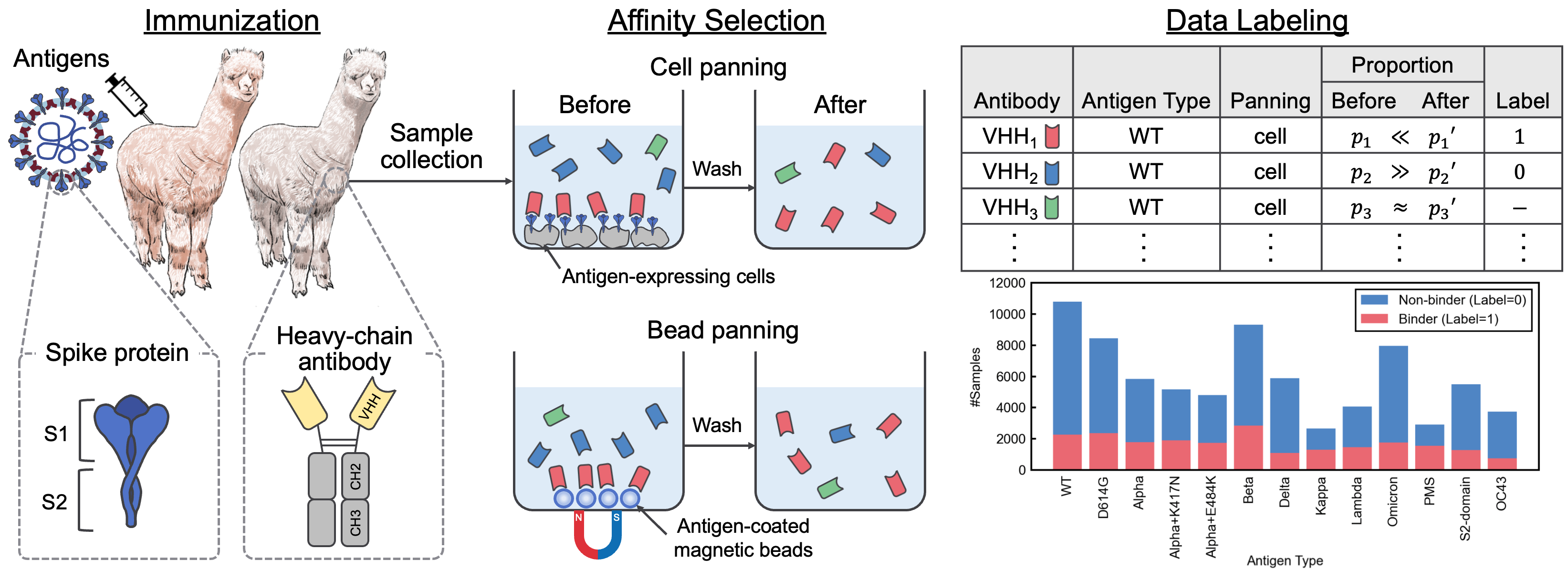
Ce référentiel contient le matériel supplémentaire accompagnant le document "un ensemble de données d'interaction SARS-COV-2 et un corpus de séquence VHH pour les modèles de langage d'anticorps". Dans cet article, nous avons introduit Avida-Sars-CoV-2, un ensemble de données étiqueté des interactions SARS-COV-2-VHH, et VHHCORPUS-2M, qui contient plus de deux millions de séquences VHH, fournissant de nouveaux ensembles de données pour l'évaluation et la pré-entraînement des modèles linguistiques d'anticorps. Les ensembles de données sont disponibles sur https://datasets.cognanous.com sous une licence CC BY-NC 4.0.
Pour commencer, clonez ce référentiel et exécutez la commande suivante pour créer un environnement virtuel.
python -m venv ./venv
source ./venv/bin/activate
pip install -r requirements.txt| Ensemble de données | Links |
|---|---|
| Vhhcorpus-2m | Page de projet HUNGING FACE HUB |
| Avida-sars-CoV-2 | Page de projet HUNGING FACE HUB |
Le code de conversion des données brutes (fichier fastq) obtenu à partir de séquençage de nouvelle génération (NGS) en ensemble de données étiqueté, Avida-Sars-CoV-2, peut être trouvé sous ./dataset . Nous avons publié les fichiers FastQ pour le type d'antigène "OC43" ici afin que le traitement des données puisse être reproduit.
Tout d'abord, vous devez créer une image Docker.
docker build -t vhh_constructor:latest ./dataset/vhh_constructor Après avoir placé les fichiers FastQ sous dataset/raw/fastq , exécutez la commande suivante pour sortir un fichier CSV étiqueté.
bash ./dataset/preprocess.shVHHBERT est un modèle basé à Roberta pré-formé sur deux millions de séquences VHH dans VHHCORPUS-2M. Vhhbert peut être formé avec les commandes suivantes.
python benchmarks/pretrain.py --vocab-file " benchmarks/data/vocab_vhhbert.txt "
--epochs 20
--batch-size 128
--save-dir " outputs "Arguments:
| Argument | Requis | Défaut | Description |
|---|---|---|---|
| - Vocab-file | Oui | Chemin du fichier de vocabulaire | |
| - époques | Non | 20 | Nombre d'époches |
| --size | Non | 128 | Taille du mini-lot |
| --graine | Non | 123 | Semences aléatoires |
| --Save-Dir | Non | ./Saved | Chemin du répertoire de sauvegarde |
Le VHHBERT pré-formé, libéré sous la licence du MIT, est disponible sur le HUBGING FACE HUB.
Pour évaluer les performances de divers modèles de langue pré-formés pour la découverte des anticorps, nous avons défini une tâche de classification binaire pour prédire la liaison ou la non-contrainte des anticorps inconnus à 13 antigènes à l'aide d'Avida-Sars-CoV-2. Pour plus d'informations sur la tâche d'analyse comparative, consultez le document.
Le réglage fin des modèles de langue peut être effectué à l'aide de la commande suivante.
python benchmarks/finetune.py --palm-type " VHHBERT "
--epochs 30
--batch-size 32
--save-dir " outputs " palm-type doit être l'un des éléments suivants:
VHHBERTVHHBERT-w/o-PTAbLangAntiBERTa2AntiBERTa2-CSSPIgBertProtBertESM-2-150MESM-2-650MArguments:
| Argument | Requis | Défaut | Description |
|---|---|---|---|
| - Type Palm | Non | Vhhbert | Nom du modèle |
| --eddings-file | Non | ./benchmarks/data/antigen_embeddings.pkl | Chemin de fichier d'intégration pour les antigènes |
| - époques | Non | 20 | Nombre d'époches |
| --size | Non | 128 | Taille du mini-lot |
| --graine | Non | 123 | Semences aléatoires |
| --Save-Dir | Non | ./Saved | Chemin du répertoire de sauvegarde |
Si vous utilisez AVIDA-SARS-COV-2, VHHCORPUS-2M ou VHHBERT dans votre recherche, veuillez utiliser la citation suivante.
@inproceedings { tsuruta2024sars ,
title = { A {SARS}-{C}o{V}-2 Interaction Dataset and {VHH} Sequence Corpus for Antibody Language Models } ,
author = { Hirofumi Tsuruta and Hiroyuki Yamazaki and Ryota Maeda and Ryotaro Tamura and Akihiro Imura } ,
booktitle = { Advances in Neural Information Processing Systems 37 } ,
year = { 2024 }
}