ตัวอย่างแพ็คเกจ R สำหรับ BCB410H: BioInformatics Applied
ย่อหน้าที่อธิบายวัตถุประสงค์ของแพ็คเกจ R และข้อมูลทางชีวภาพของคุณที่ถูกวิเคราะห์ อธิบายว่าแพ็คเกจของคุณเพิ่มหรือปรับปรุงกระแสงานปัจจุบันในชีวสารสนเทศศาสตร์หรือชีววิทยาการคำนวณ (เช่นมันเป็นเอกลักษณ์อย่างไร, ปัญหาอะไรที่อยู่?) ในที่สุดรวมรุ่น R (ไม่ใช่เวอร์ชัน RSTUDIO) และแพลตฟอร์ม (MAC, Windows, Linux (Debian, Fedora/Redhat, Ubuntu)) ใช้ในการพัฒนาแพ็คเกจ คุณอาจได้รับข้อมูลนี้โดยเรียก utils::sessionInfo() ไม่ควรมีการใช้งาน Shiny ณ จุดนี้ เช่น
TestingPackage เป็นแพ็คเกจ R เพื่อสาธิตส่วนประกอบของแพ็คเกจ R ง่าย ๆ พร้อมข้อมูลการจัดลำดับ RNA แพ็คเกจ R รวมถึงส่วนประกอบหลัก: คำอธิบายเนมสเปซไดเรกทอรีย่อย MAN และไดเรกทอรีย่อย R นอกจากนี้ยังมีการสำรวจใบอนุญาต, readMe และไดเรกทอรีย่อย, การทดสอบ, ข้อมูลและ Inst แพ็คเกจนี้มีเป้าหมายสำหรับนักเรียน BCB410H (Bioinformatics ที่ใช้แล้ว) ซึ่งจะกำหนดเครื่องมือที่มีประโยชน์สำหรับการวิเคราะห์ข้อมูลทางชีวภาพในรูปแบบของแพ็คเกจ R สาธารณะที่ตั้งอยู่บน GitHub ขอบเขตของแพ็คเกจ R คือการเพิ่มหรือปรับปรุงกระแสงานปัจจุบันในชีวสารสนเทศศาสตร์หรือชีววิทยาการคำนวณ เครื่องมือควรมีฟังก์ชั่นเพื่อทำการวิเคราะห์ข้อมูลทางชีวภาพและเพื่อสร้างเอาต์พุตกราฟิกที่น่าสนใจเพื่อสนับสนุนการวิเคราะห์เชิงสำรวจ แพ็คเกจ TestingPackage ได้รับการพัฒนาโดยใช้ R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) และ Running under: macOS Ventura 13.2
ระบุข้อความและคำสั่งต่อไปนี้ปรับแต่งให้กับแพ็คเกจ R ของคุณ เช่น
ในการติดตั้งแพ็คเกจเวอร์ชันล่าสุด:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )เพื่อเรียกใช้แอพเงา:
runTestingPackage() # not for Assessment 4; only for Assessment 5 ระบุคำสั่งต่อไปนี้ปรับแต่งตามแพ็คเกจ R ของคุณ จากนั้นจัดทำรายการฟังก์ชั่นที่สามารถเข้าถึงได้ของผู้ใช้ภายในแพ็คเกจและคำอธิบายสั้น ๆ ของแต่ละรายการ รวมภาพหนึ่งภาพที่แสดงภาพรวมของแพ็คเกจที่แสดงอินพุตและเอาต์พุต ตรวจสอบให้แน่ใจว่าภาพถูกฝากไว้ในตำแหน่งที่ถูกต้องตามที่กล่าวไว้ในชั้นเรียน ชี้ผู้ใช้ไปยังบทความสั้น ๆ สำหรับการสอนแพ็คเกจของคุณ เช่น
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage ประกอบด้วย 3 ฟังก์ชั่น
Infcriteriaculation สำหรับการคำนวณเกณฑ์ข้อมูลที่กำหนดมิติชุดข้อมูลความน่าจะเป็นบันทึกและความน่าจะเป็น
normfactors สำหรับการคำนวณปัจจัยการทำให้เป็นมาตรฐานผ่านทางค่าเฉลี่ยของค่า M (TMM)
Infcriteriaplot สำหรับการพล็อตค่าเกณฑ์ข้อมูลเป็นพล็อตกระจาย
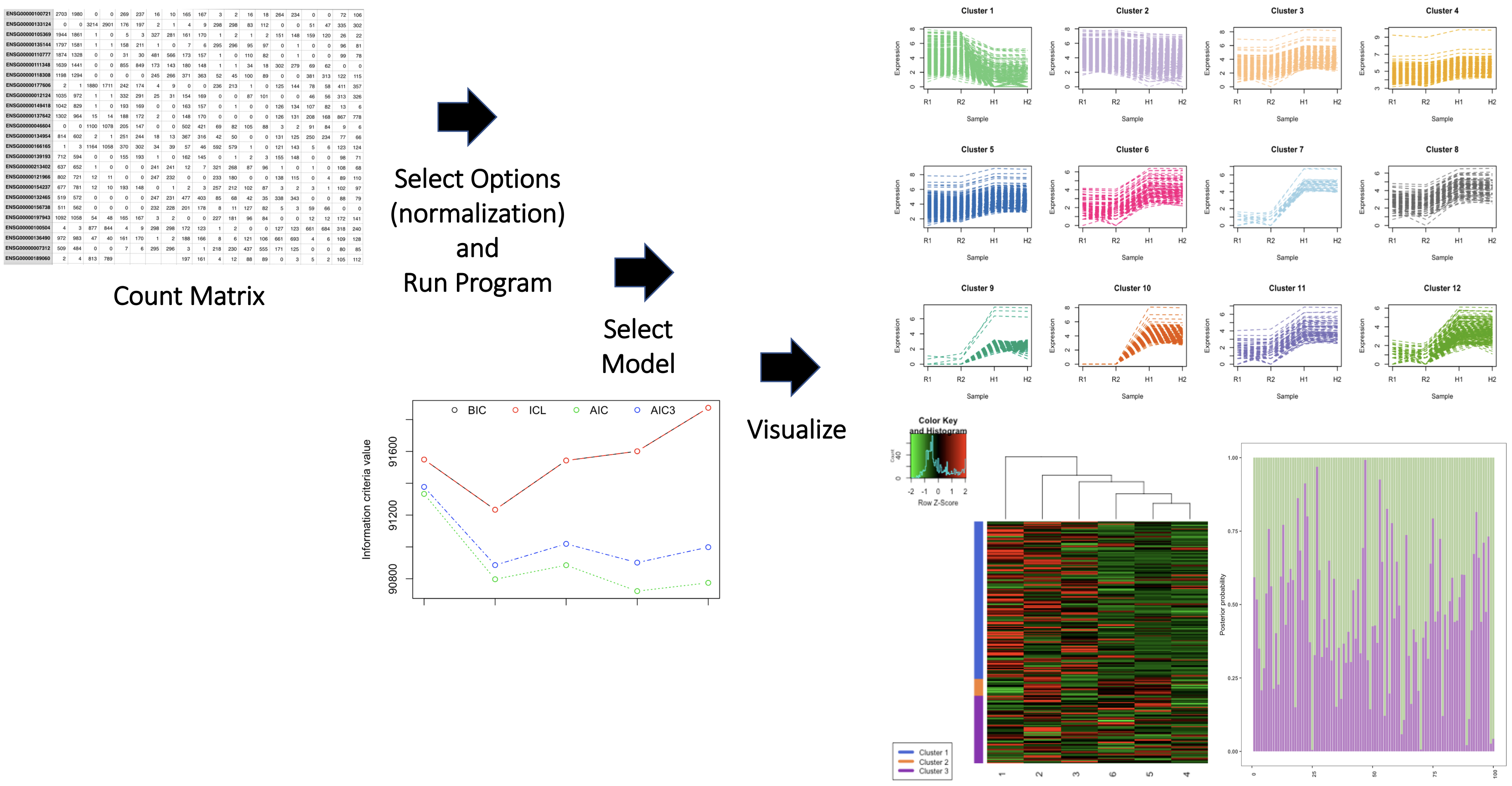
แพ็คเกจยังมีชุดข้อมูลลำดับ RNA สองชุดเรียกว่า Genecounts และ Genecounts2 อ้างถึง Vignettes แพ็คเกจสำหรับรายละเอียดเพิ่มเติม ภาพรวมของแพ็คเกจแสดงไว้ด้านล่าง
ระบุย่อหน้าที่ระบุชื่อของผู้แต่งแพ็คเกจและการมีส่วนร่วมจากผู้เขียนอย่างชัดเจน โครงร่างการมีส่วนร่วมจากแพ็คเกจ/แหล่งข้อมูลอื่น ๆ สำหรับแต่ละฟังก์ชั่น โครงร่างการมีส่วนร่วมจากเครื่องมือ AI แบบกำเนิดสำหรับแต่ละฟังก์ชั่น รวมถึงวิธีการใช้เครื่องมือและวิธีการรวมผลลัพธ์จากเครื่องมือ AI จำไว้ว่าการมีส่วนร่วมของคุณในแพ็คเกจนั้นมีความสำคัญ เช่น
ผู้เขียนแพ็คเกจคือ Anjali Silva ผู้เขียนเขียนฟังก์ชั่น infcriteriaculation ซึ่งคำนวณค่าเกณฑ์ข้อมูลที่ได้รับข้อมูลจำเพาะข้อมูล ที่นี่เกณฑ์ข้อมูลเบย์ (BIC), Akaike Information Criterion (AIC) และการคำนวณความน่าจะเป็นแบบรวม (ICL) ถูกคำนวณ ฟังก์ชั่น infcriteriaculation ใช้ประโยชน์จากฟังก์ชันแผนที่จากแพ็คเกจ mclust R เพื่อสร้างค่าเกณฑ์ข้อมูล แพ็คเกจ stats R ใช้สำหรับการสร้างเวกเตอร์หมายเลขสุ่มแบบกระจายแบบหลายระดับ ส่วนหนึ่งของรหัสสำหรับฟังก์ชั่น การคำนวณแบบ infcriteriaculation ได้มาจากแพ็คเกจ <NamePackage> r (ส่วนของรหัสที่ยืมควรระบุและอ้างอิงอย่างชัดเจนในสคริปต์ infcriteriaculation R) Infcriteriaplot เขียนโดยผู้เขียนและสร้างพล็อตของค่าเกณฑ์ข้อมูล ฟังก์ชั่น infcriteriaplot ใช้ประโยชน์จากแพ็คเกจ graphics R Normfactors เป็นฟังก์ชั่นที่คำนวณปัจจัยการทำให้เป็นมาตรฐานผ่านค่าเฉลี่ยของค่า M (TMM) ฟังก์ชั่น Normfactors ใช้ค่าเฉลี่ยของค่า M-values (TMM) ตามที่ใช้ในแพ็คเกจ edgeR R ไม่มีการใช้เครื่องมือ AI แบบกำเนิดในการพัฒนาแพ็คเกจนี้
ให้การอ้างอิงเต็มรูปแบบสำหรับทุกแหล่งที่ใช้รวมถึงแพ็คเกจและเครื่องมือที่กล่าวถึงภายใต้ 'การบริจาค' ในรูปแบบเดียว เช่น
Akaike, H. (1973) ทฤษฎีข้อมูลและส่วนขยายของหลักการโอกาสสูงสุด ใน การประชุมวิชาการนานาชาติครั้งที่สองเกี่ยวกับทฤษฎีข้อมูล นิวยอร์กสหรัฐอเมริกา 267–281 Springer Verlag https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15
Biernacki, C. , G. Celeux และ G. Govaert (2000) การประเมินแบบจำลองการผสมสำหรับการจัดกลุ่มด้วยโอกาสการจำแนกแบบบูรณาการ การทำธุรกรรม IEEE เกี่ยวกับการวิเคราะห์รูปแบบและข่าวกรองเครื่องจักร 22. https://hal.inria.fr/inria-00073163/document
Biorender (2020) รูปภาพที่สร้างโดย Silva, A. สืบค้น 30 ตุลาคม 2563 จาก https://app.biorender.com/
Chang, W. , J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023) Shiny: Web Application Framework สำหรับ r . r แพ็คเกจเวอร์ชัน 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen และ GK Smyth (2012) การวิเคราะห์การแสดงออกที่แตกต่างกันของการทดลอง RNA-seq แบบมัลติปัจจัยที่เกี่ยวข้องกับการเปลี่ยนแปลงทางชีวภาพ การวิจัยกรดนิวคลีอิก 40 4288-4297 https://pubmed.ncbi.nlm.nih.gov/22287627/
R Core Team (2023) R: ภาษาและสภาพแวดล้อมสำหรับการคำนวณทางสถิติ R Foundation สำหรับการคำนวณทางสถิติ, เวียนนา, ออสเตรีย https://www.r-project.org/
Robinson, MD, DJ McCarthy และ GK Smyth (2010) Edger: แพ็คเกจ bioconductor สำหรับการวิเคราะห์การแสดงออกที่แตกต่างกันของข้อมูลการแสดงออกของยีนดิจิตอล ชีวสารสนเทศศาสตร์ 26, 139-140 https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978) การประมาณมิติของแบบจำลอง พงศาวดารของสถิติ 6, 461–464 https://projecteuclid.org/euclid.aos/1176344136
Scrucca, L. , M. FOP, TB Murphy และ AE Raftery (2016) McLust 5: การจัดกลุ่ม, การจำแนกประเภทและการประมาณความหนาแน่นโดยใช้แบบจำลองผสม Gaussian Finite วารสาร R 8 (1), 289-317 https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. Ggplot2: กราฟิกที่สง่างามสำหรับการวิเคราะห์ข้อมูล Springer-Verlag New York, 2016
Wickham, H. และ J. Bryan (2019) R แพ็คเกจ (ฉบับที่ 2) นิวตัน, แมสซาชูเซตส์: O'Reilly Media https://r-pkgs.org/
ระบุข้อความต่อไปนี้ปรับแต่งให้กับแพ็คเกจ R ของคุณ เช่น
แพ็คเกจนี้ได้รับการพัฒนาเป็นส่วนหนึ่งของการประเมินสำหรับปี 2562-2566 BCB410H: หลักสูตรชีวสารสนเทศศาสตร์ที่มหาวิทยาลัยโตรอนโต, โตรอนโต, แคนาดา TestingPackage ยินดีต้อนรับปัญหาการร้องขอการปรับปรุงและการมีส่วนร่วมอื่น ๆ หากต้องการส่งปัญหาให้ใช้ปัญหา GitHub ขอบคุณมากสำหรับผู้ที่ให้ข้อเสนอแนะเพื่อปรับปรุงแพ็คเกจนี้
แพ็คเกจที่พัฒนาโดยศิษย์เก่า BCB410 ขอบคุณมากสำหรับผู้ที่อนุญาตให้แบ่งปันแพ็คเกจของพวกเขา!
แอตลาซ
ClinicalTranscriptLink
CompHeatmaps
Covidwastewatch
ยาเสพติด
geanaly
การรวมเข้าด้วยกัน
isoformvisrna
microbiomexplorer
Missensepathor
Openvirome
osteoanalizer
pdbcleanup
ฟีโนเจนนิรลิบ
Prostat5
psychcnvassoc
Seurattogo
TMSCOREALIGN
Trexdad
trnvalstandvis
Abunrna
dereggenes
CNVDS
การวิเคราะห์
sexdisaggregate
scrgnet
deviscomp
Covid19canada
การรวมกลุ่ม
Myomanager
snpcyp
VisualSarsdiff
คลาร์ก
สิ่งที่มีหนอง
Gscvisualizer
ไมโครคอมมิวต์
uniprotproteinview
methylexpress
Dynugene; ต้นฉบับ
PDB3D
Strexpansionanalyzer
Celltypetool
ชาว Chipanalyzer
โครงสร้างแพ็คเกจแสดงไว้ด้านล่าง:
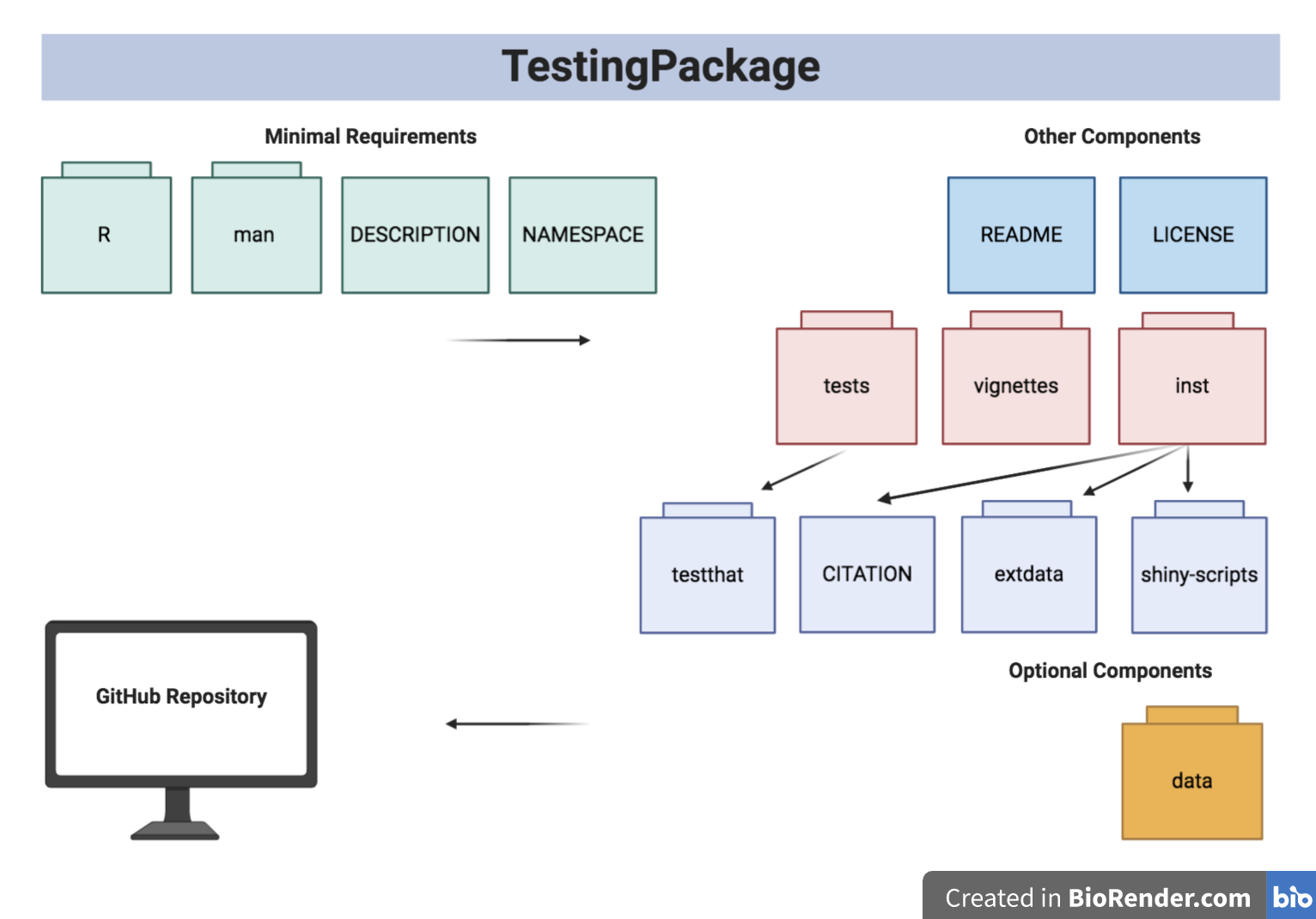
โครงสร้างต้นไม้แพ็คเกจมีอยู่ด้านล่าง
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R