Пример R -пакета для BCB410H: Applied Bioinformatics.
Абзац, который описывает цель вашего пакета R и анализируемых биологических данных. Объясните, как ваш пакет добавляет или улучшает текущий рабочий процесс в биоинформатике или вычислительной биологии (т.е. как он уникален?, Какую проблему он решает?). Наконец, включите версию R (не Rstudio версия) и платформу (Mac, Windows, Linux (Debian, Fedora/Redhat, Ubuntu)), используется для разработки пакета. Вы можете получить эту информацию, запустив utils::sessionInfo() . На этом этапе не должно быть Shiny реализации. Например,
TestingPackage - это R -пакет для демонстрации компонентов простого пакета R с данными секвенирования РНК. Пакет R включает в себя основные компоненты: описание, пространство имен, мужчина подкаталог и R -подкаталог. Кроме того, также изучаются лицензии, чтения и подкатализаторы. Пакет предназначен для студентов BCB410H (Applied Bioinformatics), которые должны определить полезный инструмент для анализа биологических данных в формате публичного пакета R, размещенного на GitHub. Объем пакета R состоит в том, чтобы добавить или улучшить текущий рабочий процесс в биоинформатике или вычислительной биологии. Инструмент должен содержать функции для проведения анализа биологических данных и создания убедительного графического вывода, в идеале для поддержки исследовательского анализа. Пакет TestingPackage был разработан с использованием R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) и Running under: macOS Ventura 13.2 .
Предоставьте следующий текст и команды, настроенные на ваш пакет R. Например,
Чтобы установить последнюю версию пакета:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )Чтобы запустить блестящее приложение:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Предоставьте следующие команды, настроенные на ваш пакет R. Затем предоставьте список доступных пользователей функций в пакете и краткое описание каждого. Включите одно изображение, иллюстрирующее обзор пакета, который показывает входы и выходы. Убедитесь, что изображение хранится в правильном месте, как обсуждалось в классе. Назначьте пользователя Vignettes для учебного пособия по вашему пакету. Например,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage содержит 3 функции.
Infcrietiacalculation для расчета информационных критериев. Указанные измерения наборов данных, логарифмическая правдоподобие и вероятность.
NormFactors для расчета коэффициентов нормализации через обрезанное среднее значение M-значений (TMM).
InfcriteriAplot для построения значений информационных критериев в качестве графика рассеяния.
Пакет также содержит два набора данных секвенирования РНК, называемые GeneCounts и GeneCounts2. Обратитесь к пакетным виньеткам для более подробной информации. Обзор пакета показан ниже.
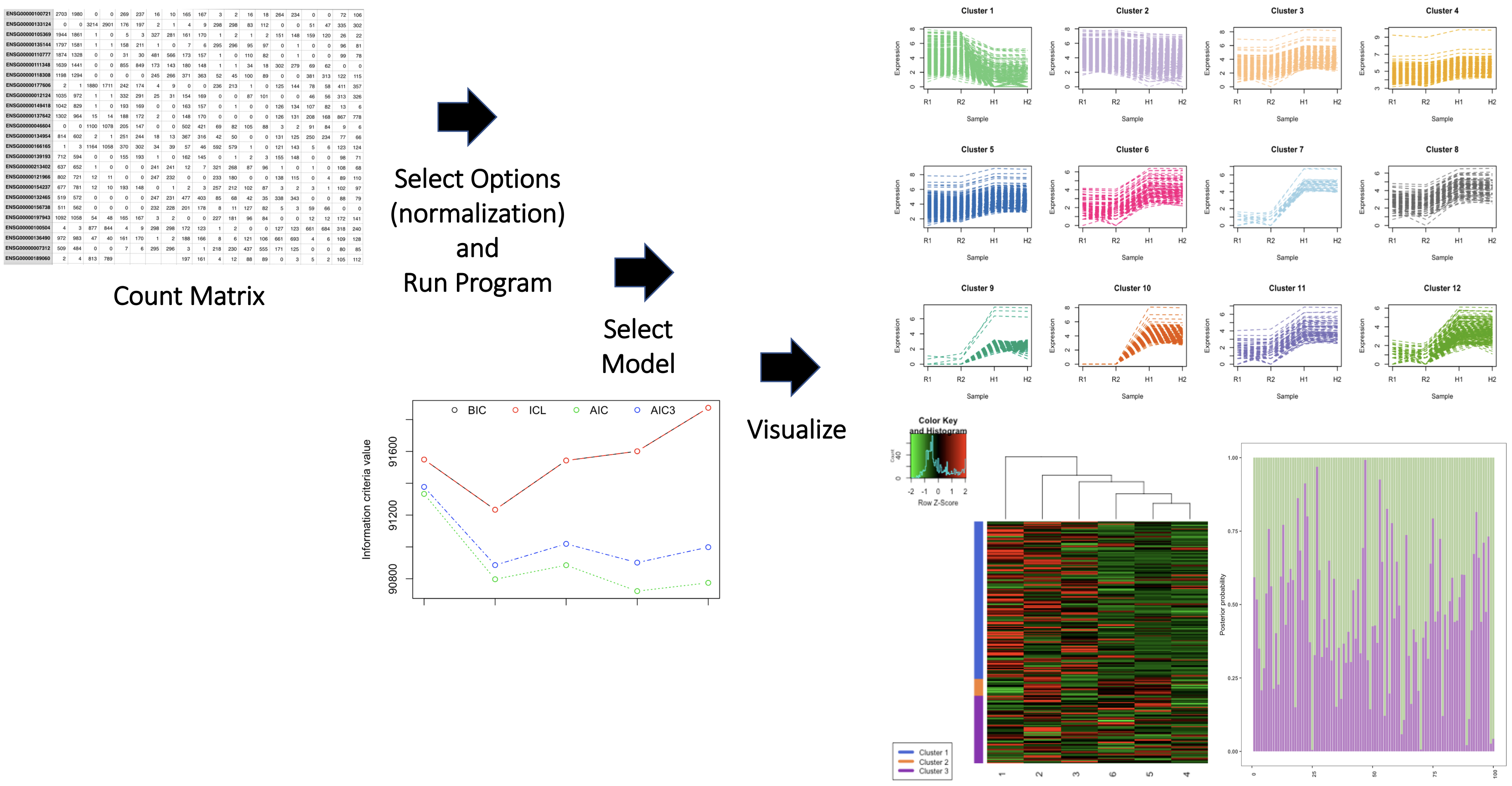
Предоставьте абзац, четко указывающий на имя автора пакета и взносы автора. Определите вклад из других пакетов/источников для каждой функции. Определите вклад из генеративного инструмента (ы) ИИ для каждой функции. Включите, как использовались инструменты и как были включены результаты инструментов AI. Помните, что ваш индивидуальный вклад в пакет важен. Например,
Автор пакета - Анджали Сильва. Автор написал функцию инфритериализации , которая вычисляет значения информационных критериев с учетом спецификаций данных. Здесь рассчитываются байесовский информационный критерий (BIC), информационный критерий AKAIKE (AIC) и интегрированная полная вероятность (ICL). Функция инфритериализации использует функцию MAP из пакета mclust R для генерации значений информационных критериев. Пакет stats R используется для генерации мультиномиально распределенных векторов случайных чисел. Часть Кодекса для функции инфритериализации была взята из пакета <NamePackage> r. (Раздел заимствованного кода должен быть четко обозначен и упоминается в сценарии r Infriceriacalculation). InfcriperiAplot написан автором и генерирует участок значений информационных критериев. Функция InfcriteriAplot использует пакет graphics R. NormFactors -это функция, которая вычисляет коэффициенты нормализации через обрезанное среднее значение M-значений (TMM). Функция NormFactors использует триммированное среднее значение M-значений (TMM), как это реализовано в пакете edgeR R. В разработке этого пакета не использовались генеративные инструменты ИИ.
Предоставьте полные ссылки для всех используемых источников, в том числе для пакетов и инструментов, упомянутых в «Взносах», в одном формате. Например,
Akaike, H. (1973). Теория информации и расширение принципа максимального правдоподобия. Во втором международном симпозиуме по теории информации , Нью -Йорк, США, 267–281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux и G. Govaert (2000). Оценка модели смеси для кластеризации с помощью интегрированной вероятности классификации. IEEE транзакции по анализу шаблонов и машинного интеллекта 22. https://hal.inria.fr/inria-00073163/document
Биоредер. (2020). Изображение, созданное Сильвой, А. Получено 30 октября 2020 года, с https://app.biorender.com/
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023). Блестящий: веб -фреймворк для р . R Версия пакета 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen и GK Smyth (2012). Дифференциальный анализ экспрессии экспериментов по многофакторной РНК-seq относительно биологических изменений. Исследование нуклеиновых кислот 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R Core Team (2023). R: Язык и среда для статистических вычислений. R Фонд статистических вычислений, Вена, Австрия. https://www.r-project.org/
Робинсон, MD, DJ McCarthy and GK Smyth (2010). Edger: пакет биоконкуртировщиков для анализа дифференциальной экспрессии цифровых данных экспрессии генов. Биоинформатика 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). Оценка измерения модели. Анналы статистики 6, 461–464. https://projecteuclid.org/euclid.aos/1176344136.
Scrucca, L., M. Fop, TB Murphy и AE Raftery (2016) McLust 5: кластеризация, классификация и оценка плотности с использованием гауссовских моделей конечных смесей. Журнал R 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. Ggplot2: Элегантная графика для анализа данных. Springer-Verlag New York, 2016.
Уикхем Х. и Дж. Брайан (2019). R пакетов (2 -е издание). Ньютон, Массачусетс: O'Reilly Media. https://r-pkgs.org/
Предоставьте следующий текст, настроенный на ваш пакет R. Например,
Этот пакет был разработан в рамках оценки на 2019-2023 гг. Bcb410H: прикладная биоинформатика в Университете Торонто, Торонто, Канада. TestingPackage приветствует проблемы, запросы на улучшение и другие взносы. Чтобы отправить проблему, используйте проблемы GitHub. Большое спасибо тем, кто предоставил обратную связь, чтобы улучшить этот пакет.
Пакеты, разработанные выпускниками BCB410. Большое спасибо тем, кто предоставил разрешение поделиться своими пакетами!
Атлаз
ClinicalTranscriptLink
Compheatmaps
Covidwastewatch
Drugseekr
Жаналия
Интеграл
Изоформвисрна
MicrobiomeExplorer
MissensePathor
OpenVirome
Остеоанализатор
PDBCleanup
Феногенрлиб
Простат5
Psychcnvassoc
Seurattogo
TMSCOREALIGN
Трексдад
Trnvalstandvis
Абунрна
Дерегены
CNVDS
Онкоанализ
Sexdisaggregate
Скринет
Deviscomp
Covid19canada
Integrationscore
Myomanager
snpcyp
VisualSarsdiff
Clustreval
Клостет
GSCVisualizer
Микрокомпет
Uniprotproteinview
Метилекспресс
Dynugene; рукопись
PDB3D
Strexpansionanalyzer
Celltypetool
Чипанализер
Структура упаковки показана ниже:
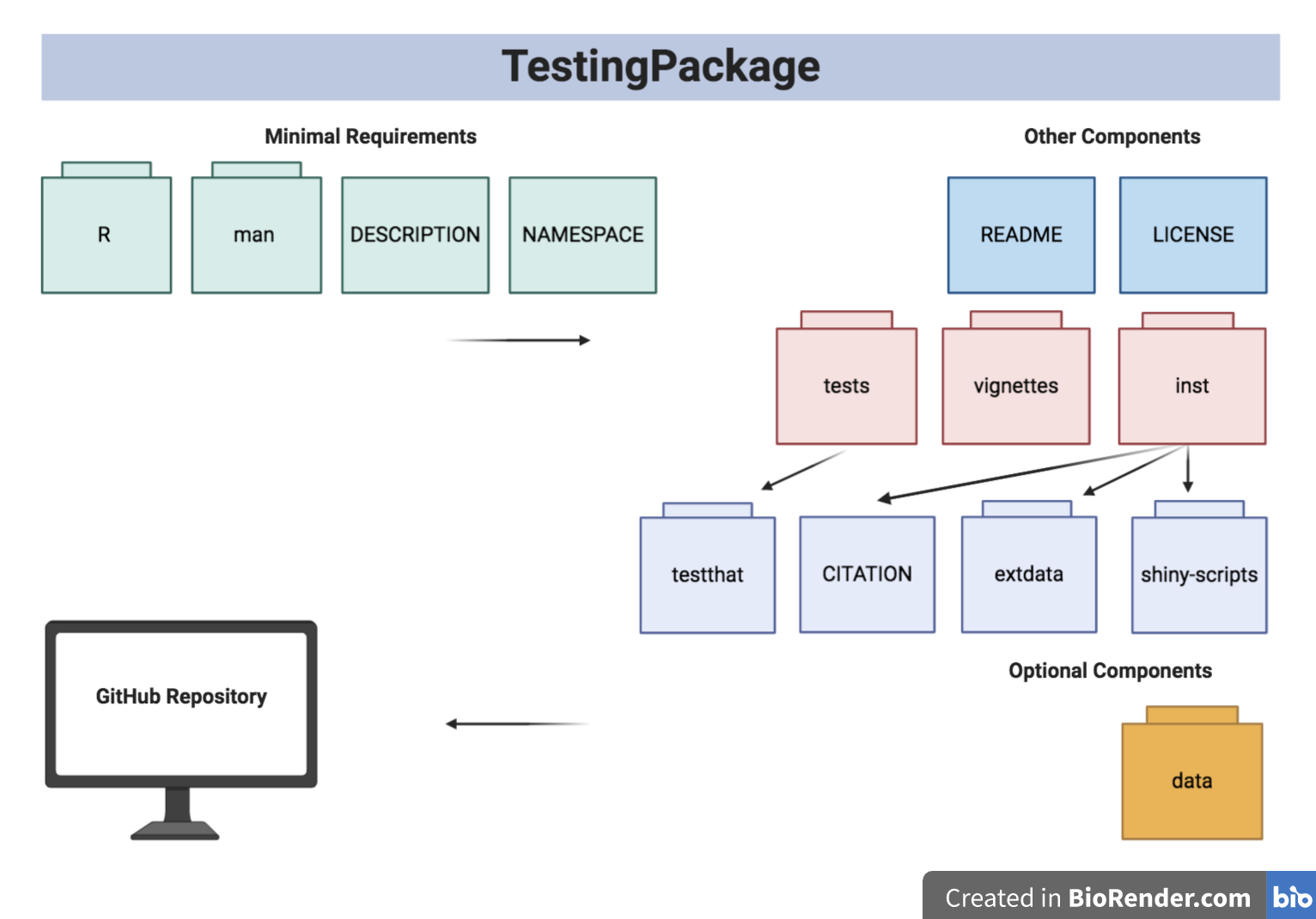
Структура дерева упаковки приведена ниже.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R