Ein Beispiel R -Paket für BCB410H: angewandte Bioinformatik.
Ein Absatz, der den Zweck Ihres R -Pakets und den analysierten biologischen Daten beschreibt. Erklären Sie, wie Ihr Paket einen aktuellen Arbeitsfluss in Bioinformatik oder Computerbiologie hinzufügt (dh es ist einzigartig?, Welches Problem geht es an?). Schließlich finden Sie die R -Version (nicht die RSTUDIO -Version) und die Plattform (Mac, Windows, Linux (Debian, Fedora/Redhat, Ubuntu), die zur Entwicklung des Pakets verwendet wird. Sie können diese Informationen erhalten, indem Sie utils::sessionInfo() ausführen. Zu diesem Zeitpunkt sollte es keine Shiny Implementierung geben. Z.B,
TestingPackage ist ein R -Paket zum Nachweis von Komponenten eines einfachen R -Pakets mit RNA -Sequenzierungsdaten. Das R -Paket enthält die Hauptkomponenten: Beschreibung, Namespace, Man Subditionory und R -Unterverzeichnis. Zusätzlich werden Lizenz-, Readme- und Unterverzeichnisse Vignetten, Tests, Daten und Installationen untersucht. Das Paket richtet sich an BCB410H -Studenten (Applied Bioinformatics), die ein nützliches Instrument zur Analyse biologischer Daten im Format eines öffentlichen R -Pakets auf GitHub definieren sollen. Der Umfang des R -Pakets besteht darin, einen aktuellen Arbeitsfluss in Bioinformatik oder Computerbiologie zu erweitern oder zu verbessern. Das Tool sollte Funktionen enthalten, um die Analyse biologischer Daten durchzuführen und eine überzeugende grafische Leistung zu erzeugen, die idealerweise für die explorative Analyse unterstützt wird. Das TestingPackage Paket wurde unter Verwendung von R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) und Running under: macOS Ventura 13.2 .
Geben Sie den folgenden Text und die folgenden Befehle an, die an Ihr R -Paket angepasst wurden. Z.B,
So installieren Sie die neueste Version des Pakets:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )Um die glänzende App auszuführen:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Geben Sie die folgenden Befehle an, die an Ihr R -Paket angepasst wurden. Geben Sie dann eine Liste der von Benutzer zugänglichen Funktionen innerhalb des Pakets und eine kurze Beschreibung der einzelnen an. Fügen Sie ein Bild hinzu, das den Überblick über das Paket veranschaulicht, das die Eingänge und Ausgänge anzeigt. Stellen Sie sicher, dass das Bild am richtigen Ort abgelagert wird, wie im Unterricht erläutert. Zeigen Sie den Benutzer auf Vignetten für ein Tutorial Ihres Pakets. Z.B,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage enthält 3 Funktionen.
Infcriteriacalculation zur Berechnung von Informationskriterien angesichts der Datensatzdimensionen, der Log-Likelihood und der Wahrscheinlichkeit.
Normfaktoren zur Berechnung der Normalisierungsfaktoren über geschnittenes Mittelwert von M-Werten (TMM).
Infrizeriaplot zur Darstellung von Informationskriterienwerten als Streudiagramm.
Das Paket enthält auch zwei RNA -Sequenzierungsdatensätze, genannt GeneCounts und GeneCounts2. Weitere Informationen finden Sie in Paketvignetten. Eine Übersicht über das Paket ist unten dargestellt.
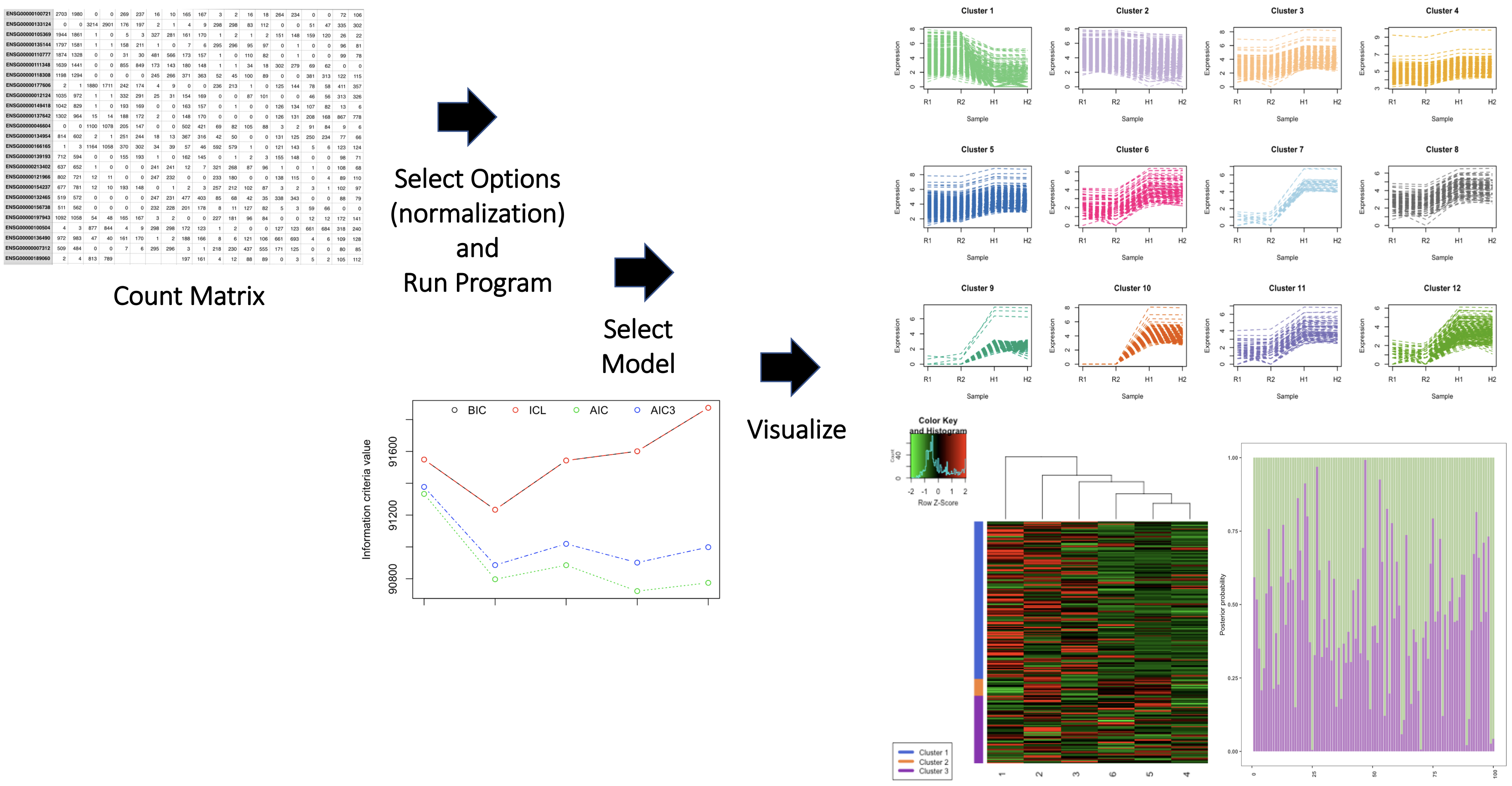
Geben Sie einen Absatz an, der deutlich den Namen des Autors des Pakets und die Beiträge des Autors angibt. Übermitteln Sie die Beiträge aus anderen Paketen/Quellen für jede Funktion. Übernehmen Sie die Beiträge aus generativen KI -Tools für jede Funktion. Geben Sie an, wie die Tools verwendet wurden und wie die Ergebnisse der KI -Tools eingebaut wurden. Denken Sie daran, dass Ihre individuellen Beiträge zum Paket wichtig sind. Z.B,
Der Autor des Pakets ist Anjali Silva. Der Autor schrieb die Function Function Infrizeriacalculationsfunktion , mit der die Datenspezifikationen der Informationskriterien berechnet werden. Hier werden das Bayesian Information Criterion (BIC), das Akaike Information Criterion (AIC) und die integrierte vollständige Wahrscheinlichkeit (ICL) berechnet. Die Infrizerikalkulationsfunktion verwendet die Kartenfunktion aus mclust R -Paket, um Informationen zu Informationen zu generieren. Das stats -R -Paket wird zur Erzeugung multinomial verteilter Zufallszahlvektoren verwendet. Ein Teil des Codes für die Funktion "Infrizeriacalculationsfunktion " wurde aus <NamePackage> R -Paket entnommen. (Der Abschnitt des geliehenen Codes sollte klar angegeben und in der Infrizerikalkulation R -Skript verwiesen werden). Der Infrizeriaplot wurde vom Autor verfasst und generiert ein Diagramm von Informationskriterienwerten. Die Infrizeriaplotfunktion nutzt das graphics -R -Paket. Normfaktoren sind eine Funktion, die Normalisierungsfaktoren über den Mittelwert von M-Werten (TMM) berechnet. Die Normfactors -Funktion verwendet mit dem im edgeR R-Paket implementierter M-Values (TMM) implementiertes Mittelwert von M-Werten (TMM). Bei der Entwicklung dieses Pakets wurden keine generativen KI -Tools verwendet.
Geben Sie in einem Format vollständige Referenzen für alle verwendeten Quellen, einschließlich für die Pakete und Tools, in einem Format an. Z.B,
Akaike, H. (1973). Informationstheorie und eine Erweiterung des Prinzips der maximalen Wahrscheinlichkeit. In Second International Symposium on Information Theory , New York, USA, 267–281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux und G. Govaert (2000). Bewertung eines Mischungsmodells zum Clustering mit der integrierten Klassifizierungswahrscheinlichkeit. IEEE-Transaktionen zur Musteranalyse und Maschineninformation 22. https://hal.inria.fr/inria-00073163/document
Biorender. (2020). Bild erstellt von Silva, A. Abgerufen am 30. Oktober 2020 von https://app.biorender.com/
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023). Shiny: Web -Anwendungsrahmen für r . R-Paketversion 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen und GK Smyth (2012). Differentielle Expressionsanalyse von Multifaktor-RNA-Seq-Experimenten in Bezug auf biologische Variation. Nukleinsäuren Forschung 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R Core Team (2023). R: Eine Sprache und Umgebung für statistisches Computer. R Foundation für statistisches Computing, Wien, Österreich. https://www.r-project.org/
Robinson, MD, DJ McCarthy und GK Smyth (2010). EDGER: Ein Biokonduktorpaket für die differentielle Expressionsanalyse digitaler Genexpressionsdaten. Bioinformatik 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). Schätzung der Dimension eines Modells. Die Annalen der Statistik 6, 461–464. https://projecteuclid.org/euclid.aos/1176344136.
Scrucca, L., M. FOP, TB Murphy und AE Raftery (2016) MCLUST 5: Clustering, Klassifizierung und Dichteschätzung unter Verwendung von Gaußschen Finite -Mischungs -Modellen. Das R Journal 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. Ggplot2: Elegante Grafiken für die Datenanalyse. Springer-Verlag New York, 2016.
Wickham, H. und J. Bryan (2019). R -Pakete (2. Auflage). Newton, Massachusetts: O'Reilly Media. https://r-pkgs.org/
Geben Sie den folgenden Text an, der an Ihr R -Paket angepasst wurde. Z.B,
Dieses Paket wurde im Rahmen einer Bewertung für 2019-2023 BCB410H entwickelt: Angewandter Bioinformatikkurs an der University of Toronto, Toronto, Kanada. TestingPackage begrüßt Probleme, Verbesserungsanfragen und andere Beiträge. Um ein Problem einzureichen, verwenden Sie die Github -Probleme. Vielen Dank an diejenigen, die Feedback zur Verbesserung dieses Pakets gegeben haben.
Pakete, die von BCB410 -Alumni entwickelt wurden. Vielen Dank an diejenigen, die die Erlaubnis erteilten, ihre Pakete zu teilen!
Atlaz
ClinicalTranscriptLink
Kompheatmaps
Covidwastewatch
Drugsekr
Geanaly
Integratrn
Isoformvisrna
Microbiomexplorer
Missensepathor
OpenVirome
Osteoanalizer
Pdbcleanup
Phenogenrlib
Prostat5
psychcnvassoc
Seurattogo
TmscoreAlign
Trexdad
Trnvalstandvis
Abunrna
Deregenes
CNVDs
Onkoanalyse
sexdisaggregate
scrgnet
Deviscomp
Covid19Canada
IntegrationScore
Myomanager
SNPCYP
Visualsarsdiff
Clustreval
Clustphy
gscvisualizer
Mikrozompet
UniProtProteinview
Methylexpress
Dynugene; Manuskript
pdb3d
StrexpansionAnalyzer
Cellypetool
Chipanalyzer
Die Paketstruktur ist unten dargestellt:
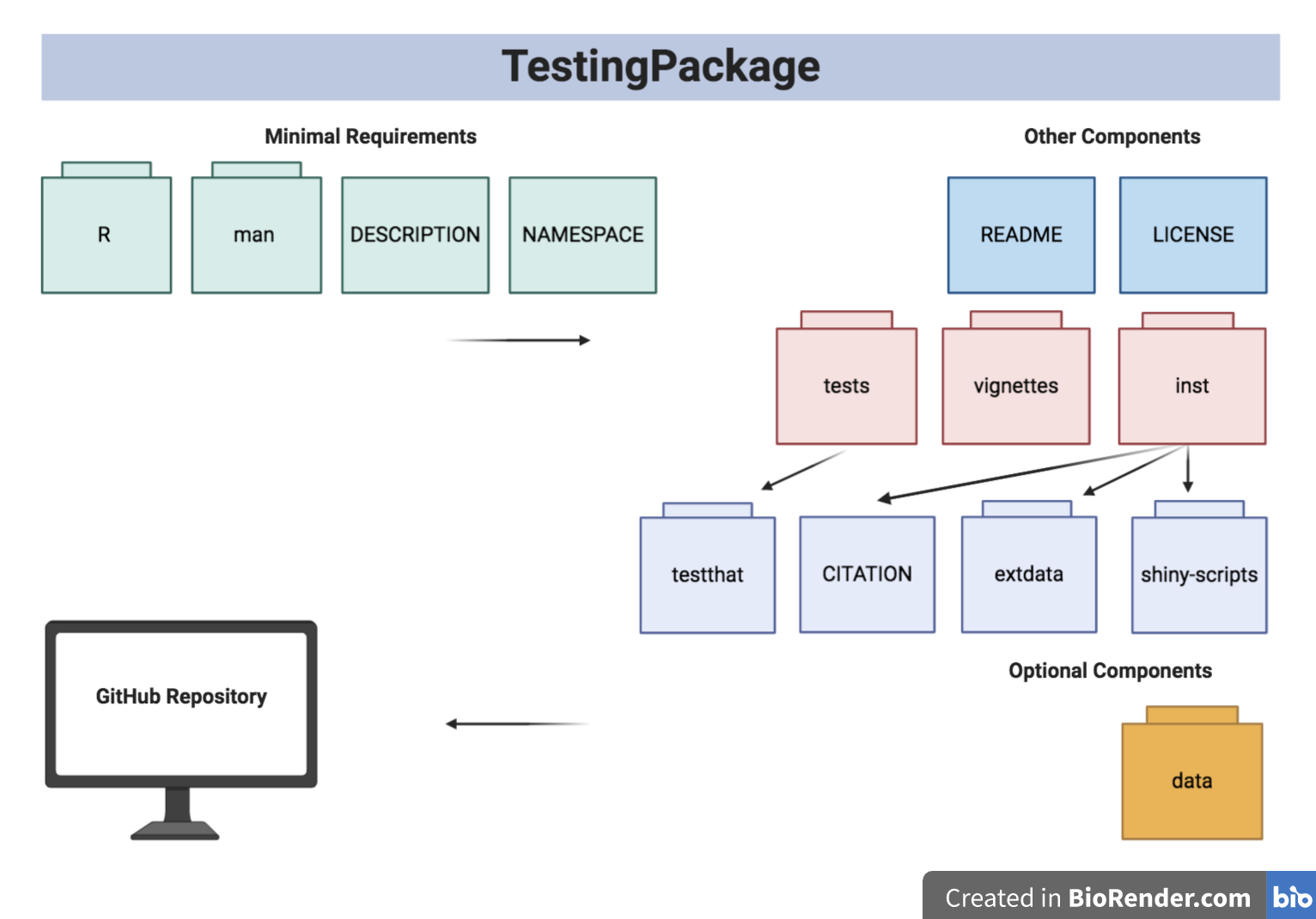
Die Packungsbaumstruktur ist unten bereitgestellt.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R