BCB410Hの例Rパッケージ:Applied Bioinformatics。
Rパッケージと分析対象の生物学的データの目的を説明する段落。パッケージは、バイオインフォマティクスまたは計算生物学の現在のワークフローをどのように追加または改善するかを説明します(つまり、それはどのようにユニークですか?、どのような問題に対処しますか?)。最後に、パッケージの開発に使用するために使用されるRバージョン(RSTUDIOバージョンではなく)とプラットフォーム(Mac、Windows、Linux(Debian、Fedora/Redhat、Ubuntu))を含めます。 utils::sessionInfo()を実行して、この情報を取得できます。この時点では、 Shiny実装はないはずです。例えば、
TestingPackage 、RNAシーケンスデータを備えた単純なRパッケージのコンポーネントを示すRパッケージです。 Rパッケージには、メインコンポーネントが含まれています:説明、名前空間、MANサブディレクトリ、Rサブディレクトリ。さらに、ライセンス、README、およびサブディレクトリのビネット、テスト、データ、INSTも検討されています。このパッケージは、GitHubに収容されているパブリックRパッケージの形式で生物学的データを分析するための有用なツールを定義するBCB410H(Applied Bioinformatics)の学生を対象としています。 Rパッケージの範囲は、バイオインフォマティクスまたは計算生物学の現在のワークフローを追加または改善することです。このツールには、生物学的データの分析を実行し、説得力のあるグラフィカルな出力を生成する機能を含める必要があります。理想的には、探索的分析をサポートするためです。 TestingPackageパッケージは、 R version 4.2.1 (2022-06-23) 、 Platform: x86_64-apple-darwin17.0 (64-bit) 、およびRunning under: macOS Ventura 13.2されました。
Rパッケージに合わせてカスタマイズされた次のテキストとコマンドを提供します。例えば、
パッケージの最新バージョンをインストールするには:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )光沢のあるアプリを実行するには:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Rパッケージに合わせてカスタマイズされた次のコマンドを提供します。次に、パッケージ内のユーザーアクセス可能な機能のリストと、それぞれの簡単な説明を提供します。入力と出力を示すパッケージの概要を示す1つの画像を含めます。クラスで説明されているように、画像が正しい場所に堆積していることを確認してください。ユーザーにパッケージのチュートリアルを求めてビネットに向けます。例えば、
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackageには3つの機能が含まれています。
データセットの寸法、対数尤度、および確率を与えられた情報基準を計算するためのINFCRIRTERIACALCULATION 。
m値(TMM)のトリミングされた平均を介して正規化係数を計算するための正規ファクトル。
情報基準を散布図としてプロットするためのinfcriteriaplot 。
パッケージには、GeneCountsとGeneCounts2と呼ばれる2つのRNAシーケンスデータセットも含まれています。詳細については、パッケージビネットを参照してください。パッケージの概要を以下に示します。
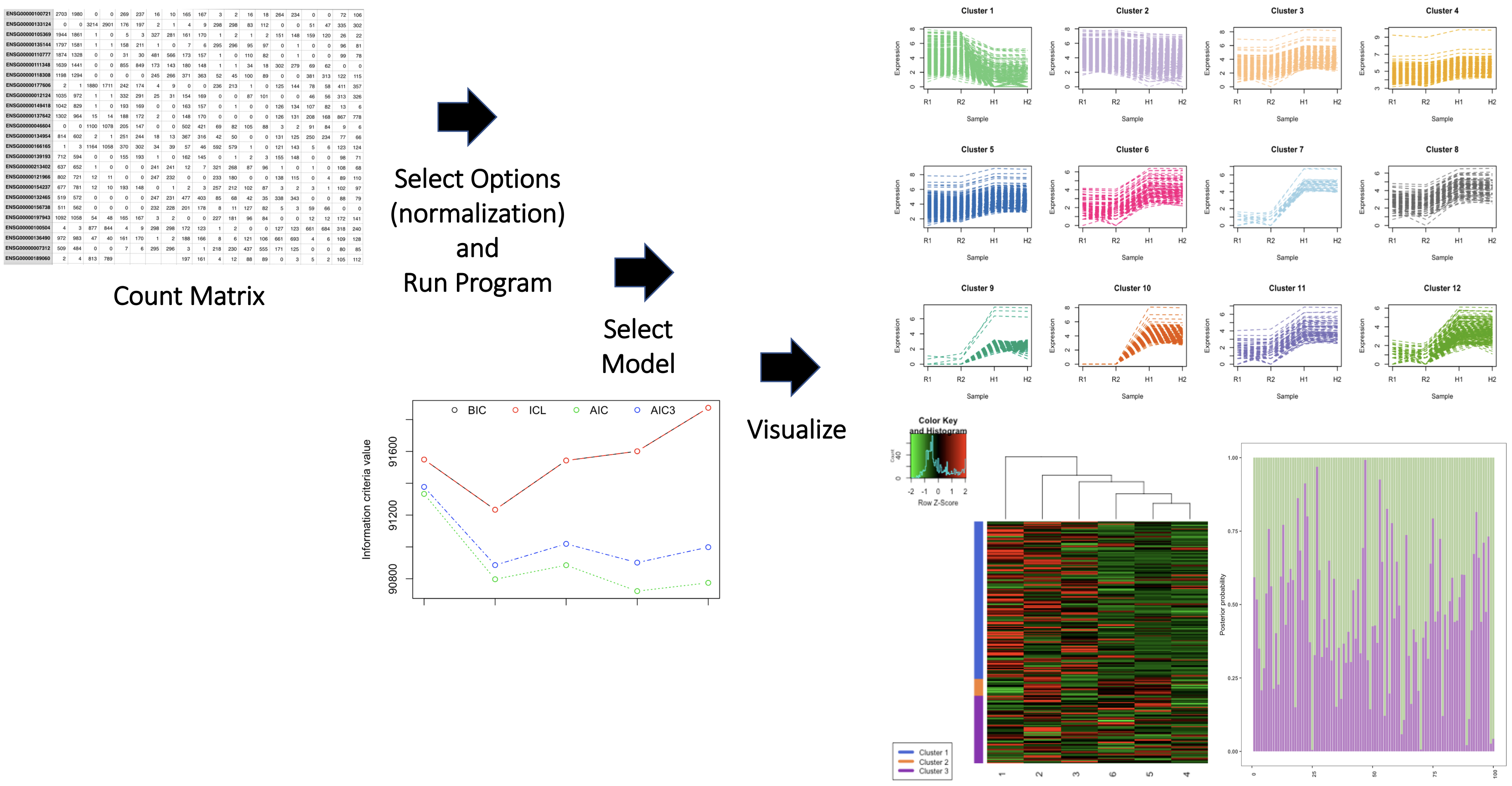
パッケージの著者の名前と著者からの貢献を明確に示す段落を提供します。各関数の他のパッケージ/ソースからの概要。各関数の生成AIツールからの概要。ツールの使用方法と、AIツールの結果がどのように組み込まれたかを含めます。パッケージへの個々の貢献が重要であることを忘れないでください。例えば、
パッケージの著者はAnjali Silvaです。著者は、データ仕様の指定された情報基準値を計算するInfcriteriacalCulation関数を書きました。ここでは、ベイジアン情報基準(BIC)、別名情報基準(AIC)、統合完全尤度(ICL)が計算されます。 INFCriteriacAlculation関数は、 mclust RパッケージのMAP関数を使用して、情報基準値を生成します。 stats Rパッケージは、多数分布の乱数ベクトルを生成するために使用されます。 InfcriteriacalCulation関数のコードの一部は、 <NamePackage> Rパッケージから取得されています。 (借りたコードのセクションは、infcriteriacalculation rスクリプトで明確に示され、参照する必要があります)。 INFCriteriaplotは著者によって書かれており、情報基準値のプロットを生成します。 Infcriteriaplot関数は、 graphics Rパッケージを使用します。 Normfactorsは、m値(TMM)のトリミングされた平均を介して正規化係数を計算する関数です。 Normfactors関数は、 edgeR Rパッケージに実装されているように、m値(TMM)のトリミングされた平均を使用します。このパッケージの開発には、生成的AIツールは使用されませんでした。
「寄付」に記載されているパッケージやツールを含む、使用されるすべてのソースに1つの形式で完全な参照を提供します。例えば、
アカケ、H。(1973)。情報理論と最尤原則の拡張。米国ニューヨーク州ニューヨークの情報理論に関する第2回国際シンポジウム、267–281。 Springer Verlag。 https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15。
Biernacki、C.、G。Celeux、およびG. Govaert(2000)。統合された分類の可能性を備えたクラスタリングのための混合モデルの評価。パターン分析とマシンインテリジェンスに関するIEEEトランザクション22。https://hal.inria.fr/inria-00073163/document
バイオレンダー。 (2020)。 Silva、A。が作成した画像2020年10月30日、https://app.biorender.com/から取得
Chang、W.、J。Cheng、J。Allaire、C。Sievert、B。Schloerke、Y。Xie、J。Allen、J。Mcpherson、A。Dipert、B。Borges(2023)。光沢:RのWebアプリケーションフレームワーク。 Rパッケージバージョン1.8.0、https://cran.r-project.org/package = shiny
McCarthy、DJ、Y。Chenand Gk Smyth(2012)。生物学的変動に関する多因子RNA-seq実験の微分発現分析。核酸研究40。4288-4297。 https://pubmed.ncbi.nlm.nih.gov/22287627/
Rコアチーム(2023)。 R:統計コンピューティングの言語と環境。 R統計コンピューティングのための基礎、ウィーン、オーストリア。 https://www.r-project.org/
ロビンソン、MD、DJ McCarthyおよびGK Smyth(2010)。 Edger:デジタル遺伝子発現データの微分発現分析のためのバイオコンダクタパッケージ。バイオインフォマティクス26、139-140。 https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz、G。(1978)。モデルの次元の推定。統計6、461–464の年代記。 https://projecteuclid.org/euclid.aos/1176344136。
Scrucca、L.、M。Fop、TB Murphy and Ae Raftery(2016)Mclust 5:ガウス有限混合モデルを使用したクラスタリング、分類、密度推定。 Rジャーナル8(1)、289-317。 https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham、H。Ggplot2:データ分析のためのエレガントなグラフィックス。 Springer-Verlag New York、2016年。
ウィッカム、H。、J。ブライアン(2019)。 Rパッケージ(第2版)。マサチューセッツ州ニュートン:オライリーメディア。 https://r-pkgs.org/
Rパッケージに合わせてカスタマイズされた次のテキストを提供します。例えば、
このパッケージは、2019-2023 BCB410H:カナダのトロント大学トロント大学の応用バイオインフォマティクスコースの評価の一環として開発されました。 TestingPackage 、問題、強化要求、その他の貢献を歓迎します。問題を提出するには、GitHubの問題を使用します。このパッケージを改善するためにフィードバックを提供してくれた人に感謝します。
BCB410卒業生によって開発されたパッケージ。パッケージを共有する許可を提供してくれた人に感謝します!
アトラズ
ClinicalTranscriptlink
compheatmaps
covidwastewatch
Drugseekr
Geanaly
Integratrn
ISOFORMVISRNA
MicrobiomeExplorer
ミスセンスパター
OpenVirome
骨アナライザー
pdbcleanup
PhenogenRlib
ProStat5
Psychcnvassoc
Seurattogo
tmscorealign
Trexdad
trnvalstandvis
アバンナ
デレゲネス
CNVDS
オンコーナリシス
sexdisaggregate
scrgnet
Deviscomp
Covid19Canada
IntegrationScore
Myomanager
snpcyp
VisualSarsDiff
クラストラバル
Clustphy
gscvisualizer
マイクロコンペット
uniprotproteinview
メチレックスプレス
王朝;原稿
PDB3D
strexpansionanalyzer
CellTypetool
チパナリザー
パッケージ構造を以下に示します。
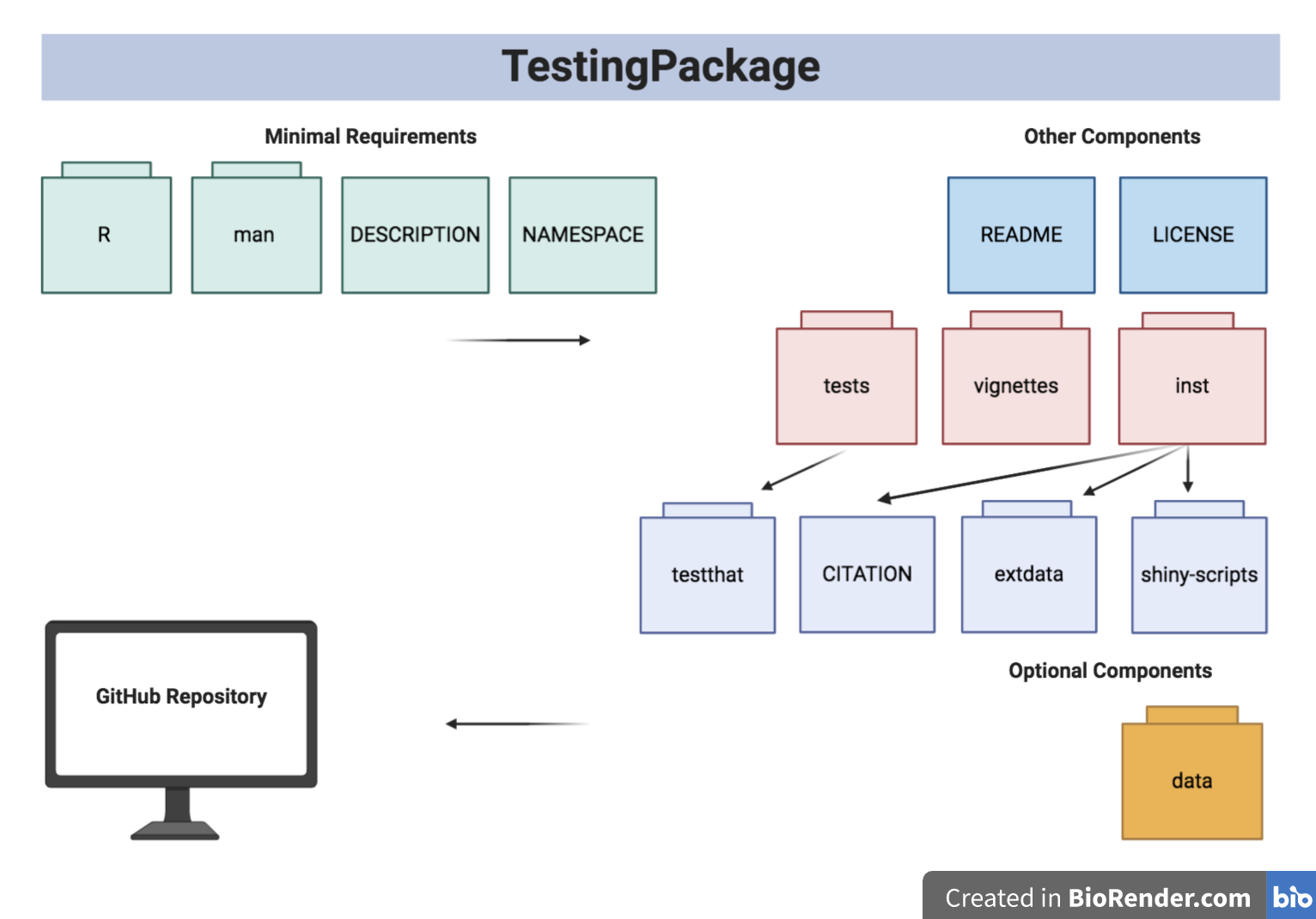
パッケージツリーの構造を以下に示します。
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R