Un paquete R de ejemplo R para BCB410H: Bioinformática aplicada.
Un párrafo que describe el propósito de su paquete R y datos biológicos que se analizan. Explique cómo su paquete se suma o mejora un flujo de trabajo actual en bioinformática o biología computacional (es decir, ¿cómo es único?, ¿Qué problema aborda?). Finalmente, incluya la versión R (no versión RStudio) y la plataforma (Mac, Windows, Linux (Debian, Fedora/Redhat, Ubuntu)), utilizada para desarrollar el paquete. Puede obtener esta información ejecutando utils::sessionInfo() . No debe haber una implementación Shiny en este momento. P.ej,
TestingPackage es un paquete R para demostrar componentes de un paquete R simple con datos de secuenciación de ARN. El paquete R incluye los componentes principales: descripción, espacio de nombres, subdirectorio de hombre y subdirectorio R. Además, también se exploran las viñetas, pruebas, datos e Inst de licencia, lectura y subdirectorios. El paquete está dirigido a los estudiantes BCB410H (Bioinformática Aplicada), que deben definir una herramienta útil para el análisis de datos biológicos en el formato de un paquete P público alojado en Github. El alcance del paquete R es agregar o mejorar un flujo de trabajo actual en bioinformática o biología computacional. La herramienta debe contener funciones para realizar el análisis de datos biológicos y producir una salida gráfica convincente, idealmente para admitir el análisis exploratorio. El paquete TestingPackage se desarrolló utilizando R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) y Running under: macOS Ventura 13.2 .
Proporcione el siguiente texto y comandos, personalizado en su paquete R. P.ej,
Para instalar la última versión del paquete:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )Para ejecutar la aplicación brillante:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Proporcione los siguientes comandos, personalizados en su paquete R. Luego proporcione una lista de funciones accesibles para el usuario dentro del paquete y una breve descripción de cada una. Incluya una imagen que ilustra la descripción general del paquete que muestra las entradas y salidas. Asegúrese de que la imagen se deposite en la ubicación correcta, como se discutió en la clase. Apunte al usuario a Vignettes para obtener un tutorial de su paquete. P.ej,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage contiene 3 funciones.
Infcriteriacalcule para calcular los criterios de información dadas las dimensiones del conjunto de datos, la verificación log y la probabilidad.
Normfactores para calcular los factores de normalización a través de la media recortada de los valores M (TMM).
InfcriteriAllot para trazar los valores de los criterios de información como una gráfica de dispersión.
El paquete también contiene dos conjuntos de datos de secuenciación de ARN, llamados Genecounts y Genecounts2. Consulte las viñetas de paquete para obtener más detalles. A continuación se ilustra una descripción general del paquete.
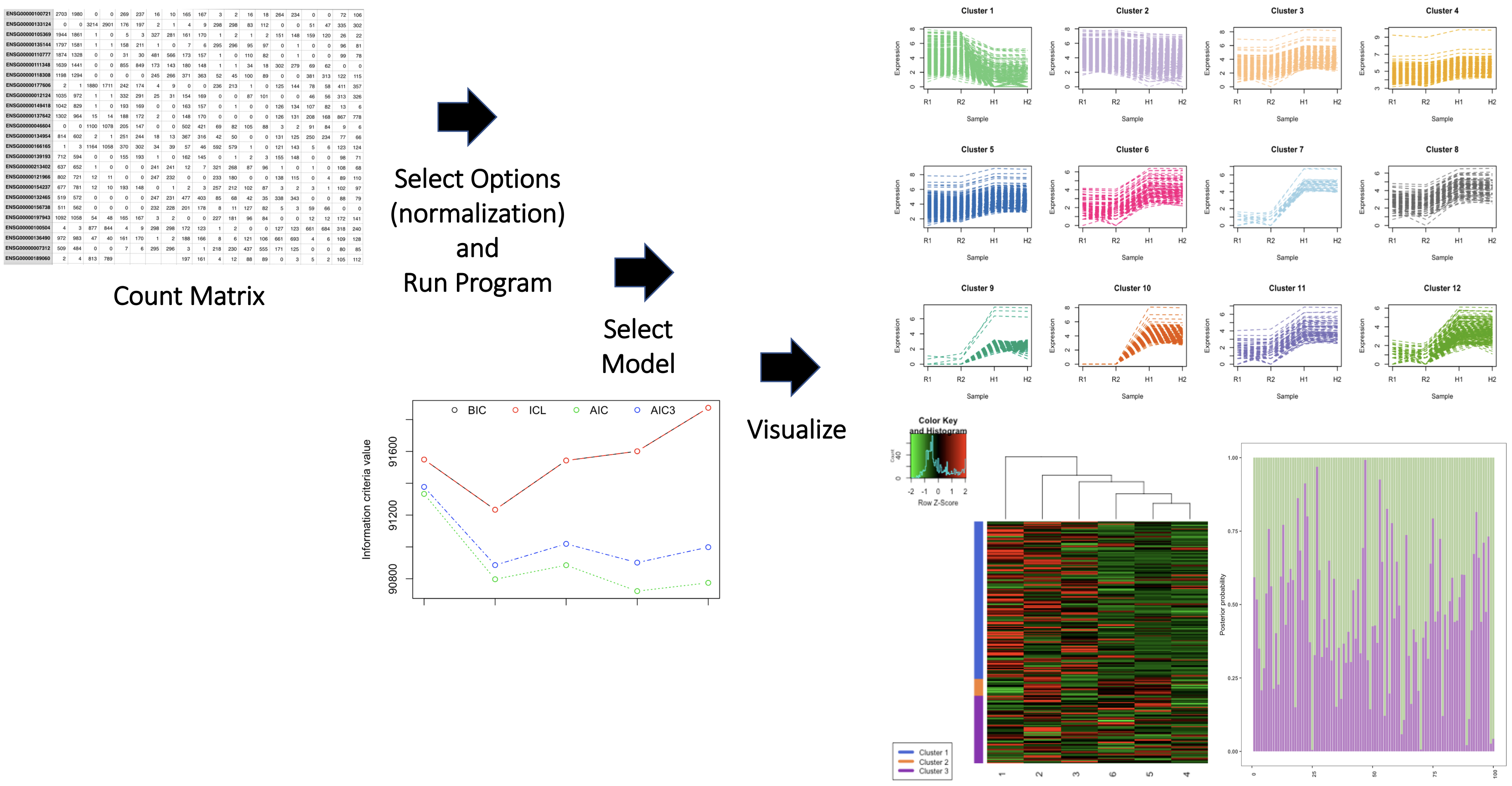
Proporcione un párrafo que indique claramente el nombre del autor del paquete y las contribuciones del autor. Esbozar las contribuciones de otros paquetes/fuentes para cada función. Cumplir las contribuciones de las herramientas de IA generativas para cada función. Incluya cómo se usaron las herramientas y cómo se incorporaron los resultados de las herramientas de IA. Recuerde que sus contribuciones individuales al paquete son importantes. P.ej,
El autor del paquete es Anjali Silva. El autor escribió la función Infcriteriacalculation , que calcula los valores de los criterios de información dadas las especificaciones de datos. Aquí, se calculan el criterio de información bayesiano (BIC), el criterio de información de Akaike (AIC) y la probabilidad completa integrada (ICL). La función InfcrIteriacalcation utiliza la función MAP del paquete mclust R para generar valores de criterios de información. El paquete stats R se utiliza para generar vectores de números aleatorios distribuidos multinomialmente. Se ha tomado parte del código para la función de infriteriacalculación del paquete <NamePackage> R. (La sección del código prestado debe estar claramente indicada y referenciada en el script infcriteriacalculation R). El autor de InfCriteriAl es escrito por el autor y genera un gráfico de valores de criterios de información. La función InfCriterIAPLOT utiliza el paquete graphics R. NormFactors es una función que calcula los factores de normalización a través de la media recortada de los valores M (TMM). La función NormFactors utiliza la media recortada de los valores M (TMM) como se implementa en el paquete edgeR R. No se utilizaron herramientas generativas de IA en el desarrollo de este paquete.
Proporcione referencias completas para todas las fuentes utilizadas, incluso para los paquetes y herramientas mencionadas en 'Contribuciones', en un solo formato. P.ej,
Akaike, H. (1973). Teoría de la información y una extensión del principio de máxima probabilidad. En el segundo Simposio Internacional sobre Teoría de la Información , Nueva York, EE. UU., 267–281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux y G. Govaert (2000). Evaluar un modelo de mezcla para la agrupación con la probabilidad de clasificación integrada. Transacciones IEEE sobre análisis de patrones e inteligencia de máquinas 22. https://hal.inria.fr/inria-00073163/document
Biorrender. (2020). Imagen creada por Silva, A. Recuperado el 30 de octubre de 2020, a partir de https://app.biorender.com/
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023). Shiny: Marco de aplicaciones web para r . R PAGACE Versión 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen y Gk Smyth (2012). Análisis de expresión diferencial de experimentos multifactoriales de ARN-seq con respecto a la variación biológica. Nucleic Acids Research 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R EQUIPO CORE (2023). R: Un lenguaje y un entorno para la computación estadística. R Fundación para la Computación Estadística, Viena, Austria. https://www.r-project.org/
Robinson, MD, DJ McCarthy y GK Smyth (2010). Edger: un paquete de bioconductores para análisis de expresión diferencial de datos de expresión génica digital. Bioinformática 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). Estimación de la dimensión de un modelo. The Annals of Statistics 6, 461–464. https://procheuclid.org/euclid.aos/1176344136.
Scrucca, L., M. Fop, TB Murphy y AE Raftery (2016) McLust 5: Estimación de agrupación, clasificación y densidad utilizando modelos de mezcla finitas gaussianas. El R Journal 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. GGPlot2: gráficos elegantes para el análisis de datos. Springer-Verlag Nueva York, 2016.
Wickham, H. y J. Bryan (2019). R paquetes (2ª edición). Newton, Massachusetts: O'Reilly Media. https://r-pkgs.org/
Proporcione el siguiente texto, personalizado a su paquete R. P.ej,
Este paquete se desarrolló como parte de una evaluación para 2019-2023 BCB410H: curso de bioinformática aplicada en la Universidad de Toronto, Toronto, Canadá. TestingPackage da la bienvenida a problemas, solicitudes de mejora y otras contribuciones. Para enviar un problema, use los problemas de GitHub. Muchas gracias a aquellos que proporcionaron comentarios para mejorar este paquete.
Paquetes desarrollados por ex alumnos de BCB410. ¡Muchas gracias a aquellos que proporcionaron permiso para compartir sus paquetes!
Parto
Clinicaltranscriptlink
CompHeatMaps
Covidwwewatch
Drogadizo
Regio
Integrar
Isoformvisrna
Microbiomexplorer
Maldito
Viromo abierto
Osteoanalizador
Pdbcleanup
Fenógeno
Prostat5
psiccnvassoc
Seurattogo
Tmscorealign
Trexdad
Trnvalstandvis
Abunrna
Desagregenes
CNVDS
oncoanálisis
Sexdisaggregate
scrgnet
Perseguir
covid19canada
Integrationscore
Myomanerer
snpcyp
Visualsarsdiff
cluscar
Clustphy
gscvisualizador
microcompet
uniprotproteinview
Metylexpress
Dynugene; manuscrito
pdb3d
Stractuguanalizador
Typetool de celda
Chipanalzer
La estructura del paquete se ilustra a continuación:
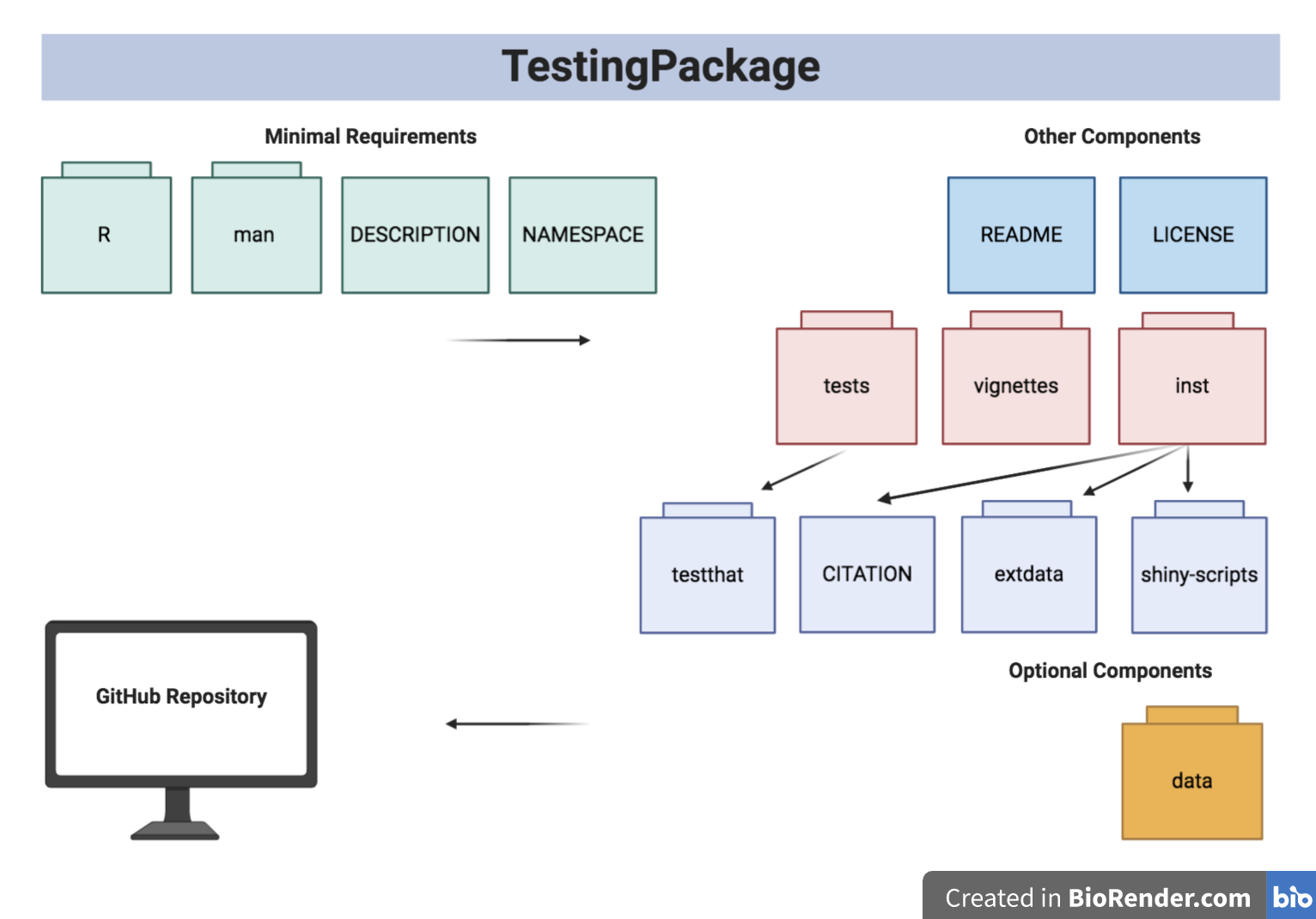
La estructura del árbol del paquete se proporciona a continuación.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R