حزمة مثال R لـ BCB410H: المعلوماتية الحيوية التطبيقية.
فقرة تصف الغرض من حزمة R وبياناتك البيولوجية التي يتم تحليلها. اشرح كيف تضيف الحزمة الخاصة بك إلى تدفق العمل الحالي في المعلوماتية الحيوية أو البيولوجيا الحسابية (أي كيف هي فريدة من نوعها؟ ، ما هي المشكلة التي تتناولها؟). أخيرًا ، قم بتضمين إصدار R (وليس إصدار Rstudio) والمنصة (Mac ، Windows ، Linux (Debian ، Fedora/Redhat ، Ubuntu)) ، المستخدمة لتطوير الحزمة. يمكنك الحصول على هذه المعلومات عن طريق تشغيل utils::sessionInfo() . يجب ألا يكون هناك تنفيذ Shiny في هذه المرحلة. على سبيل المثال ،
TestingPackage هي حزمة R لإظهار مكونات حزمة R بسيطة مع بيانات تسلسل RNA. تتضمن حزمة R المكونات الرئيسية: الوصف ، مساحة الاسم ، دليل الرجل الفرعي والدليل الفرعي R. بالإضافة إلى ذلك ، يتم أيضًا استكشاف الترخيص والإسغليين والمقالات الفرعية والاختبارات والبيانات و Inst. يتم استهداف الحزمة للطلاب BCB410H (المعلوماتية الحيوية التطبيقية) ، الذين يحددون أداة مفيدة لتحليل البيانات البيولوجية بتنسيق حزمة R العامة الموجودة على GitHub. يتمثل نطاق حزمة R في إضافة أو تحسين تدفق العمل الحالي في المعلوماتية الحيوية أو البيولوجيا الحسابية. يجب أن تحتوي الأداة على وظائف لإجراء تحليل للبيانات البيولوجية وإنتاج إخراج رسومي مقنع ، من الناحية المثالية لدعم التحليل الاستكشافي. تم تطوير حزمة TestingPackage باستخدام R version 4.2.1 (2022-06-23) ، Platform: x86_64-apple-darwin17.0 (64-bit) Running under: macOS Ventura 13.2 .
قدم النص والأوامر التالية ، مخصصة لحزمة R الخاصة بك. على سبيل المثال ،
لتثبيت أحدث إصدار من الحزمة:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )لتشغيل التطبيق اللامع:
runTestingPackage() # not for Assessment 4; only for Assessment 5 قدم الأوامر التالية ، مخصصة إلى حزمة R الخاصة بك. ثم قدم قائمة بالوظائف التي يمكن الوصول إليها من قبل المستخدم داخل الحزمة ووصف موجز لكل منها. قم بتضمين صورة واحدة توضح نظرة عامة على الحزمة التي توضح المدخلات والمخرجات. تأكد من إيداع الصورة في الموقع الصحيح ، كما تمت مناقشته في الفصل. أشر المستخدم إلى المقالات القصيرة للحصول على برنامج تعليمي للحزمة الخاصة بك. على سبيل المثال ،
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage يحتوي على 3 وظائف.
infcriteriacalculation لحساب معايير المعلومات المعطاة أبعاد مجموعة البيانات ، احتمالية السجل والاحتمال.
Normfactors لحساب عوامل التطبيع عن طريق المتوسط من قيم M المقلدة (TMM).
infcriteriapplot للتخطيط لمعايير المعلومات قيم كآمنة مبعثرة.
تحتوي الحزمة أيضًا على مجموعات بيانات تسلسل الحمض النووي الريبي ، تسمى GeneCounts و GeneCounts2. الرجوع إلى الحزمة المقالات القصيرة لمزيد من التفاصيل. يتم توضيح نظرة عامة على الحزمة أدناه.
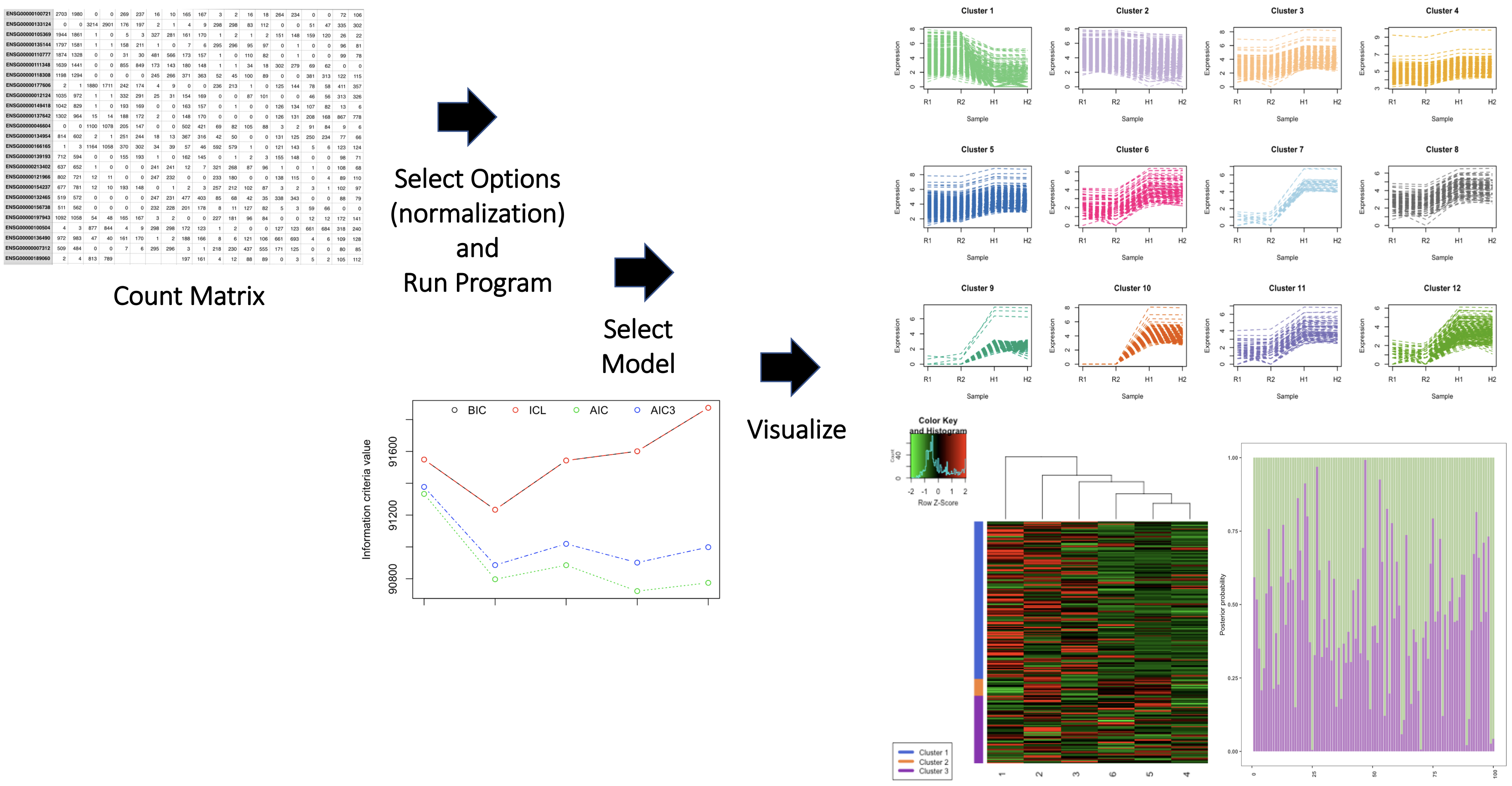
توفير فقرة تشير بوضوح إلى اسم مؤلف الحزمة والمساهمات من المؤلف. الخطوط العريضة للمساهمات من الحزم/المصادر الأخرى لكل وظيفة. تسديد المساهمات من أدوات (أدوات) الذكاء الاصطناعي لكل وظيفة. قم بتضمين كيفية استخدام الأدوات وكيف تم دمج نتائج أدوات الذكاء الاصطناعي. تذكر أن مساهماتك الفردية في الحزمة مهمة. على سبيل المثال ،
مؤلف الحزمة هو أنجالي سيلفا. كتب المؤلف وظيفة infcriteriacalculation ، التي تحسب قيم معايير المعلومات المعطاة مواصفات البيانات. هنا ، يتم حساب معيار المعلومات Bayesian (BIC) ومعيار معلومات Akaike (AIC) والاحتمالية الكاملة المتكاملة (ICL). تستخدم وظيفة infcriteriacalculation وظيفة الخريطة من حزمة mclust R لإنشاء قيم معايير المعلومات. يتم استخدام حزمة stats R لإنشاء متجهات رقم عشوائية موزعة متعددة الموزع. تم أخذ جزء من رمز وظيفة infcriteriacalculation من حزمة <NamePackage> r. (يجب الإشارة بوضوح إلى قسم الكود المقترض والرجوع إليه في البرنامج النصي R infcriteriacalculation). يتم كتابة infcriteriapplot من قبل المؤلف ويولد مؤامرة من قيم معايير المعلومات. تستخدم وظيفة infcriteriapplot حزمة graphics R. NormFactors هي وظيفة تحسب عوامل التطبيع عبر متوسط القيم M (TMM). تستخدم وظيفة NormFactors متوسط متوسط القيم M (TMM) كما هو الحال في حزمة edgeR R. لم يتم استخدام أدوات الذكاء الاصطناعي التوليدي في تطوير هذه الحزمة.
قم بتوفير مراجع كاملة لجميع المصادر المستخدمة ، بما في ذلك الحزم والأدوات المذكورة تحت "المساهمات" ، بتنسيق واحد. على سبيل المثال ،
Akaike ، H. (1973). نظرية المعلومات وتمديد مبدأ احتمال الحد الأقصى. في الندوة الدولية الثانية حول نظرية المعلومات ، نيويورك ، الولايات المتحدة الأمريكية ، 267-281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki ، C. ، G. Celeux ، and G. Govaert (2000). تقييم نموذج الخليط للتجميع مع احتمال التصنيف المتكامل. معاملات IEEE على تحليل الأنماط وذكاء الآلة 22. https://hal.inria.fr/inria-00073163/document
Biorender. (2020). الصورة التي تم إنشاؤها بواسطة Silva ، A. Retrieved في 30 أكتوبر 2020 ، من https://app.biorender.com/
تشانغ ، و. لامعة: إطار تطبيق الويب لـ R. R حزمة الإصدار 1.8.0 ، https://cran.r-project.org/package=shiny
McCarthy ، DJ ، Y. Chen and GK Smyth (2012). تحليل التعبير التفاضلي لتجارب RNA-Seq متعددة العوامل فيما يتعلق بالتغير البيولوجي. أبحاث الأحماض النووية 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R Core Team (2023). R: لغة وبيئة للحوسبة الإحصائية. R Foundation للحوسبة الإحصائية ، فيينا ، النمسا. https://www.r-project.org/
Robinson ، MD ، DJ McCarthy and GK Smyth (2010). Edger: حزمة الموصل الحيوي لتحليل التعبير التفاضلي لبيانات التعبير الجيني الرقمي. المعلوماتية الحيوية 26 ، 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
شوارز ، ج. (1978). تقدير بُعد النموذج. حوليات الإحصاء 6 ، 461-464. https://projecteuclid.org/euclid.aos/1176344136.
Scrucca ، L. ، M. FOP ، TB Murphy and Ae Raftery (2016) McLust 5: التجميع ، التصنيف وتقدير الكثافة باستخدام نماذج الخليط المحدودة Gaussian. The R Journal 8 (1) ، 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham ، H. Ggplot2: رسومات أنيقة لتحليل البيانات. Springer-Verlag New York ، 2016.
Wickham ، H. and J. Bryan (2019). حزم ص (الطبعة الثانية). نيوتن ، ماساتشوستس: O'Reilly Media. https://r-pkgs.org/
قدم النص التالي ، مخصص لحزمة R الخاصة بك. على سبيل المثال ،
تم تطوير هذه الحزمة كجزء من تقييم لعام 2019-2023 BCB410H: دورة المعلوماتية الحيوية التطبيقية في جامعة تورنتو ، تورونتو ، كندا. ترحب TestingPackage بالمشكلات وطلبات التحسين والمساهمات الأخرى. لتقديم مشكلة ، استخدم مشكلات GitHub. شكرا جزيلا لأولئك الذين قدموا ملاحظات لتحسين هذه الحزمة.
الحزم التي طورها خريجي BCB410. شكرا جزيلا لأولئك الذين قدموا الإذن لمشاركة حزمهم!
أتلز
ClinicalTranscriptLink
compheatmaps
Covidwastewatch
المخدرات
Geanaly
Integratrn
isoformvisrna
microbiomeexplorer
missensepathor
OpenVirome
عظمية
PDBCleanup
Phenogenrlib
prostat5
Psychcnvassoc
Seurattogo
tmscorealign
Trexdad
trnvalstandvis
abunrna
Dereggenes
CNVDs
oncoanalysis
sexdisaggregate
scrgnet
Deviscomp
Covid19canada
IntegrationsCore
Myomanager
SNPCYP
VisualSarsdiff
الكسور
clustphy
gscvisualizer
microcompet
uniprotproteinview
methylexpress
Dynugene مخطوطة
PDB3D
strexpansionananalzer
celltypetool
Chipanalyzer
تم توضيح بنية الحزمة أدناه:
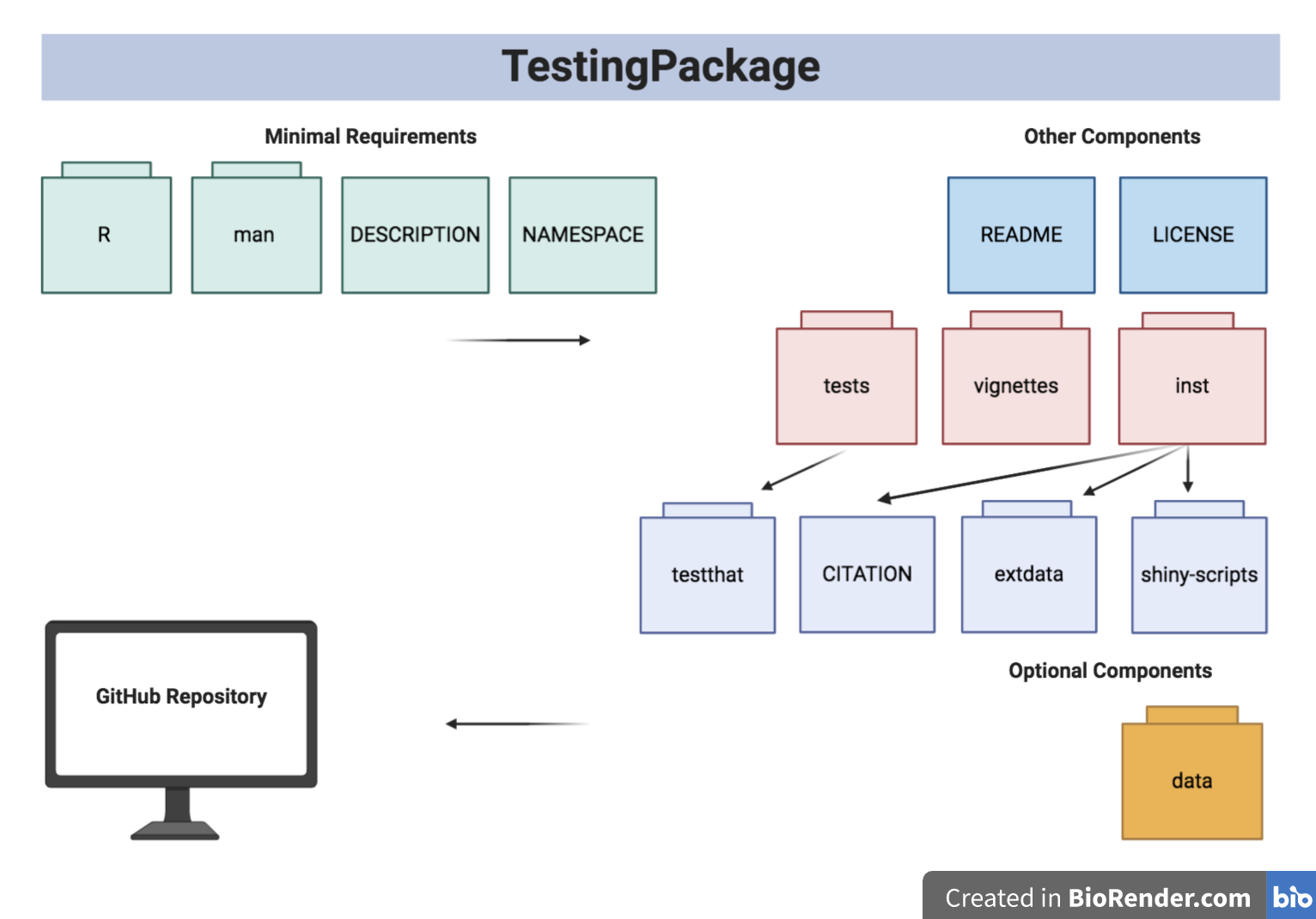
ويرد بنية شجرة الحزمة أدناه.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R