Um exemplo de pacote R para BCB410H: Bioinformática aplicada.
Um parágrafo que descreve o objetivo do seu pacote R e dados biológicos sendo analisados. Explique como o seu pacote é adicionado ou melhorar um fluxo de trabalho atual em bioinformática ou biologia computacional (ou seja, como é único?, Que problema ele aborda?). Por fim, inclua a versão R (não a versão rstudio) e a plataforma (Mac, Windows, Linux (Debian, Fedora/Redhat, Ubuntu)), usada para desenvolver o pacote. Você pode obter essas informações executando utils::sessionInfo() . Não deve haver implementação Shiny neste momento. Por exemplo,
TestingPackage é um pacote R para demonstrar componentes de um pacote R simples com dados de sequenciamento de RNA. O pacote R inclui os principais componentes: descrição, espaço para nome, subdiretório MAN e subdiretório R. Além disso, também são exploradas vinhetas, testes, dados e instituições de leitura e subdiretos também são exploradas. O pacote é direcionado para os alunos do BCB410H (Applied Bioinformatics), que devem definir uma ferramenta útil para a análise de dados biológicos no formato de um pacote público r alojado no GitHub. O escopo do pacote R é adicionar ou melhorar um fluxo de trabalho atual em bioinformática ou biologia computacional. A ferramenta deve conter funções para executar a análise de dados biológicos e produzir uma saída gráfica atraente, idealmente para apoiar a análise exploratória. O pacote TestingPackage foi desenvolvido usando R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) e Running under: macOS Ventura 13.2 .
Forneça o seguinte texto e comandos, personalizado para o seu pacote R. Por exemplo,
Para instalar a versão mais recente do pacote:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )Para executar o aplicativo brilhante:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Forneça os seguintes comandos, personalizados para o seu pacote R. Em seguida, forneça uma lista de funções acessíveis ao usuário dentro do pacote e uma breve descrição de cada uma. Inclua uma imagem que ilustra a visão geral do pacote que mostra as entradas e saídas. Verifique se a imagem é depositada no local correto, conforme discutido na aula. Aponte o usuário às vinhetas para um tutorial do seu pacote. Por exemplo,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage contém 3 funções.
InfcriteriacalCulation para calcular os critérios de informação, dadas as dimensões do conjunto de dados, probabilidade de log e probabilidade.
Normactors para calcular fatores de normalização via média aparada de valores M (TMM).
Infeccriteria PloPt para plotar os valores dos critérios de informação como um gráfico de dispersão.
O pacote também contém dois conjuntos de dados de sequenciamento de RNA, chamados GeneCounts e GeneCounts2. Consulte as vinhetas do pacote para obter mais detalhes. Uma visão geral do pacote é ilustrada abaixo.
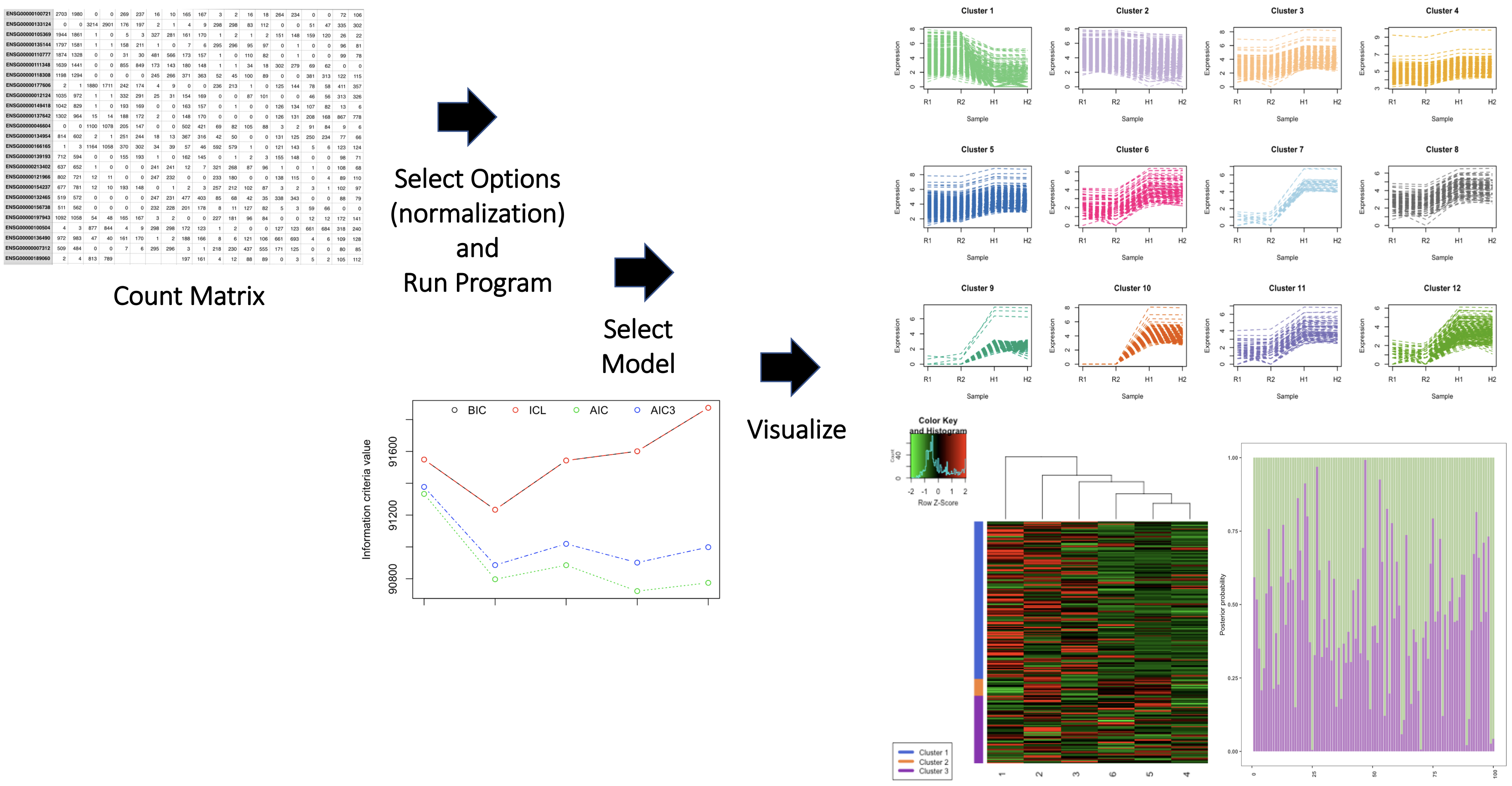
Forneça um parágrafo indicando claramente o nome do autor do pacote e as contribuições do autor. Descreva as contribuições de outros pacotes/fontes para cada função. Contribuições de esboço das ferramentas generativas de IA para cada função. Inclua como as ferramentas foram usadas e como os resultados das ferramentas de IA foram incorporados. Lembre -se de que suas contribuições individuais para o pacote são importantes. Por exemplo,
O autor do pacote é Anjali Silva. O autor escreveu a função de infecculação , que calcula os valores dos critérios de informação, dadas as especificações de dados. Aqui, são calculados o critério de informação bayesiano (BIC), o critério de informação de Akaike (AIC) e a probabilidade completa integrada (ICL). A função de infcritriacalCulation faz uso da função de mapa do pacote mclust R para gerar valores de critérios de informação. O pacote stats R é usado para gerar vetores de números aleatórios distribuídos multinomialmente. Parte do código para função de infeccloculação foi retirada do pacote <NamePackage> r. (A seção do código emprestado deve ser claramente indicada e referenciada no script de infecclealculation r). O plot infectria é escrito pelo autor e gera um gráfico de valores de critérios de informação. A função InFCRITERIAPLOT usa o pacote graphics R. Normfactors é uma função que calcula os fatores de normalização por meio de valores M (TMM) aparados (TMM). A função dos normatores usa a média aparada dos valores M (TMM), conforme implementado no pacote edgeR R. Nenhuma ferramenta de IA generativa foi usada no desenvolvimento deste pacote.
Forneça referências completas para todas as fontes utilizadas, inclusive para os pacotes e ferramentas mencionados em 'contribuições', em um formato. Por exemplo,
Akaike, H. (1973). Teoria da informação e uma extensão do princípio da máxima verossimilhança. Em Segundo Simpósio Internacional sobre Teoria da Informação , Nova York, EUA, 267-281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux e G. Govaert (2000). Avaliando um modelo de mistura para agrupamento com a probabilidade de classificação integrada. Transações IEEE sobre análise de padrões e inteligência de máquina 22. https://hal.inria.fr/inria-00073163/document
Biornder. (2020). Imagem criada por Silva, A. Recuperado em 30 de outubro de 2020, de https://app.biornder.com/
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipesert, B. Borges (2023). brilhante: estrutura de aplicativos da web para r . R Versão 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen e GK Smyth (2012). Análise de expressão diferencial de experimentos de RNA-seq multifatoriais em relação à variação biológica. Pesquisa de ácidos nucleicos 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R Equipe Core (2023). R: Um idioma e ambiente para computação estatística. R Fundação para Computação Estatística, Viena, Áustria. https://www.r-project.org/
Robinson, MD, DJ McCarthy e GK Smyth (2010). Edger: Um pacote de biocondutor para análise de expressão diferencial de dados de expressão de genes digitais. Bioinformatics 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). Estimando a dimensão de um modelo. The Annals of Statistics 6, 461-464. https://projeteuclid.org/euclid.aos/1176344136.
Scrucca, L., M. Fop, TB Murphy e Ae Raftery (2016) McLust 5: Estimativa de agrupamento, classificação e densidade usando modelos de mistura finita gaussiana. The R Journal 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. GGPlot2: gráficos elegantes para análise de dados. Springer-Verlag Nova York, 2016.
Wickham, H. e J. Bryan (2019). R Pacotes (2ª edição). Newton, Massachusetts: O'Reilly Media. https://r-pkgs.org/
Forneça o seguinte texto, personalizado para o seu pacote R. Por exemplo,
Este pacote foi desenvolvido como parte de uma avaliação para 2019-2023 BCB410H: Curso de Bioinformática Aplicada na Universidade de Toronto, Toronto, Canadá. TestingPackage recebe questões, solicitações de aprimoramento e outras contribuições. Para enviar um problema, use os problemas do GitHub. Muito obrigado a aqueles que forneceram feedback para melhorar este pacote.
Pacotes desenvolvidos pelos ex -alunos do BCB410. Muito obrigado a aqueles que forneceram permissão para compartilhar seus pacotes!
Atlaz
ClinicalTranscriptLink
Compheatmaps
Covidwastewatch
DrugSeekr
Geanaly
Integratrn
Isoformvisrna
MicrobiomeExplorer
MissensePathor
OpenVirome
Osteoanalizador
PDBCLEANUP
Fenogenrlib
Prostat5
Psychcnvassoc
Seurattogo
Tmscorealign
Trexdad
Trnvalstandvis
Abunrna
Dereggenes
CNVDS
ONONALIAL
SexDisagregate
scrgnet
Deviscomp
CoVID19CANADA
IntegrationsCore
Myomanager
snpcyp
VisualSarsDiff
Clusteval
Clustphy
gscvisualizador
microcompet
UniprotProteinView
Methylexpress
Dynugene; manuscrito
PDB3D
Strexpansionanalyzer
CellTypetool
Chipanalyzer
A estrutura do pacote é ilustrada abaixo:
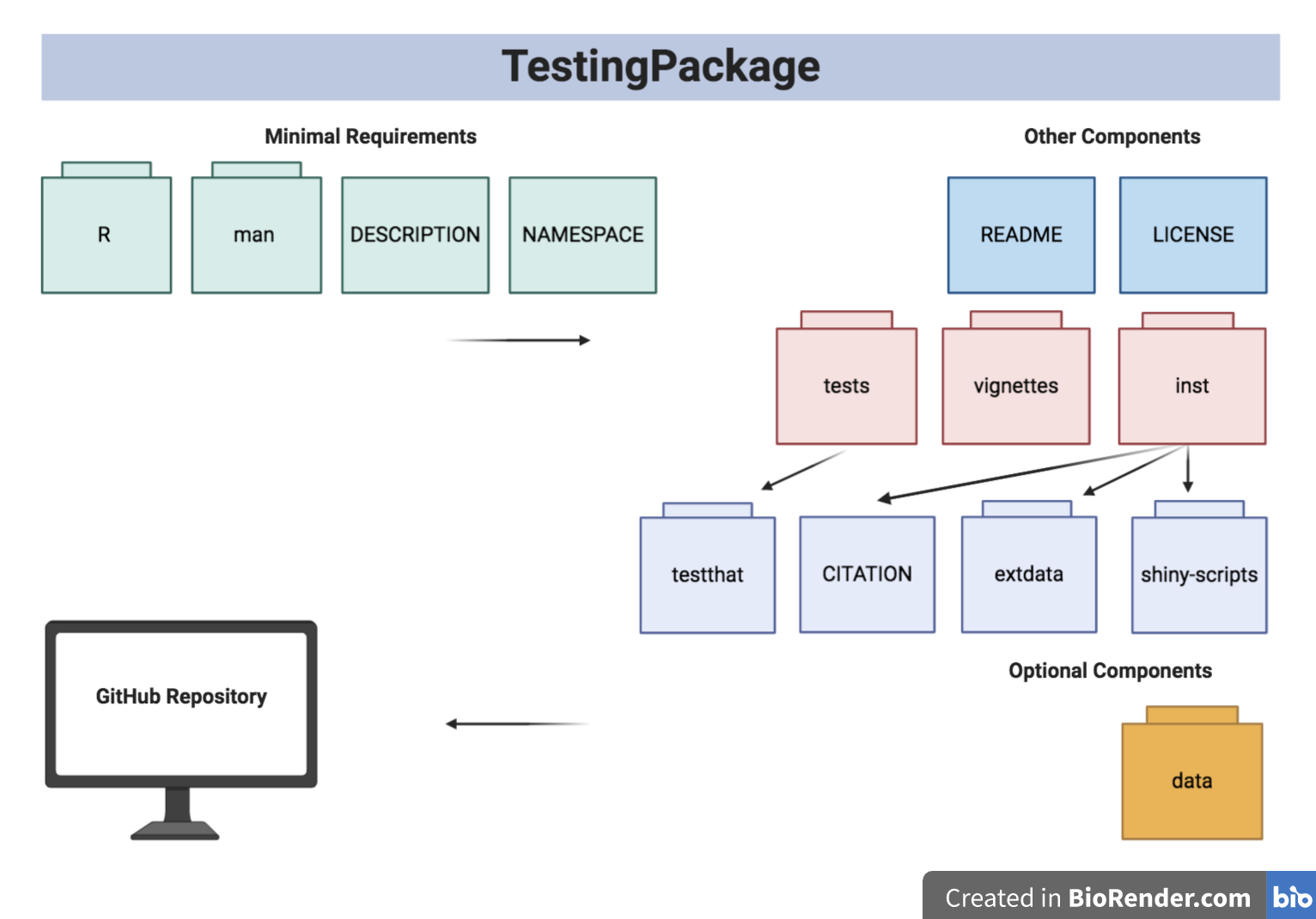
A estrutura da árvore da embalagem é fornecida abaixo.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R