Un exemple de package R pour BCB410H: bioinformatique appliquée.
Un paragraphe qui décrit l'objectif de votre package R et des données biologiques analysées. Expliquez comment votre package ajoute ou améliore un flux de travail actuel en bioinformatique ou biologie informatique (c'est-à-dire comment est-il unique ?, Quel problème s'adresse-t-il?). Enfin, incluez la version R (pas la version RStudio) et la plate-forme (Mac, Windows, Linux (Debian, Fedora / Redhat, Ubuntu)), utilisé pour développer le package. Vous pouvez obtenir ces informations en exécutant utils::sessionInfo() . Il ne devrait pas y avoir de mise en œuvre Shiny à ce stade. Par exemple,
TestingPackage est un package R pour démontrer les composants d'un package R simple avec des données de séquençage d'ARN. Le package R comprend les principaux composants: Description, Espace de noms, sous-répertoire man et sous-répertoire R. De plus, les vignettes, les tests, les données, les données et l'inst sont également explorés. Le package est ciblé pour les étudiants BCB410H (Bioinformatique appliquée), qui doivent définir un outil utile pour l'analyse des données biologiques sous le format d'un package R Public sur GitHub. La portée du package R est d'ajouter ou d'améliorer un flux de travail actuel en bioinformatique ou en biologie informatique. L'outil doit contenir des fonctions pour effectuer une analyse des données biologiques et pour produire une sortie graphique convaincante, idéalement pour prendre en charge une analyse exploratoire. Le package TestingPackage a été développé à l'aide de R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) et Running under: macOS Ventura 13.2 .
Fournissez le texte et les commandes suivants, personnalisés dans votre package R. Par exemple,
Pour installer la dernière version du package:
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )Pour exécuter l'application brillante:
runTestingPackage() # not for Assessment 4; only for Assessment 5 Fournissez les commandes suivantes, personnalisées à votre package R. Fournissez ensuite une liste des fonctions accessibles aux utilisateurs dans le package et une brève description de chacun. Incluez une image illustrant l'aperçu du package qui montre les entrées et sorties. Assurez-vous que l'image est déposée dans le bon endroit, comme discuté en classe. Poignez l'utilisateur vers des vignettes pour un tutoriel de votre package. Par exemple,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage contient 3 fonctions.
Infrgiteriacalcul pour calculer les critères d'information compte tenu des dimensions de l'ensemble de données, du journal log-vraisemblance et de la probabilité.
Normfacteurs pour calculer les facteurs de normalisation via via la moyenne garnie des valeurs M (TMM).
InfrcriteriAplot pour tracer les valeurs des critères d'information sous forme de tracé de dispersion.
Le package contient également deux ensembles de données de séquençage d'ARN, appelés genecounts et genecounts2. Reportez-vous aux vignettes du package pour plus de détails. Un aperçu du package est illustré ci-dessous.
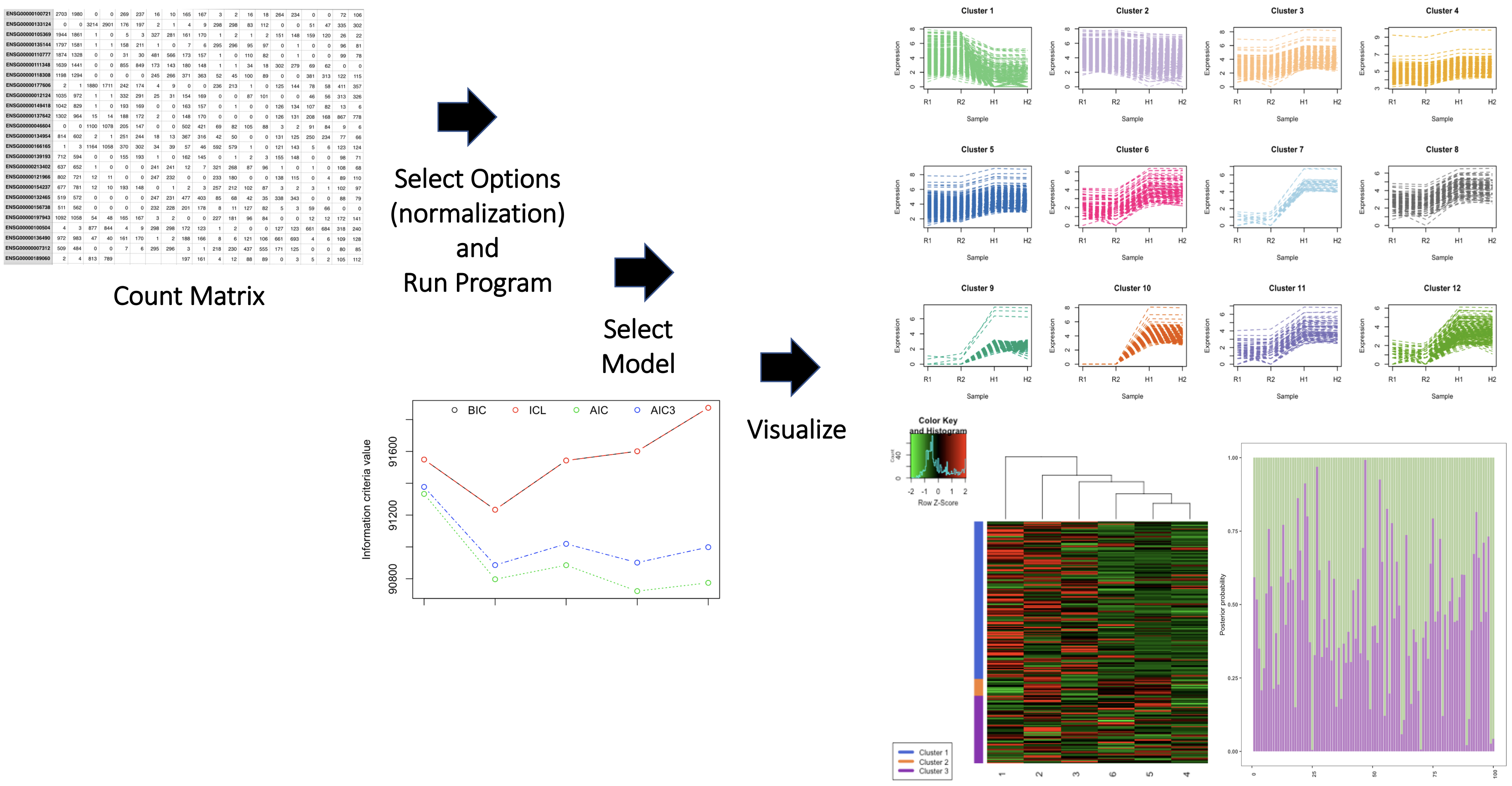
Fournir un paragraphe indiquant clairement le nom de l'auteur du package et des contributions de l'auteur. Décrivez les contributions d'autres packages / sources pour chaque fonction. Décrivez les contributions de l'outil d'IA génératif pour chaque fonction. Incluez la façon dont les outils ont été utilisés et comment les résultats des outils d'IA ont été incorporés. N'oubliez pas que vos contributions individuelles au package sont importantes. Par exemple,
L'auteur du package est Anjali Silva. L'auteur a rédigé la fonction Infrcriteriacalcul , qui calcule les valeurs des critères d'information compte tenu des spécifications de données. Ici, le critère d'information bayésien (BIC), le critère d'information Akaike (AIC) et la vraisemblance intégrale (ICL) sont calculés. La fonction Infrcriteriacalcul utilise la fonction MAP à partir du package mclust R pour générer des valeurs de critères d'information. Le package stats R est utilisé pour générer des vecteurs de nombres aléatoires distribués multinomialement. Une partie du code de fonction Infrcriteriacalcul a été tirée du package <NamePackage> r. (La section du code emprunté doit être clairement indiquée et référencée dans le script RENCRITERIACALCULATION R). L' engradiaplot est rédigé par l'auteur et génère une parcelle de valeurs de critères d'information. La fonction InfrcriteriAplot utilise le package graphics R. Normfactors est une fonction qui calcule les facteurs de normalisation via la moyenne coupée des valeurs M (TMM). La fonction NORMFACTORS utilise la moyenne coupée des valeurs M (TMM) telles qu'implémentées dans le package edgeR R. Aucun outil d'IA génératif n'a été utilisé dans le développement de ce package.
Fournir des références complètes pour toutes les sources utilisées, y compris pour les packages et les outils mentionnés sous «contributions», dans un format. Par exemple,
Akaike, H. (1973). Théorie de l'information et extension du principe du maximum de vraisemblance. Dans Second International Symposium on Information Theory , New York, États-Unis, 267-281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux et G. Govaert (2000). Évaluation d'un modèle de mélange pour le regroupement avec la probabilité de classification intégrée. Transactions IEEE sur l'analyse des modèles et le renseignement des machines 22. https://hal.inria.fr/inria-00073163/document
Biorender. (2020). Image créée par Silva, A. Extrait le 30 octobre 2020 à partir de https://app.biorender.com/
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023). Shiny: Framework d'application Web pour R. R Package Version 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen et Gk Smyth (2012). Analyse de l'expression différentielle des expériences d'ARN-Seq multifactoraires en ce qui concerne la variation biologique. Recherche des acides nucléiques 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/22287627/
R Core Team (2023). R: Un langage et un environnement pour l'informatique statistique. R Foundation for Statistical Computing, Vienne, Autriche. https://www.r-project.org/
Robinson, MD, DJ McCarthy et GK Smyth (2010). EDGER: un package de bioconducteur pour l'analyse de l'expression différentielle des données d'expression des gènes numériques. Bioinformatics 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). Estimation de la dimension d'un modèle. Les annales des statistiques 6, 461–464. https://projecteuclid.org/euclid.aos/1176344136.
Scrucca, L., M. Fop, TB Murphy et Ae Raftery (2016) McLust 5: regroupement, classification et estimation de la densité à l'aide de modèles de mélange fini gaussien. The R Journal 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. GGPLOT2: Graphiques élégants pour l'analyse des données. Springer-Verlag New York, 2016.
Wickham, H. et J. Bryan (2019). Packages R (2e édition). Newton, Massachusetts: O'Reilly Media. https://r-pkgs.org/
Fournissez le texte suivant, personnalisé à votre package R. Par exemple,
Ce package a été développé dans le cadre d'une évaluation pour le cours de bioinformatique 2019-2023: le cours de bioinformatique appliqué à l'Université de Toronto, Toronto, Canada. TestingPackage accueille des problèmes, des demandes d'amélioration et d'autres contributions. Pour soumettre un problème, utilisez les problèmes GitHub. Un grand merci à ceux qui ont fourni des commentaires pour améliorer ce package.
Packages développés par les anciens de BCB410. Un grand merci à ceux qui ont donné la permission de partager leurs forfaits!
Atlaz
ClinicalTranscriptLink
Compheatmaps
CovidWastewatch
Drogueseekr
Geanaly
Intégatrn
Isoformvisrna
Microbiomeexplorer
MissounsePathor
Viromaire
Ostéoanaliseur
Pdbcleanup
Phénogènerlib
Prostat5
psychcnvassoc
Seurattogo
TMScoreAlign
Trexdad
Trnvalstandvis
Abunrna
Dégins
CNVDS
oncoanalyse
sexdisaggregate
recruter
Deviscompitre
Covid19Canada
Intégrations
Myomanager
snpcyp
VisualSarsDiff
clustreval
Mine
GSCVisualizer
microcompété
UniprotproteinView
Méthylexpress
dynugène; manuscrit
pdb3d
Strexpansionanalyzer
Celltypetool
Chipanalyzer
La structure du package est illustrée ci-dessous:
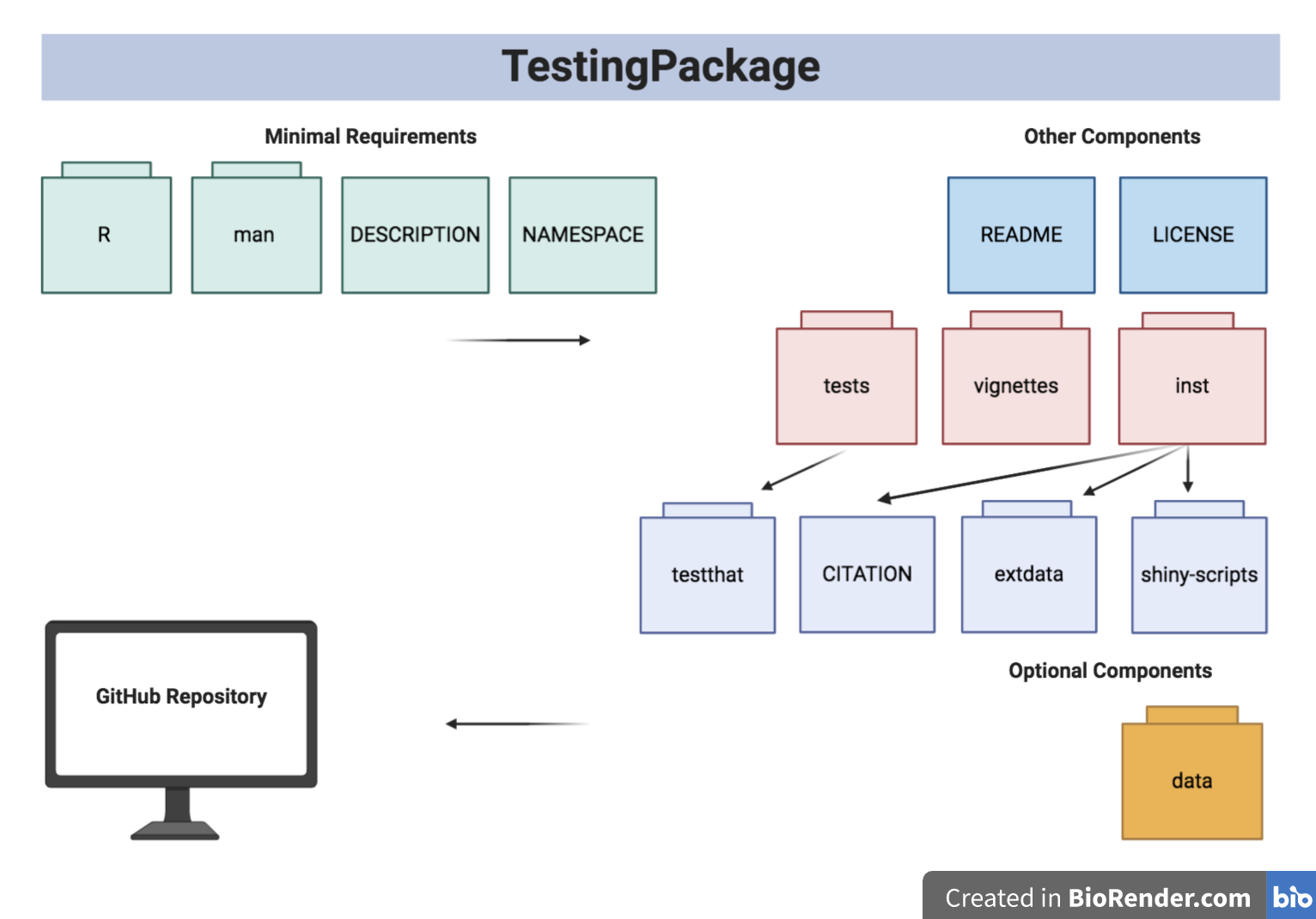
La structure de l'arbre de l'emballage est fournie ci-dessous.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R