BCB410H의 예제 R 패키지 : 응용 생물 정보학.
R 패키지의 목적을 설명하는 단락 및 분석중인 생물학적 데이터. 패키지가 생물 정보학 또는 계산 생물학의 현재 작업 흐름에 어떻게 추가되거나 개선되는지 설명하십시오 (예 : 어떻게 독특합니까?, 어떤 문제가 해결됩니까?). 마지막으로, 패키지를 개발하는 데 사용되는 R 버전 (Rstudio 버전이 아님) 및 플랫폼 (Mac, Windows, Linux (Debian, Fedora/Redhat, Ubuntu))를 포함하십시오. utils::sessionInfo() 실행 하여이 정보를 얻을 수 있습니다. 이 시점에서 Shiny 구현은 없어야합니다. 예,
TestingPackage 는 RNA 시퀀싱 데이터가있는 간단한 R 패키지의 구성 요소를 보여주는 R 패키지입니다. R 패키지에는 설명, 네임 스페이스, MAN 하위 디렉토리 및 R 하위 디렉토리의 주요 구성 요소가 포함되어 있습니다. 또한 라이센스, 읽기 및 하위 디렉토리 비네팅, 테스트, 데이터 및 Inst도 탐색됩니다. 이 패키지는 GitHub에 보관 된 공개 R 패키지 형식으로 생물학적 데이터 분석에 유용한 도구를 정의 해야하는 BCB410H (Applied Bioinformatics) 학생을 대상으로합니다. R 패키지의 범위는 생물 정보학 또는 계산 생물학의 현재 작업 흐름을 추가하거나 개선하는 것입니다. 이 도구에는 생물학적 데이터 분석을 수행하고 탐색 적 분석을 지원하기 위해 강력한 그래픽 출력을 생성하는 기능이 포함되어야합니다. TestingPackage 패키지는 R version 4.2.1 (2022-06-23) , Platform: x86_64-apple-darwin17.0 (64-bit) Running under: macOS Ventura 13.2 하여 개발되었습니다.
R 패키지에 맞춤화 된 다음 텍스트 및 명령을 제공하십시오. 예,
최신 버전의 패키지를 설치하려면 :
install.packages( " devtools " )
library( " devtools " )
devtools :: install_github( " anjalisilva/TestingPackage " , build_vignettes = TRUE )
library( " TestingPackage " )반짝이는 앱을 실행하려면 :
runTestingPackage() # not for Assessment 4; only for Assessment 5 R 패키지에 사용자 정의 된 다음 명령을 제공하십시오. 그런 다음 패키지 내에서 사용자 액세스 가능한 기능 목록과 각각의 간단한 설명을 제공하십시오. 입력 및 출력을 보여주는 패키지 개요를 보여주는 이미지 하나를 포함하십시오. 수업에서 논의 된대로 이미지가 올바른 위치에 입금되어 있는지 확인하십시오. 패키지 튜토리얼을 위해 사용자에게 비네팅을 지적하십시오. 예,
ls( " package:TestingPackage " )
data( package = " TestingPackage " )
browseVignettes( " TestingPackage " ) TestingPackage 에는 3 가지 기능이 포함되어 있습니다.
데이터 세트 차원, 로그 유명성 및 확률이 주어진 정보 기준 계산을위한 인프스틱-계산 .
M- 값의 평균 (TMM)을 통해 정규화 계수를 계산하기위한 NORMFACTORS .
정보 기준 값을 도표로 플로팅하기위한 InfcriteriaPlot .
패키지에는 또한 Genecounts 및 Genecounts2라는 두 개의 RNA 시퀀싱 데이터 세트가 포함되어 있습니다. 자세한 내용은 패키지 비네팅을 참조하십시오. 패키지 개요는 다음과 같습니다.
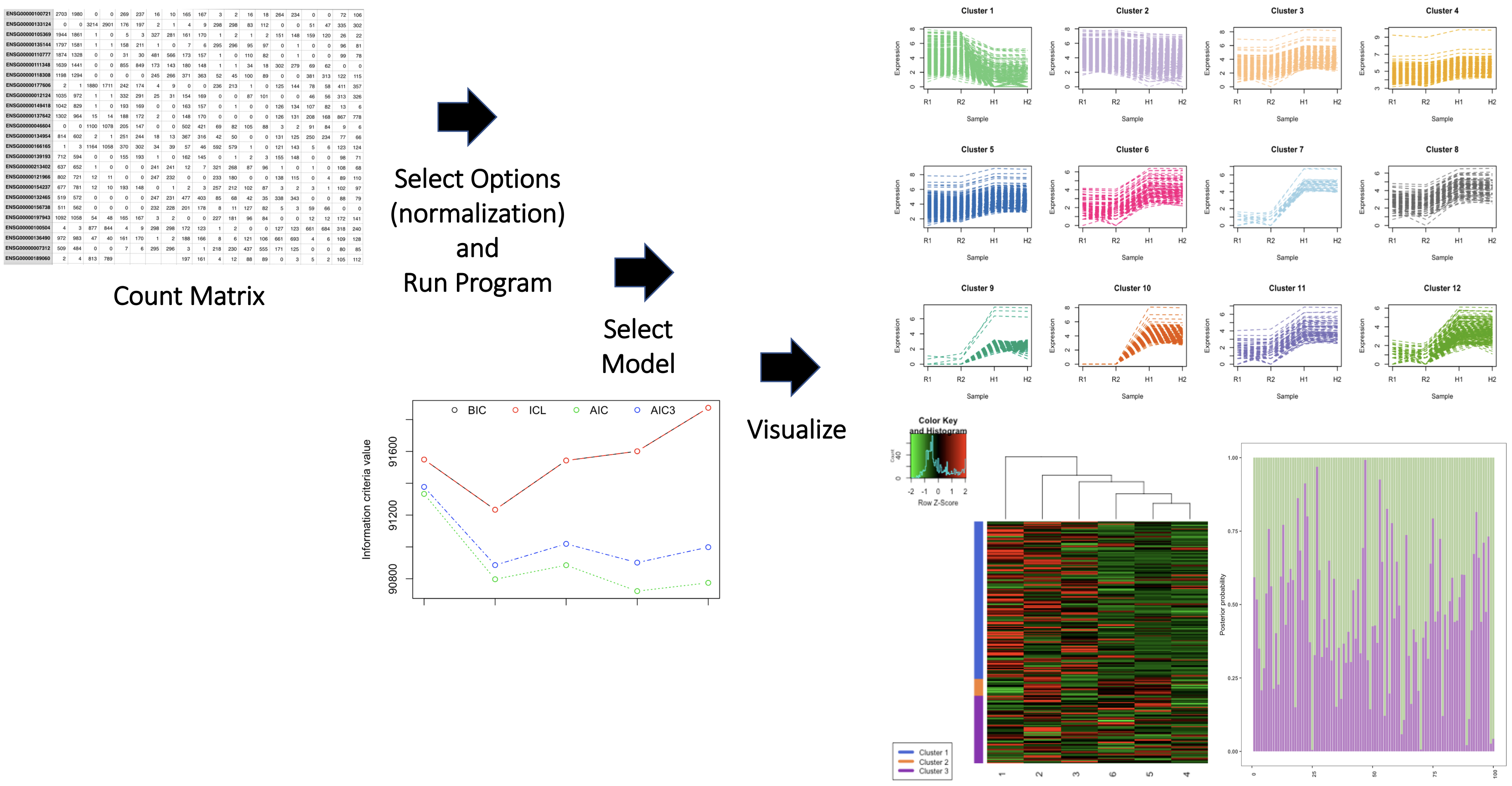
패키지의 저자의 이름과 저자의 기여를 명확하게 나타내는 단락을 제공하십시오. 각 함수에 대한 다른 패키지/소스의 기여를 간략하게 설명합니다. 각 함수에 대한 생성 AI 도구의 기여도를 개요합니다. 도구 사용 방법과 AI 도구의 결과가 통합 된 방법을 포함하십시오. 패키지에 대한 개인의 기여가 중요하다는 것을 기억하십시오. 예,
패키지의 저자는 Anjali Silva입니다. 저자는 데이터 사양에 따른 정보 기준 값을 계산하는 인프스틱 계산 기능을 작성했습니다. 여기서는 베이지안 정보 기준 (BIC), AKAIKE 정보 기준 (AIC) 및 통합 완전한 가능성 (ICL)이 계산됩니다. Infcriteriacalculation 함수는 mclust R 패키지의 맵 함수를 사용하여 정보 기준 값을 생성합니다. stats R 패키지는 다중 분산 임의의 숫자 벡터를 생성하는 데 사용됩니다. Infcriteriacalculation 기능 코드의 일부는 <NamePackage> r 패키지에서 가져 왔습니다. (차용 된 코드의 섹션은 Infcriteriacalculation r 스크립트에 명확하게 표시되고 참조되어야합니다). InfcriteriaPlot 은 저자가 작성하고 정보 기준 값의 플롯을 생성합니다. InfcriteriaPlot 함수는 graphics r 패키지를 사용합니다. NormFactors는 M- 값의 평균 (TMM)을 통해 정규화 계수를 계산하는 함수입니다. NormFactors 함수는 edgeR R 패키지에서 구현 된대로 TMM (Trimmed Mean of M-Values)을 사용합니다. 이 패키지 개발에는 생성 AI 도구가 사용되지 않았습니다.
'기부금'에 언급 된 패키지 및 도구를 포함하여 사용 된 모든 소스에 대해 한 형식의 전체 참조를 제공하십시오. 예,
Akaike, H. (1973). 정보 이론과 최대 가능성 원칙의 확장. 정보 이론에 관한 제 2 차 국제 심포지엄 , 뉴욕, 미국, 267–281. Springer Verlag. https://link.springer.com/chapter/10.1007/978-1-4612-1694-0_15.
Biernacki, C., G. Celeux 및 G. Govaert (2000). 통합 분류 가능성으로 클러스터링을위한 혼합 모델 평가. 패턴 분석 및 기계 지능에 대한 IEEE 거래 22. https://hal.inria.fr/inria-00073163/document
바이오 렌더. (2020). Silva, A. 2020 년 10 월 30 일에 https://app.biorender.com/에서 작성된 이미지
Chang, W., J. Cheng, J. Allaire, C. Sievert, B. Schloerke, Y. Xie, J. Allen, J. McPherson, A. Dipert, B. Borges (2023). Shiny : r에 대한 웹 응용 프로그램 프레임 워크 . R 패키지 버전 1.8.0, https://cran.r-project.org/package=shiny
McCarthy, DJ, Y. Chen 및 GK Smyth (2012). 생물학적 변화에 대한 다중 인자 RNA-Seq 실험의 미분 발현 분석. 핵산 연구 40. 4288-4297. https://pubmed.ncbi.nlm.nih.gov/2287627/
r 핵심 팀 (2023). R : 통계 컴퓨팅을위한 언어 및 환경. R 오스트리아 비엔나 통계 컴퓨팅 재단. https://www.r-project.org/
Robinson, MD, DJ McCarthy 및 GK Smyth (2010). EDGER : 디지털 유전자 발현 데이터의 차등 발현 분석을위한 생체 결핵 패키지. 생물 정보학 26, 139-140. https://pubmed.ncbi.nlm.nih.gov/19910308/
Schwarz, G. (1978). 모델의 차원 추정. 통계 6, 461–464의 연대기 . https://projecteuclid.org/euclid.aos/1176344136.
Scrucca, L., M. Fop, TB Murphy 및 Ae Raftery (2016) McLust 5 : 가우스 유한 혼합 모델을 사용한 클러스터링, 분류 및 밀도 추정. R Journal 8 (1), 289-317. https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5096736/
Wickham, H. GGPLOT2 : 데이터 분석을위한 우아한 그래픽. Springer-Verlag New York, 2016.
Wickham, H. 및 J. Bryan (2019). R 패키지 (2 판). 뉴턴, 매사추세츠 : 오라일리 미디어. https://r-pkgs.org/
R 패키지에 사용자 정의 된 다음 텍스트를 제공하십시오. 예,
이 패키지는 2019-2023 BCB410H : 캐나다 토론토 대학교에서 Applied Bioinformatics Course의 평가의 일부로 개발되었습니다. TestingPackage 문제, 강화 요청 및 기타 기부금을 환영합니다. 문제를 제출하려면 Github 문제를 사용하십시오. 이 패키지를 개선하기 위해 피드백을 제공 한 분들께 감사드립니다.
BCB410 동문이 개발 한 패키지. 패키지를 공유 할 수있는 권한을 제공 한 분들께 감사드립니다!
Atlaz
ClinicalTranscriptLink
Compheatmaps
Covidwastewatch
Drugseekr
Geanaly
integrn
이소 ormvisrna
미생물 수거서
MissensePathor
Openvirome
오스테 오아 날화제
pdbcleanup
Phenogenrlib
전립선 5
Psychcnvassoc
서라토고
tmscorealign
트렉 다드
trnvalstandvis
abunrna
Dereggenes
CNVD
종양 분석
sexdisaggregate
scrgnet
deviscomp
Covid19canada
IntegrationsCore
MYONAGER
snpcyp
visualsarsdiff
clustreval
Clustphy
gscvisualizer
마이크로 컴퓨터
UnipRotProteInview
Methylexpress
Dynugene; 원고
PDB3D
Strexpansionanalyzer
Celltypetool
Chipanalyzer
패키지 구조는 다음과 같습니다.
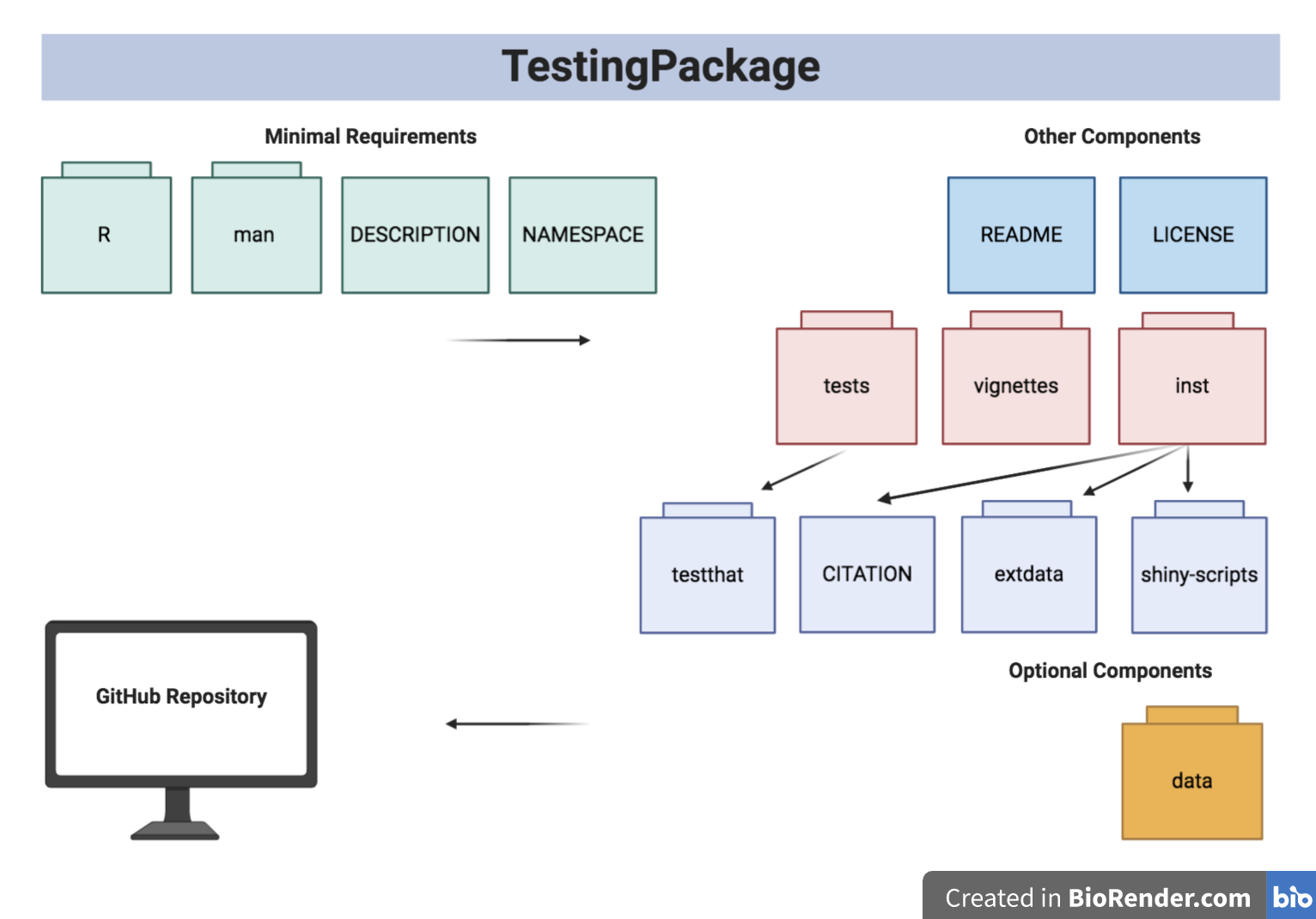
패키지 트리 구조는 아래에 제공됩니다.
- TestingPackage
| - TestingPackage.Rproj
| - DESCRIPTION
| - NAMESPACE
| - LICENSE
| - README
| - data
| - GeneCounts.rda
| - GeneCounts2.rda
| - inst
CITATION
| - extdata
| - SILVA_A_A1.png
| - GeneCountsData2.csv
| - shiny - scripts
| - app.R
| - man
| - GeneCounts.Rd
| - InfCriteriaCalculation.Rd
| - NormFactors.Rd
| - InfCriteriaPlot.Rd
| - R
| - data.R
| - InfCriteriaCalculation.R
| - InfCriteriaPlot.R
| - NormFactorCalculation.R
| - vignettes
| - TestingPackageVignette.Rmd
| - tests
| - testthat.R
| - testthat
| - test - InfCriteriaCalculation.R