การใช้งาน Pytorch ของ 3D U-Net และตัวแปร:
UNet3D มาตรฐาน 3D U-NET ตาม 3D U-NET: การเรียนรู้การแบ่งส่วนปริมาตรหนาแน่นจากคำอธิบายประกอบแบบกระจัดกระจาย
ResidualUNet3D เหลือ 3D U-Net ที่เหลืออยู่บนพื้นฐานของความแม่นยำเหนือมนุษย์ในการแข่งขัน SNEMI3D Connectomics Challenge
ResidualUNetSE3D คล้ายกับ ResidualUNet3D ด้วยการเพิ่มการบีบและบล็อกการกระตุ้นขึ้นอยู่กับการแบ่งส่วนความหมายการเรียนรู้ลึกสำหรับปริมาณการแพทย์ที่มีความละเอียดสูง กระดาษบีบดั้งเดิมและ Excite: เครือข่ายการบีบและการเร่งความเร็ว
รหัสอนุญาตให้ฝึกอบรม U-NET สำหรับทั้งสอง: การแบ่งส่วนความหมาย (ไบนารีและหลายคลาส) และปัญหา การถดถอย (เช่นการยกเลิกการเรียนรู้การเรียนรู้การเรียนรู้)
นอกจากนี้ยังรองรับ 2D U-NET ดู 2DUNET_CONFOCAL หรือ 2DUNET_DSB2018 สำหรับการกำหนดค่า เพียงตรวจสอบให้แน่ใจว่าได้เก็บมิติซิงเกิล Z ในชุดข้อมูล H5 ของคุณ (เช่น (1, Y, X) (1, 3, 3) (Y, X) )) เนื่องจากการโหลดข้อมูล / การเพิ่มข้อมูลต้องใช้เทนเซอร์ของอันดับ 3. 2D U-NET ใช้เลเยอร์ 2D มาตรฐาน
ข้อมูลอินพุตควรเก็บไว้ในไฟล์ HDF5 ไฟล์ HDF5 สำหรับการฝึกอบรมควรมีชุดข้อมูลสองชุด: raw และ label ทางเลือกเมื่อการฝึกอบรมกับ PixelWiseCrossEntropyLoss หนึ่งควรจัดเตรียมชุดข้อมูล weight ชุดข้อมูล raw ควรมีข้อมูลอินพุตในขณะที่ชุดข้อมูล label ป้ายความจริงภาคพื้นดิน ชุดข้อมูล weight เสริมควรมีค่าสำหรับการถ่วงน้ำหนักฟังก์ชั่นการสูญเสียในภูมิภาคต่าง ๆ ของอินพุตและควรมีขนาดเท่ากันกับชุดข้อมูล label รูปแบบของชุดข้อมูล raw / label ขึ้นอยู่กับว่าปัญหาคือ 2D หรือ 3D และไม่ว่าข้อมูลจะเป็นช่องทางเดียวหรือหลายช่องทางดูตารางด้านล่าง:
| 2d | 3D | |
|---|---|---|
| ช่องเดี่ยว | (1, y, x) | (z, y, x) |
| หลายช่องทาง | (c, 1, y, x) | (c, z, y, x) |
pytorch-3dunet เป็นแพ็คเกจข้ามแพลตฟอร์มและทำงานบน Windows และ OS X เช่นกัน
pytorch-3dunet คือผ่าน Conda/Mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
หลังจากการติดตั้งคำสั่งต่อไปนี้สามารถเข้าถึงได้ภายในสภาพแวดล้อม conda: train3dunet สำหรับการฝึกอบรมเครือข่ายและ predict3dunet สำหรับการทำนาย (ดูด้านล่าง)
python setup.py install
ตรวจสอบให้แน่ใจว่า pytorch ที่ติดตั้งเข้ากันได้กับรุ่น CUDA ของคุณมิฉะนั้นการฝึกอบรม/การทำนายจะล้มเหลวในการทำงานบน GPU
เนื่องจากแพ็คเกจ pytorch-3dunet ได้รับการติดตั้งผ่าน conda ตามที่อธิบายไว้ข้างต้นเราสามารถฝึกอบรมเครือข่ายได้โดยเพียงแค่เรียก:
train3dunet --config <CONFIG>
โดยที่ CONFIG เป็นพา ธ ไปยังไฟล์กำหนดค่า YAML ซึ่งระบุทุกด้านของขั้นตอนการฝึกอบรม
เพื่อฝึกอบรมข้อมูลของคุณเองเพียงแค่ให้เส้นทางไปยังชุดข้อมูลการฝึกอบรมและการตรวจสอบความถูกต้องของ HDF5 ในการกำหนดค่า
หนึ่งสามารถตรวจสอบความคืบหน้าการฝึกอบรมด้วย Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (คุณต้องใช้ tensorflow ที่ติดตั้งใน Conda Env ของคุณ) โดยที่ checkpoint_dir เป็นเส้นทางไปยังไดเรกทอรีจุดตรวจสอบที่ระบุไว้ในการกำหนดค่า
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : ข้อมูลเป้าหมายจะต้องเป็น 4D (หน้ากากไบนารีเป้าหมายหนึ่งตัวต่อช่อง) เมื่อการฝึกอบรมกับ WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ชุดข้อมูลเป้าหมายจะต้องเป็น 3D ดูเอกสาร pytorch สำหรับการสูญเสีย CE: https://pytorch.org/docs/master/generated/torch.nn.nn.nn.nn.nn.nn.nn.nn.nn.nn.final_sigmoid ในส่วนการกำหนดค่า model ใช้เฉพาะกับเวลาอนุมาน (การตรวจสอบความถูกต้องการทดสอบ):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) ตั้งค่า final_sigmoid=False เพื่อให้การทำให้เป็นมาตรฐาน Softmax ถูกนำไปใช้กับผลลัพธ์ เนื่องจากแพ็คเกจ pytorch-3dunet ได้รับการติดตั้งผ่าน conda ตามที่อธิบายไว้ข้างต้นเราสามารถเรียกใช้การทำนายผ่าน:
predict3dunet --config <CONFIG>
ในการทำนายข้อมูลของคุณเองเพียงจัดเตรียมเส้นทางไปยังโมเดลของคุณเช่นเดียวกับเส้นทางไปยังไฟล์ทดสอบ HDF5 (ดูตัวอย่าง test_config_segmentation.yaml)
LazyHDF5Dataset และ LazyPredictor ในการกำหนดค่า สิ่งนี้จะช่วยประหยัดหน่วยความจำโดยการโหลดข้อมูลได้ทันทีที่ค่าใช้จ่ายในการทำนายช้าลง ดู test_config_lazy สำหรับการกำหนดค่าตัวอย่างsave_segmentation: true ในส่วน predictor ของการกำหนดค่า (ดู test_config_multiclass) โดยค่าเริ่มต้นหาก GPU หลายตัวมีการฝึกอบรม/การทำนายที่พร้อมใช้งานจะทำงานบน GPU ทั้งหมดโดยใช้ DataParallel หากการฝึกอบรม/การทำนายเกี่ยวกับ GPU ที่มีอยู่ทั้งหมดไม่เป็นที่ต้องการให้ จำกัด จำนวน GPU โดยใช้ CUDA_VISIBLE_DEVICES เช่น
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >หรือ
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (ไบนารี cross-entropy)DiceLoss ( DiceLoss มาตรฐานกำหนดเป็น 1 - DiceCoefficient ที่ใช้สำหรับการแบ่งส่วนความหมายแบบไบนารีเมื่อมีมากกว่า 2 คลาสที่มีอยู่ในความจริงพื้นดินมันจะคำนวณ DiceLoss ต่อช่องและค่าเฉลี่ยค่า)BCEDiceLoss (การรวมกันเชิงเส้นของ BCE และการสูญเสียลูกเต๋าเช่น alpha * BCE + beta * Dice alpha, beta สามารถระบุได้ในส่วน loss ของการกำหนดค่า)CrossEntropyLoss (หนึ่งสามารถระบุน้ำหนักคลาสผ่าน weight: [w_1, ..., w_k] ในส่วน loss ของการกำหนดค่า)PixelWiseCrossEntropyLoss (หนึ่งสามารถระบุน้ำหนักต่อพิกเซลเพื่อให้การไล่ระดับสีมากขึ้นในภูมิภาคที่สำคัญ/ต่ำกว่าตัวแทนในความจริงพื้นดินชุดข้อมูล weight จะต้องจัดเตรียมไว้ในไฟล์ H5 สำหรับการฝึกอบรมและการตรวจสอบดูตัวอย่างการกำหนดค่าใน train_config.ymlWeightedCrossEntropyLoss (ดู 'ถ่วงน้ำหนักข้าม-เอนโทรปี (WCE)' ในกระดาษด้านล่างเพื่ออธิบายรายละเอียด)GeneralizedDiceLoss (ดู 'การสูญเสียลูกเต๋าทั่วไป (GDL)' ในกระดาษด้านล่างสำหรับคำอธิบายโดยละเอียด) หมายเหตุ: ใช้ฟังก์ชั่นการสูญเสียนี้เฉพาะในกรณีที่ฉลากในชุดข้อมูลการฝึกอบรมนั้นไม่สมดุลมากเช่นคลาสหนึ่งที่มีขนาดอย่างน้อย 3 คำสั่งซื้อ voxels มากกว่าคนอื่น ๆ มิฉะนั้นให้ใช้ DiceLoss มาตรฐานสำหรับคำอธิบายโดยละเอียดของฟังก์ชั่นการสูญเสียที่รองรับบางอย่างดู: ลูกเต๋าทั่วไปทับซ้อนกันเป็นฟังก์ชั่นการสูญเสียการเรียนรู้อย่างลึกซึ้งสำหรับการแบ่งส่วนที่ไม่สมดุลสูง
MSELoss (หมายถึงการสูญเสียข้อผิดพลาดกำลังสอง)L1Loss (ค่าเฉลี่ยการสูญเสียข้อผิดพลาดสัมบูรณ์)SmoothL1Loss (ไวต่อค่าผิดปกติน้อยกว่า mseloss)WeightedSmoothL1Loss (ส่วนขยายของ SmoothL1Loss ซึ่งช่วยให้น้ำหนักค่า voxel เหนือ/ต่ำกว่าเกณฑ์ที่กำหนดแตกต่างกัน) MeanIoU (ค่าเฉลี่ยสี่แยกสหภาพ)DiceCoefficient (คำนวณค่าสัมประสิทธิ์ลูกเต๋าช่องสัญญาณและส่งคืนค่าเฉลี่ย) หาก 3D U-NET ได้รับการฝึกฝนให้ทำนายขอบเขตของเซลล์เราสามารถใช้ตัวชี้วัดการแบ่งกลุ่มอินสแตนซ์แบบความหมายต่อไปนี้ (ตัวชี้วัดด้านล่างถูกคำนวณโดยการใช้ส่วนประกอบที่เชื่อมต่อบนแผนที่ขอบเขตขีด จำกัดBoundaryAveragePrecision (ความแม่นยำเฉลี่ยที่ใช้กับแผนที่ความน่าจะเป็นขอบเขต: เกณฑ์เอาต์พุตจากเครือข่ายใช้ส่วนประกอบที่เชื่อมต่อเพื่อรับการแบ่งส่วนและคำนวณ AP ระหว่างการแบ่งส่วนที่เกิดขึ้นและความจริงพื้นฐาน)AdaptedRandError (ดู http://brainiac2.mit.edu/snemi3d/evaluation สำหรับคำอธิบายโดยละเอียด)AveragePrecision (ดู https://www.kaggle.com/stkbailey/step-by-step-explanation-of-scoring-metric) หากไม่ได้ระบุ MeanIoU จะถูกใช้โดยค่าเริ่มต้น
PSNR (อัตราส่วนสัญญาณสูงสุดต่อเสียงรบกวน)MSE (ค่าเฉลี่ยข้อผิดพลาดกำลังสอง) การฝึกอบรม/การคาดการณ์สามารถพบได้ใน 3DUNET_LIGHTSheet_Boundary น้ำหนักรุ่นที่ผ่านการฝึกอบรมมาก่อนมีอยู่ที่นี่ เพื่อใช้โมเดลที่ผ่านการฝึกอบรมมาก่อนข้อมูลของคุณเอง:
best_checkpoint.pytorch จากลิงค์ด้านบนpredict3dunet --config test_config.ymlpre_trained ในการกำหนดค่า YAML เพื่อชี้ไปที่เส้นทาง best_checkpoint.pytorchข้อมูลที่ใช้สำหรับการฝึกอบรมสามารถดาวน์โหลดได้จากโครงการ OSF ต่อไปนี้:
ตัวอย่างการทำนาย z-slice ในชุดทดสอบ (ด้านบน: อินพุตดิบ, ด้านล่าง: การทำนายขอบเขต):
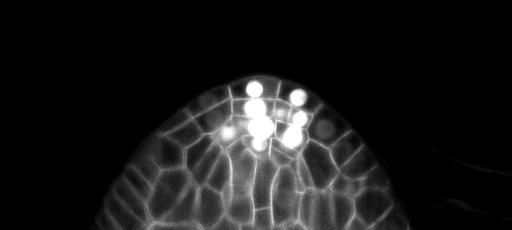

การฝึกอบรม/การคาดการณ์สามารถพบได้ใน 3DUNET_CONFOCAL_BONDARY น้ำหนักรุ่นที่ผ่านการฝึกอบรมมาก่อนมีอยู่ที่นี่ เพื่อใช้โมเดลที่ผ่านการฝึกอบรมมาก่อนข้อมูลของคุณเอง:
best_checkpoint.pytorch จากลิงค์ด้านบนpredict3dunet --config test_config.ymlpre_trained ในการกำหนดค่า YAML เพื่อชี้ไปที่เส้นทาง best_checkpoint.pytorchข้อมูลที่ใช้สำหรับการฝึกอบรมสามารถดาวน์โหลดได้จากโครงการ OSF ต่อไปนี้:
ตัวอย่างการทำนาย z-slice ในชุดทดสอบ (ด้านบน: อินพุตดิบ, ด้านล่าง: การทำนายขอบเขต):
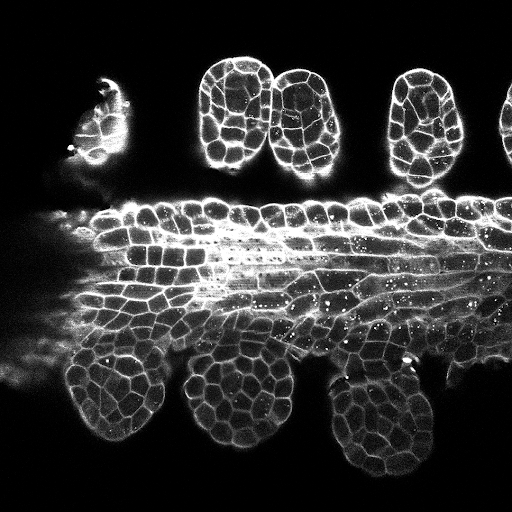
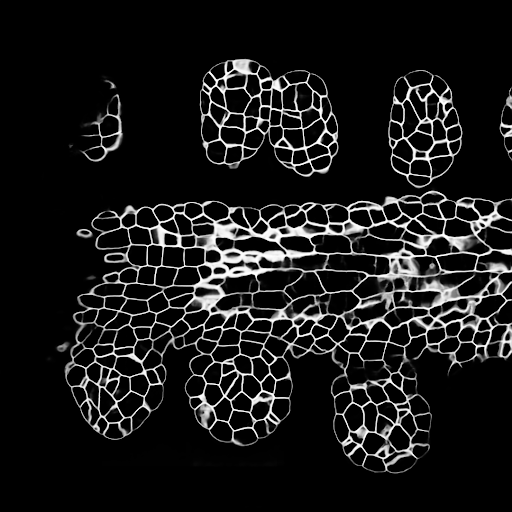
การฝึกอบรม/การคาดการณ์สามารถพบได้ใน 3DUNET_LIGHTSheet_Nuclei น้ำหนักรุ่นที่ผ่านการฝึกอบรมมาก่อนมีอยู่ที่นี่ เพื่อใช้โมเดลที่ผ่านการฝึกอบรมมาก่อนข้อมูลของคุณเอง:
best_checkpoint.pytorch จากลิงค์ด้านบนpredict3dunet --config test_config.ymlpre_trained ในการกำหนดค่า YAML เพื่อชี้ไปที่เส้นทาง best_checkpoint.pytorchชุดการฝึกอบรมและการตรวจสอบสามารถดาวน์โหลดได้จากโครงการ OSF ต่อไปนี้: https://osf.io/thxzn/
ตัวอย่างการทำนาย z-slice ในชุดทดสอบ (ด้านบน: อินพุตดิบ, ด้านล่าง: การทำนายนิวเคลียส):
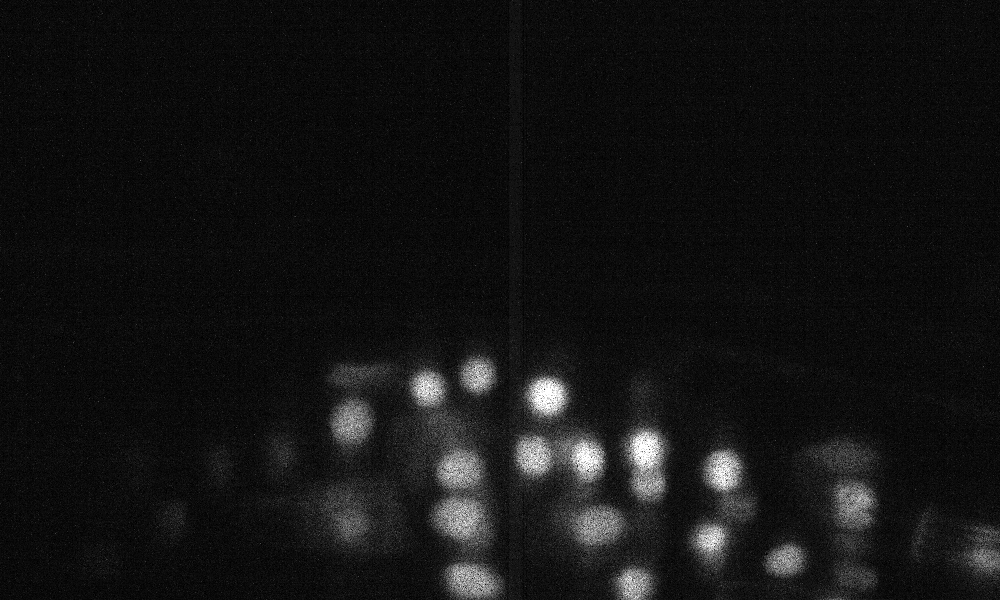

ข้อมูลสามารถดาวน์โหลดได้จาก: https://www.kaggle.com/c/data-science-bowl-2018/data
การฝึกอบรม/การคาดการณ์สามารถพบได้ใน 2DUNET_DSB2018
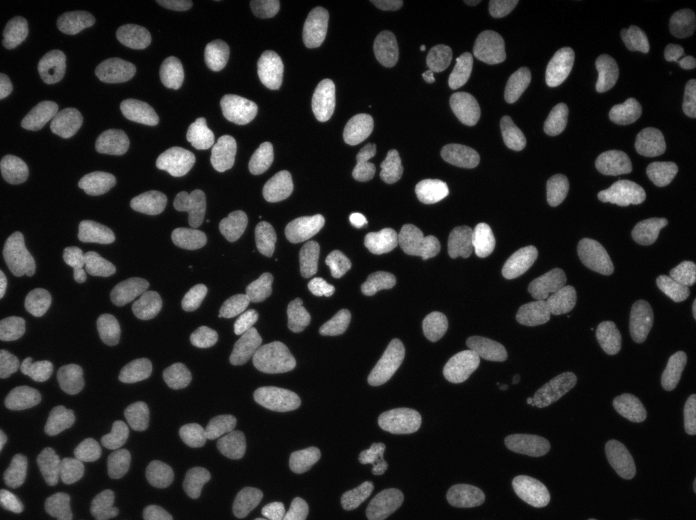
การทำนายตัวอย่างบนภาพทดสอบ (ด้านบน: อินพุตดิบด้านล่าง: การคาดการณ์ของนิวเคลียส):

หากคุณต้องการมีส่วนร่วมกลับมาโปรดทำการร้องขอการดึง
หากคุณใช้รหัสนี้สำหรับการวิจัยของคุณโปรดอ้างอิงเป็น:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}