3D U-NetとそのバリエーションのPytorch実装:
3D U-NETに基づくUNet3D標準3D U-NET:スパースアノテーションからの密な体積セグメンテーションの学習
Snemi3d Connectomics Challengeの超人的精度に基づくResidualUNet3D残差3D U-Net
ResidualUNetSE3D ResidualUNet3D 、高解像度の医療ボリュームの深い学習セマンティックセグメンテーションに基づいて、スクイーズおよび励起ブロックを追加することと同様です。オリジナルの絞りと励起紙:スクイーズアンドエクスケートネットワーク
このコードでは、セマンティックセグメンテーション(バイナリとマルチクラス)と回帰問題(例:脱線、学習デコンボリューションなど)の両方のU-NETをトレーニングできます。
2D U-NETもサポートされています。たとえば、2Dunet_Confocalまたは2Dunet_DSB2018を参照してください。データの負荷 /データの増強にはランク3のテンソルが必要であるため、H5データセット( (Y, X) (1, Y, X) (1, 3, 3)にシングルトンz次元を保持してください。
入力データはHDF5ファイルに保存する必要があります。トレーニング用のHDF5ファイルには、 rawとlabel 2つのデータセットを含める必要があります。オプションで、 PixelWiseCrossEntropyLossでトレーニングするときは、 weightデータセットを提供する必要があります。 rawデータセットには入力データが含まれ、 labelセットにはグラウンドトゥルースラベルが含まれている必要があります。オプションのweightデータセットには、入力の異なる領域で損失関数を重み付けする値を含める必要があり、 labelデータセットと同じサイズである必要があります。 raw / labelデータセットの形式は、問題が2Dまたは3Dであるかどうか、およびデータがシングルチャネルまたはマルチチャネルであるかどうかに依存します。以下の表を参照してください。
| 2d | 3D | |
|---|---|---|
| シングルチャネル | (1、y、x) | (z、y、x) |
| マルチチャネル | (c、1、y、x) | (C、Z、Y、X) |
pytorch-3dunetはクロスプラットフォームパッケージであり、WindowsとOS Xでも実行されます。
pytorch-3dunetパッケージをインストールする最も簡単な方法は、Conda/Mamba経由です。 conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
インストール後、次のコマンドにアクセスできます環境内でアクセスできます: train3dunetネットワークをトレーニングするための3Dunetと予測用のpredict3dunet (以下を参照)。
python setup.py install
インストールされているpytorch CUDAバージョンと互換性があることを確認してください。そうしないと、トレーニング/予測がGPUで実行されません。
上記のように、 pytorch-3dunetパッケージがCondaを介してインストールされたことを考えると、単に呼び出すだけでネットワークをトレーニングできます。
train3dunet --config <CONFIG>
ここでCONFIG YAML構成ファイルへのパスであり、トレーニング手順のすべての側面を指定します。
独自のデータをトレーニングするために、構成内のHDF5トレーニングと検証データセットへのパスを提供するだけです。
Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (Conda envにtensorflowがインストールされる必要があります)。CheckPoint_Dir checkpoint_dir 、構成で指定されたチェックポイントディレクトリへのパスです。
BCEWithLogitsLoss 、 DiceLoss 、 BCEDiceLoss 、 GeneralizedDiceLoss :ターゲットデータは4d(チャネルごとに1つのターゲットバイナリマスク)でなければなりません。 WeightedCrossEntropyLoss 、 CrossEntropyLoss 、 PixelWiseCrossEntropyLossでトレーニングする場合、ターゲットデータセットは3Dでなければなりません。CE損失のPytorchドキュメントも参照してください:https://pytorch.org/docs/master/generated/torch.nn.model構成セクションのfinal_sigmoid 、推論時間にのみ適用されます(検証、テスト):BCEWithLogitsLoss 、 DiceLoss 、 BCEDiceLoss 、 GeneralizedDiceLoss set final_sigmoid=TrueでトレーニングするときWeightedCrossEntropyLoss 、 CrossEntropyLoss 、 PixelWiseCrossEntropyLoss )でトレーニングする場合、 final_sigmoid=Falseを設定して、 Softmax正規化が出力に適用されるようにします。 上記のように、 pytorch-3dunetパッケージがCondaを介してインストールされたことを考えると、次の予測を実行できます。
predict3dunet --config <CONFIG>
独自のデータを予測するために、モデルへのパスとHDF5テストファイルへのパスを提供するだけです(Test_config_segmentation.yamlの例を参照)。
LazyHDF5DatasetとLazyPredictor使用を検討してください。これにより、予測時間が遅いため、データをその場でロードすることでメモリを保存します。 configの例については、test_config_lazyを参照してください。save_segmentation: true configのpredictorセクションでtrueを設定します(test_config_multiclassを参照)。 デフォルトでは、DataPar Allelを使用して、すべてのGPUで複数のGPUが利用可能な場合、トレーニング/予測が実行されます。利用可能なすべてのGPUのトレーニング/予測が望ましくない場合は、 CUDA_VISIBLE_DEVICESを使用してGPUの数を制限します。
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >または
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (バイナリクロスエントロピー)DiceLoss (バイナリセマンティックセグメンテーションに使用される1 - DiceCoefficientとして定義された標準DiceLoss 。グラウンドトゥルースに2つ以上のクラスが存在する場合、チャネルごとのDiceLossを計算し、値を平均します)BCEDiceLoss (BCEとダイス損失の線形組み合わせ、つまりalpha * BCE + beta * Dice 、 alpha, beta構成のlossセクションで指定できます)CrossEntropyLoss ( weight: [w_1, ..., w_k] configのlossセクションで)PixelWiseCrossEntropyLoss (グラウンドトゥルースの重要な/過小表現された領域により勾配を与えるためにピクセルあたりの重みを指定できます。トレーニングと検証のためにH5ファイルでweightデータセットを提供する必要があります。WeightedCrossEntropyLoss (詳細な説明については、下の論文の「加重交差点(WCE)」を参照)GeneralizedDiceLoss (詳細な説明のために下の論文の「Generalized Dice Loss(GDL)」を参照)注:トレーニングデータセットのラベルが非常に不均衡な場合にのみ、この損失関数を使用します。それ以外の場合は、標準のDiceLossを使用します。サポートされている損失関数のいくつかの詳細な説明については、一般化されたサイコロが非常に不均衡なセグメンテーションの深い学習損失関数として重複しています。
MSELoss (平均二乗誤差損失)L1Loss (平均絶対誤差損失)SmoothL1Loss (mselossよりも外れ値に敏感ではない)WeightedSmoothL1Loss (特定のしきい値の上/下にボクセル値を異なる方法で重量化できるSmoothL1Lossの拡張) MeanIoU (ユニオン上の平均交差点)DiceCoefficient (平均を返します)。以下のメトリックは、しきい値境界マップで接続されたコンポーネントを実行し、結果のインスタンスをグラウンドトゥルースインスタンスの分離と比較することによって計算されます):BoundaryAveragePrecision (境界確率マップに適用される平均精度:ネットワークからの出力をしきい値、接続されたコンポーネントを実行してセグメンテーションを取得し、結果のセグメンテーションとグラウンドトゥルースの間でAPを計算します)AdaptedRandError (詳細な説明については、http://brainiac2.mit.edu/snemi3d/evaluationを参照)AveragePrecision (https://www.kaggle.com/stkbailey/step-by-step-explanation-of-scoring-metricを参照)指定されていない場合はMeanIoUデフォルトで使用されます。
PSNR (ピーク信号対騒音比)MSE (平均二乗エラー) トレーニング/予測構成は、3Dunet_lightsheet_boundaryにあります。ここで入手可能な事前に訓練されたモデルの重み。あなた自身のデータで事前に訓練されたモデルを使用するために:
best_checkpoint.pytorchをダウンロードしますpredict3dunet --config test_config.ymlを実行しますpre_trained属性をbest_checkpoint.pytorchパスを指すように設定することにより、事前に訓練されたモデルを独自のデータで微調整しますトレーニングに使用されるデータは、次のOSFプロジェクトからダウンロードできます。
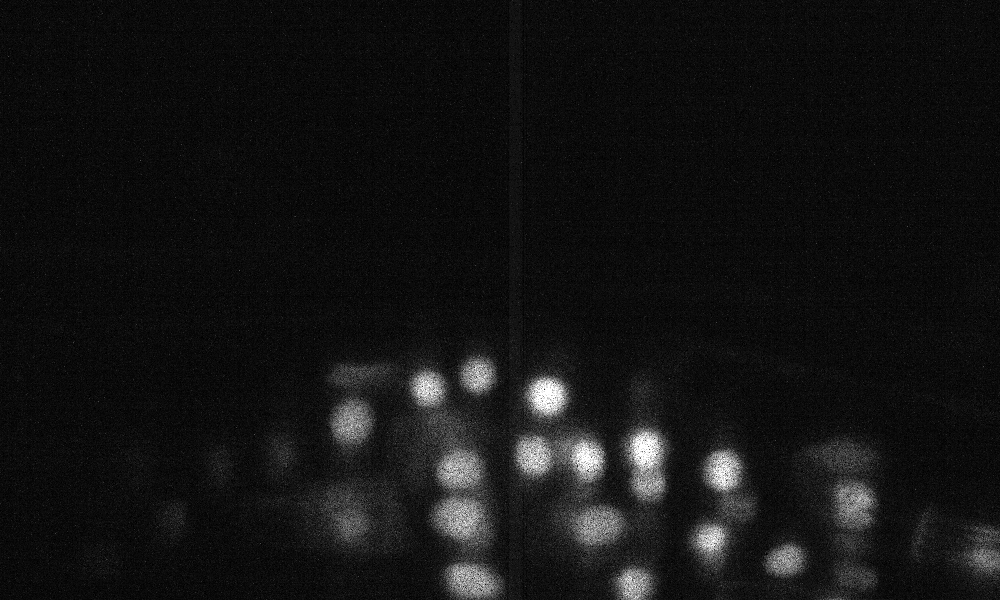
テストセットのZ-Slice予測のサンプル(TOP:RAW入力、下:境界予測):

トレーニング/予測構成は、3DUNET_CONFOCAL_BOUNDARYにあります。ここで入手可能な事前に訓練されたモデルの重み。あなた自身のデータで事前に訓練されたモデルを使用するために:
best_checkpoint.pytorchをダウンロードしますpredict3dunet --config test_config.ymlを実行しますpre_trained属性をbest_checkpoint.pytorchパスを指すように設定することにより、事前に訓練されたモデルを独自のデータで微調整しますトレーニングに使用されるデータは、次のOSFプロジェクトからダウンロードできます。
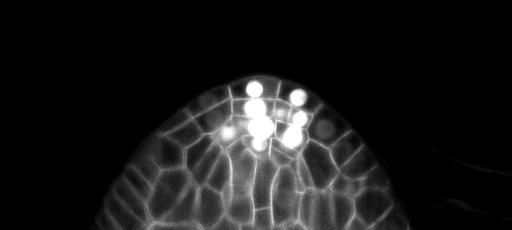
テストセットのZ-Slice予測のサンプル(TOP:RAW入力、下:境界予測):
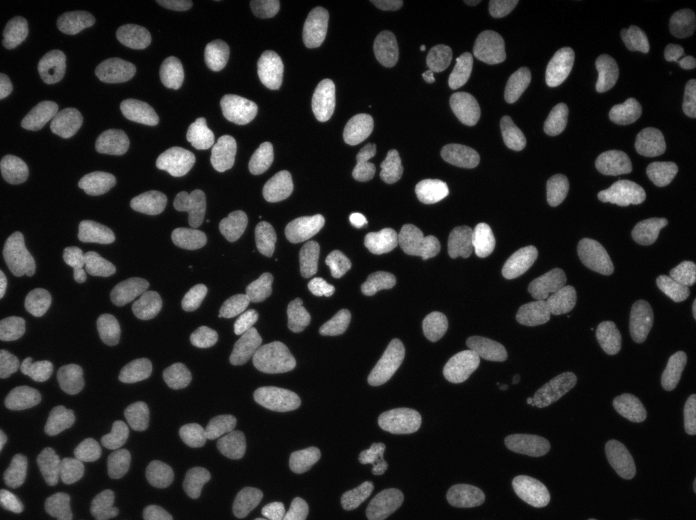
トレーニング/予測構成は、3DUNET_LIGHTSHEET_NUCLEIにあります。ここで入手可能な事前に訓練されたモデルの重み。あなた自身のデータで事前に訓練されたモデルを使用するために:
best_checkpoint.pytorchをダウンロードしますpredict3dunet --config test_config.ymlを実行しますpre_trained属性をbest_checkpoint.pytorchパスを指すように設定することにより、事前に訓練されたモデルを独自のデータで微調整しますトレーニングと検証セットは、次のOSFプロジェクトからダウンロードできます:https://osf.io/thxzn/
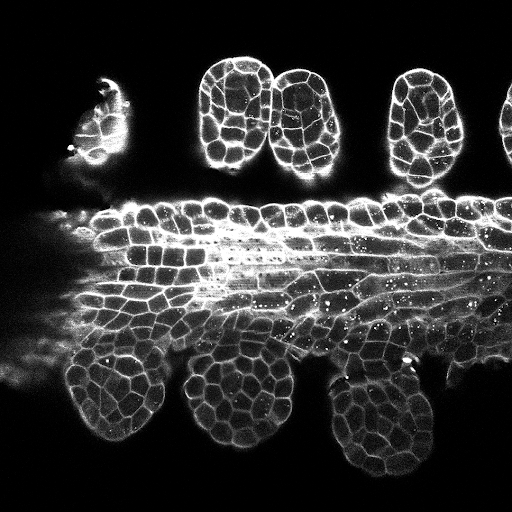
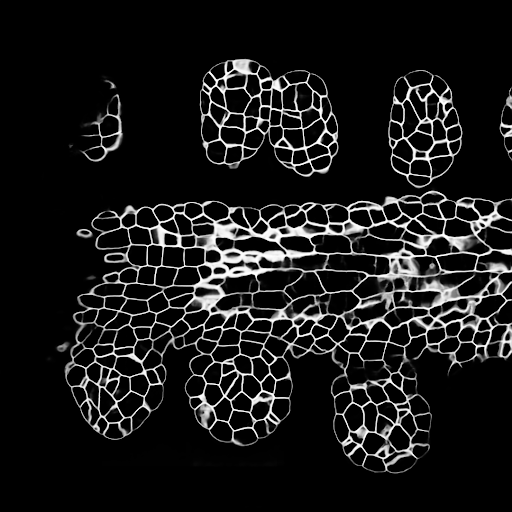
テストセットのZ-Slice予測のサンプル(TOP:RAW入力、下:核予測):

データは、https://www.kaggle.com/c/data-science-bowl-2018/dataからダウンロードできます
トレーニング/予測構成は、2DUNET_DSB2018にあります。
テスト画像のサンプル予測(TOP:raw入力、下:核予測):

貢献したい場合は、プルリクエストをお願いします。
このコードを調査に使用する場合は、次のように引用してください。
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}