Implementasi PyTorch dari 3D U-NET dan variannya:
UNet3D Standard 3D U-Net Berdasarkan 3D U-Net: Pembelajaran Segmentasi Volumetrik Dense dari Anotasi Jarang
ResidualUNet3D residual 3d u-net berdasarkan akurasi manusia super pada tantangan snemi3d connectomics
ResidualUNetSE3D mirip dengan ResidualUNet3D dengan penambahan blok pemerasan dan eksitasi berdasarkan segmentasi semantik pembelajaran mendalam untuk volume medis resolusi tinggi. Squeeze dan Kertas Bersama Asli: Jaringan Perasan dan Eksitasi
Kode ini memungkinkan untuk melatih U-NET untuk keduanya: segmentasi semantik (biner dan multi-kelas) dan masalah regresi (misalnya de-noising, pembelajaran dekonvolusi).
2D U-NET juga didukung, lihat 2Dunet_Confocal atau 2Dunet_DSB2018 misalnya konfigurasi. Pastikan untuk menyimpan singleton z-dimensi dalam dataset H5 Anda (yaitu (1, Y, X) alih-alih (Y, X) ), karena pembebanan data / augmentasi data membutuhkan tensor peringkat 3. U-Net 2D itu sendiri menggunakan kinerja 2D convolutional, bukan konvolusi 3D dengan ukuran kernel (1, 3, 3) untuk alasan kinerja.
Data input harus disimpan dalam file HDF5. File HDF5 untuk pelatihan harus berisi dua set data: raw dan label . Secara opsional, saat pelatihan dengan PixelWiseCrossEntropyLoss seseorang harus memberikan dataset weight . Dataset raw harus berisi data input, sedangkan dataset label label kebenaran ground. Dataset weight opsional harus berisi nilai untuk menimbang fungsi kerugian di berbagai daerah input dan harus dengan ukuran yang sama dengan dataset label . Format dataset raw / label tergantung pada apakah masalahnya adalah 2D atau 3D dan apakah data adalah saluran tunggal atau multi-channel, lihat tabel di bawah ini:
| 2d | 3d | |
|---|---|---|
| saluran tunggal | (1, y, x) | (Z, y, x) |
| multi-channel | (C, 1, y, x) | (C, Z, Y, X) |
pytorch-3dunet adalah paket lintas platform dan berjalan di Windows dan OS X juga.
pytorch-3dunet adalah melalui conda/mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
Setelah instalasi, perintah -perintah berikut dapat diakses dalam lingkungan Conda: train3dunet untuk melatih jaringan dan predict3dunet untuk prediksi (lihat di bawah).
python setup.py install
Pastikan pytorch yang diinstal kompatibel dengan versi CUDA Anda, jika tidak, pelatihan/prediksi akan gagal berjalan pada GPU.
Mengingat bahwa paket pytorch-3dunet diinstal melalui conda seperti dijelaskan di atas, seseorang dapat melatih jaringan dengan hanya memohon:
train3dunet --config <CONFIG>
di mana CONFIG adalah jalur ke file konfigurasi YAML, yang menentukan semua aspek prosedur pelatihan.
Untuk melatih data Anda sendiri, hanya menyediakan jalur ke pelatihan HDF5 dan set data validasi di konfigurasi.
Seseorang dapat memantau kemajuan pelatihan dengan Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (Anda perlu tensorflow yang diinstal di conda env Anda), di mana checkpoint_dir adalah jalur ke direktori pos pemeriksaan yang ditentukan dalam konfigurasi.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : data target harus 4D (satu mask biner target per saluran). When training with WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss the target dataset has to be 3D, see also pytorch documentation for CE loss: https://pytorch.org/docs/master/generated/torch.nn.CrossEntropyLoss.htmlfinal_sigmoid di bagian model Config hanya berlaku untuk waktu inferensi (validasi, tes):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) mengatur final_sigmoid=False sehingga normalisasi Softmax diterapkan pada output. Mengingat bahwa paket pytorch-3dunet diinstal melalui conda seperti dijelaskan di atas, seseorang dapat menjalankan prediksi melalui:
predict3dunet --config <CONFIG>
Untuk memprediksi data Anda sendiri, cukup berikan jalur ke model Anda serta jalur ke file uji HDF5 (lihat contoh test_config_segmentation.yaml).
LazyHDF5Dataset dan LazyPredictor di konfigurasi. Ini akan menghemat memori dengan memuat data dengan cepat dengan biaya waktu prediksi yang lebih lambat. Lihat test_config_lazy untuk konfigurasi contoh.save_segmentation: true di bagian predictor konfigurasi (lihat test_config_multiclass). Secara default, jika beberapa GPU tersedia pelatihan/prediksi akan dijalankan pada semua GPU menggunakan dataparallel. Jika pelatihan/prediksi pada semua GPU yang tersedia tidak diinginkan, batasi jumlah GPU menggunakan CUDA_VISIBLE_DEVICES , misalnya
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >atau
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (Biner Cross-Encropy)DiceLoss ( DiceLoss standar yang didefinisikan sebagai 1 - DiceCoefficient yang digunakan untuk segmentasi semantik biner; ketika lebih dari 2 kelas hadir dalam kebenaran dasar, ia menghitung DiceLoss per saluran dan rata -rata nilai -nilai)BCEDiceLoss (kombinasi linear dari kerugian bce dan dadu, yaitu alpha * BCE + beta * Dice , alpha, beta dapat ditentukan di bagian loss konfigurasi)CrossEntropyLoss (seseorang dapat menentukan bobot kelas melalui weight: [w_1, ..., w_k] Di bagian loss konfigurasi)PixelWiseCrossEntropyLoss (seseorang dapat menentukan bobot per-piksel untuk memberikan lebih banyak gradien ke daerah penting/kurang terwakili dalam kebenaran tanah; dataset weight harus disediakan dalam file H5 untuk pelatihan dan validasi; lihat Contoh Konfigurasi di train_config.ymlWeightedCrossEntropyLoss (lihat 'Weighted Cross-Enropy (WCE)' di dalam kertas di bawah ini untuk penjelasan terperinci)GeneralizedDiceLoss (lihat 'Generalized Dice Loss (GDL)' dalam makalah di bawah ini untuk penjelasan terperinci) CATATAN: Gunakan fungsi kerugian ini hanya jika label dalam dataset pelatihan sangat tidak seimbang misalnya satu kelas yang memiliki setidaknya 3 urutan besarnya voxel daripada yang lain. Jika tidak, gunakan DiceLoss standar.Untuk penjelasan terperinci tentang beberapa fungsi kerugian yang didukung, lihat: Dadu umum tumpang tindih sebagai fungsi kehilangan pembelajaran yang mendalam untuk segmentasi yang sangat tidak seimbang.
MSELoss (rata -rata kehilangan kesalahan kuadrat)L1Loss (kerugian kesalahan absolut)SmoothL1Loss (kurang sensitif terhadap outlier daripada Mseloss)WeightedSmoothL1Loss (ekstensi SmoothL1Loss yang memungkinkan untuk menimbang nilai voxel di atas/di bawah ambang batas yang diberikan secara berbeda) MeanIoU (persimpangan rata -rata atas persatuan)DiceCoefficient (menghitung koefisien dadu per saluran dan mengembalikan rata-rata) jika 3D U-NET dilatih untuk memprediksi batas sel, orang dapat menggunakan metrik segmentasi instance semantik berikut (metrik di bawah ini dihitung dengan menjalankan komponen yang terhubung pada peta batas ambang batas dan membandingkan instance yang dihasilkan dengan segmen kebenaran ground):BoundaryAveragePrecision (presisi rata -rata diterapkan pada peta probabilitas batas: ambang batas output dari jaringan, menjalankan komponen yang terhubung untuk mendapatkan segmentasi dan menghitung AP antara segmentasi yang dihasilkan dan kebenaran dasar)AdaptedRandError (lihat http://brainiac2.mit.edu/snemi3d/evaluasi untuk penjelasan terperinci)AveragePrecision (lihat https://www.kaggle.com/stkbailey/step-by-step-explanation-of-scoring-metric) Jika tidak ditentukan MeanIoU akan digunakan secara default.
PSNR (rasio sinyal puncak ke noise)MSE (rata -rata kesalahan kuadrat) Konfigurasi pelatihan/prediksi dapat ditemukan di 3dunet_lightsheet_boundary. Bobot model pra-terlatih tersedia di sini. Untuk menggunakan model pra-terlatih pada data Anda sendiri:
best_checkpoint.pytorch dari tautan di ataspredict3dunet --config test_config.ymlpre_trained di konfigurasi yaml untuk menunjuk ke jalur best_checkpoint.pytorchData yang digunakan untuk pelatihan dapat diunduh dari proyek OSF berikut:
Sampel prediksi z-slice pada set tes (atas: input mentah, bawah: prediksi batas):
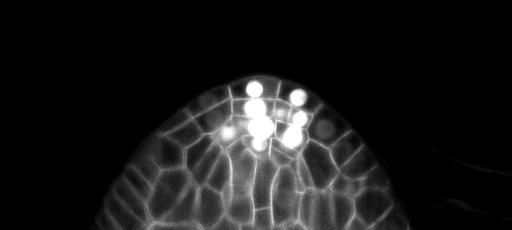

Konfigurasi pelatihan/prediksi dapat ditemukan di 3dunet_confocal_boundary. Bobot model pra-terlatih tersedia di sini. Untuk menggunakan model pra-terlatih pada data Anda sendiri:
best_checkpoint.pytorch dari tautan di ataspredict3dunet --config test_config.ymlpre_trained di konfigurasi yaml untuk menunjuk ke jalur best_checkpoint.pytorchData yang digunakan untuk pelatihan dapat diunduh dari proyek OSF berikut:
Sampel prediksi z-slice pada set tes (atas: input mentah, bawah: prediksi batas):
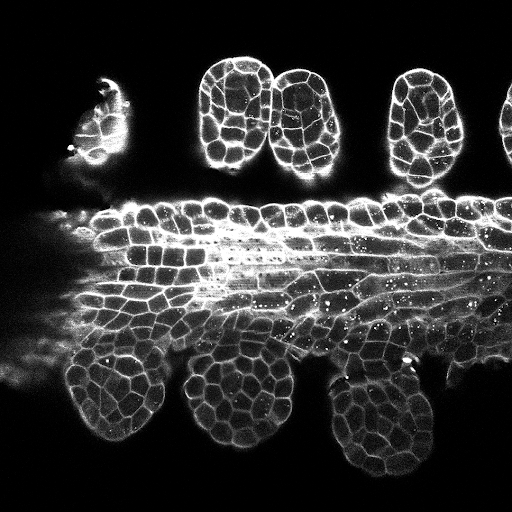
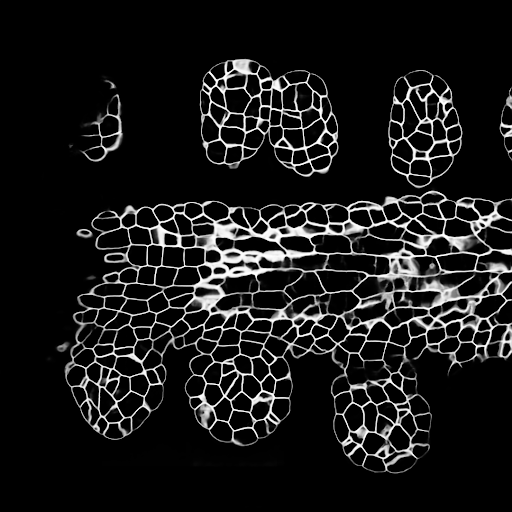
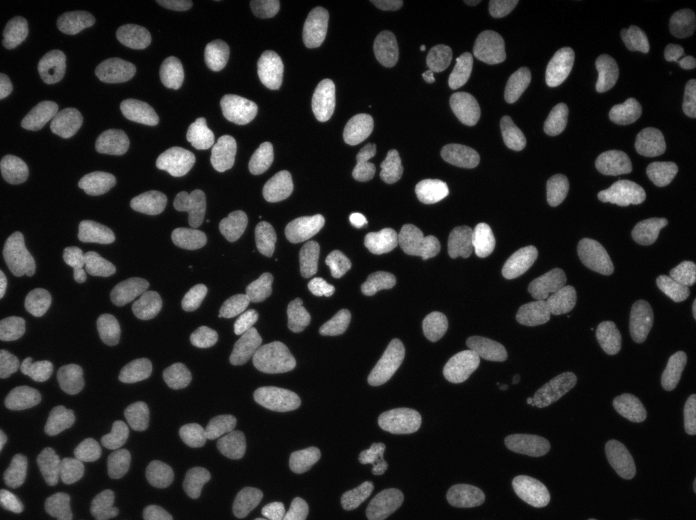
Konfigurasi pelatihan/prediksi dapat ditemukan di 3dunet_lightsheet_nuclei. Bobot model pra-terlatih tersedia di sini. Untuk menggunakan model pra-terlatih pada data Anda sendiri:
best_checkpoint.pytorch dari tautan di ataspredict3dunet --config test_config.ymlpre_trained di konfigurasi yaml untuk menunjuk ke jalur best_checkpoint.pytorchSet pelatihan dan validasi dapat diunduh dari proyek OSF berikut: https://osf.io/thxzn/
Sampel prediksi z-slice pada set uji (atas: input mentah, bawah: prediksi nuklei):
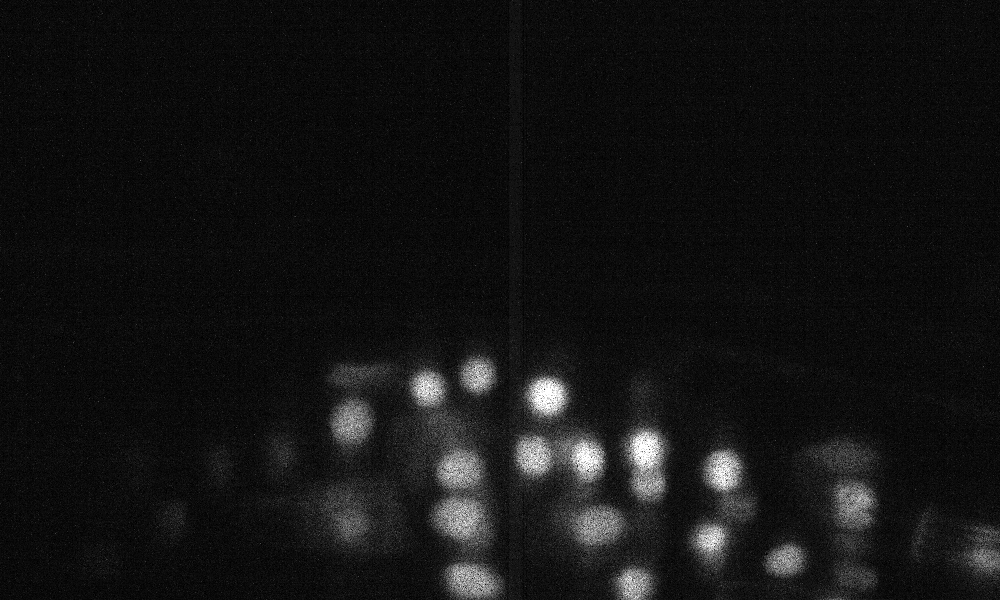

Data dapat diunduh dari: https://www.kaggle.com/c/data-cience-bowl-2018/data
Konfigurasi pelatihan/prediksi dapat ditemukan di 2Dunet_DSB2018.
Prediksi sampel pada gambar uji (atas: input mentah, bawah: prediksi nuklei):

Jika Anda ingin berkontribusi kembali, silakan buat permintaan tarik.
Jika Anda menggunakan kode ini untuk penelitian Anda, silakan kutip sebagai:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}