Implémentation Pytorch de 3D U-Net et de ses variantes:
UNet3D Standard 3D U-Net basé sur 3D U-Net: Apprentissage de la segmentation volumétrique dense de l'annotation clairsemée
ResidualUNet3D résiduel 3d U-Net basé sur la précision surhumaine du défi SNEMI3D Connectomics
ResidualUNetSE3D similaire à ResidualUNet3D avec l'ajout de blocs de compression et d'excitation basés sur la segmentation sémantique d'apprentissage en profondeur pour les volumes médicaux à haute résolution. Papier d'origine et de papier excité: réseaux de compression et d'excitation
Le code permet de former le U-NET pour les deux: les problèmes de segmentation sémantique (binaires et multi-classes) et de régression (par exemple, la décontention, les déconvolutions d'apprentissage).
UN-NET 2D est également pris en charge, voir 2Dunet_Confocal ou 2Dunet_DSB2018 Par exemple Configuration. Assurez-vous simplement de conserver la dimension Z Singleton dans votre ensemble de données H5 (c'est-à-dire (1, Y, X) au lieu de (Y, X) ), car l'augmentation des données / les données nécessite des tenseurs du rang 3. Le 2D U-Net lui-même utilise les couches convolutionnelles 2D standard au lieu de convolutions 3D avec la taille du noyau (1, 3, 3) pour des raisons de performance.
Les données d'entrée doivent être stockées dans des fichiers HDF5. Les fichiers HDF5 pour la formation doivent contenir deux ensembles de données: raw et label . Facultativement, lors de l'entraînement avec PixelWiseCrossEntropyLoss il faut fournir un ensemble de données weight . L'ensemble de données raw doit contenir les données d'entrée, tandis que l'ensemble de données label les étiquettes de vérité au sol. L'ensemble de données weight en option doit contenir les valeurs de pondération de la fonction de perte dans différentes régions de l'entrée et doit être de la même taille que l'ensemble de données label . Le format des ensembles de données raw / label dépend de la question de savoir si le problème est 2D ou 3D et si les données sont à canal unique ou multicanal, voir le tableau ci-dessous:
| 2d | 3D | |
|---|---|---|
| canal unique | (1, y, x) | (Z, y, x) |
| à canaux multiples | (C, 1, y, x) | (C, Z, Y, X) |
pytorch-3dunet est un package multiplateforme et s'exécute également sur Windows et OS X.
pytorch-3dunet est via Conda / Mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
Après l'installation, les commandes suivantes sont accessibles dans l'environnement Conda: train3dunet pour la formation du réseau et predict3dunet pour la prédiction (voir ci-dessous).
python setup.py install
Assurez-vous que le pytorch installé est compatible avec votre version CUDA, sinon la formation / prédiction ne parviendra pas à fonctionner sur GPU.
Étant donné que le package pytorch-3dunet a été installé via Conda comme décrit ci-dessus, on peut former le réseau en invoquant simplement:
train3dunet --config <CONFIG>
où CONFIG est le chemin d'accès à un fichier de configuration YAML, qui spécifie tous les aspects de la procédure de formation.
Afin de s'entraîner sur vos propres données, fournissez simplement les chemins de données de vos ensembles de données de formation et de validation HDF5 dans la configuration.
On peut surveiller la progression de la formation avec Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (vous avez besoin tensorflow installé dans votre conda Env), où checkpoint_dir est le chemin du répertoire de contrôle spécifié dans la configuration.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : les données cibles doivent être 4D (un masque binaire cible par canal). Lors de l'entraînement avec WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss L'ensemble de données cible doit être 3D, voir également la documentation Pytorch pour la perte de CE: https://pytorch.org/docs/master/generated/torch.nn.crossentropyloss.htmlfinal_sigmoid dans la section de configuration model ne s'applique qu'au temps d'inférence (validation, test):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) Définit final_sigmoid=False pour que la normalisation Softmax soit appliquée à la sortie. Étant donné que le package pytorch-3dunet a été installé via Conda comme décrit ci-dessus, on peut exécuter la prédiction via:
predict3dunet --config <CONFIG>
Afin de prédire sur vos propres données, fournissez simplement le chemin d'accès à votre modèle ainsi que des chemins de chemin vers les fichiers de test HDF5 (voir Exemple Test_config_segmentation.yaml).
LazyHDF5Dataset et LazyPredictor dans la configuration. Cela économisera la mémoire en chargeant des données à la volée au prix du temps de prédiction plus lent. Voir test_config_lazy pour un exemple de configuration.save_segmentation: true dans la section predictor de la configuration (voir test_config_multiclass). Par défaut, si plusieurs GPU sont disponibles, une formation / prédiction sera exécutée sur tous les GPU à l'aide de DataParallel. Si la formation / la prédiction sur tous les GPU disponibles n'est pas souhaitable, limitez le nombre de GPU à l'aide de CUDA_VISIBLE_DEVICES , par exemple
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >ou
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (entropie croisée binaire)DiceLoss ( DiceLoss standard défini comme 1 - DiceCoefficient utilisé pour la segmentation sémantique binaire; lorsque plus de 2 classes sont présentes dans la vérité du sol, il calcule le DiceLoss par canal et fait les moyennes des valeurs)BCEDiceLoss (combinaison linéaire des pertes de BCE et de dés, c'est-à-dire alpha * BCE + beta * Dice , alpha, beta peut être spécifié dans la section loss de la configuration)CrossEntropyLoss (on peut spécifier des poids de classe via le weight: [w_1, ..., w_k] dans la section loss de la configuration)PixelWiseCrossEntropyLoss (on peut spécifier des poids par pixel afin de donner plus de gradient aux régions importantes / sous-représentées dans la vérité du sol; l'ensemble de données weight doit être fourni dans les fichiers H5 pour la formation et la validation; voir l'échantillon de configuration dans Train_config.ymlWeightedCrossEntropyLoss (voir «Entropie croisée pondérée (WCE)» dans le document ci-dessous pour une explication détaillée)GeneralizedDiceLoss (voir 'Generalized Dice Loss (GDL)' dans le document ci-dessous pour une explication détaillée) Remarque: Utilisez cette fonction de perte uniquement si les étiquettes de l'ensemble de données de formation sont très déséquilibrées, par exemple une classe ayant au moins 3 ordres de grandeur de voxels que les autres. Sinon, utilisez DiceLoss standard.Pour une explication détaillée de certaines des fonctions de perte prise en charge, consultez: le chevauchement des dés généralisés en tant que fonction de perte d'apprentissage en profondeur pour les segmentations très déséquilibrées.
MSELoss (perte d'erreur carrée moyenne)L1Loss (perte d'erreur absolue moyenne)SmoothL1Loss (moins sensible aux valeurs aberrantes que mseloss)WeightedSmoothL1Loss (extension du SmoothL1Loss qui permet de pondérer les valeurs de voxel supérieures / inférieures à un seuil donné différemment) MeanIoU (intersection moyenne sur l'union)DiceCoefficient (calcul par coefficient de dés à canal et renvoie la moyenne) Si un réseau U 3D a été formé pour prédire les limites des cellules, on peut utiliser les mesures de segmentation des instances sémantiques suivantes (les métriques ci-dessous sont calculées en exécutant les composants connectés sur la carte limite de seuil et en comparant les instances qui en résultent à la segmentation des instances de vérité du sol):BoundaryAveragePrecision (précision moyenne appliquée aux cartes de probabilité des limites: seuils la sortie du réseau, exécute les composants connectés pour obtenir la segmentation et calcule AP entre la segmentation résultante et la vérité au sol)AdaptedRandError (voir http://brainiac2.mit.edu/snemi3d/évaliation pour une explication détaillée)AveragePrecision (voir https://www.kaggle.com/stkbailey/step-by-tep-explanation-of-scoring-metric) Si ce n'est pas spécifié, MeanIoU sera utilisé par défaut.
PSNR (rapport de signal de bruit / bruit)MSE (erreur carrée moyenne) Les configurations de formation / prédictions peuvent être trouvées dans 3Dunet_lightSheet_Boundary. Poids du modèle pré-formés disponibles ici. Afin d'utiliser le modèle pré-formé sur vos propres données:
best_checkpoint.pytorch à partir du lien ci-dessuspredict3dunet --config test_config.ymlpre_trained dans la configuration YAML pour pointer le chemin best_checkpoint.pytorchLes données utilisées pour la formation peuvent être téléchargées à partir du projet OSF suivant:
Échantillon de prédictions de coupe Z sur l'ensemble de tests (en haut: entrée brute, en bas: prédictions limites):

Les configurations de formation / prédictions peuvent être trouvées dans 3DUNET_CONFOCAL_BOUNDARY. Poids du modèle pré-formés disponibles ici. Afin d'utiliser le modèle pré-formé sur vos propres données:
best_checkpoint.pytorch à partir du lien ci-dessuspredict3dunet --config test_config.ymlpre_trained dans la configuration YAML pour pointer le chemin best_checkpoint.pytorchLes données utilisées pour la formation peuvent être téléchargées à partir du projet OSF suivant:
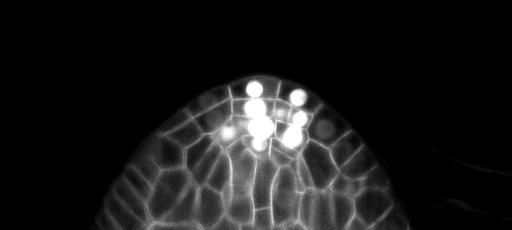
Échantillon de prédictions de coupe Z sur l'ensemble de tests (en haut: entrée brute, en bas: prédictions limites):
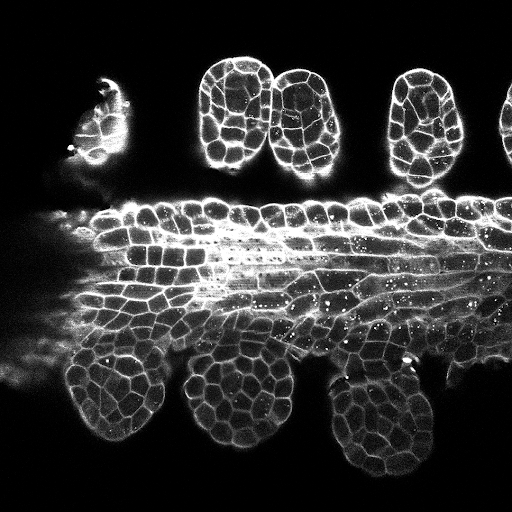
Les configurations de formation / prédictions peuvent être trouvées dans 3Dunet_lightheet_nuclei. Poids du modèle pré-formés disponibles ici. Afin d'utiliser le modèle pré-formé sur vos propres données:
best_checkpoint.pytorch à partir du lien ci-dessuspredict3dunet --config test_config.ymlpre_trained dans la configuration YAML pour pointer le chemin best_checkpoint.pytorchLes ensembles de formation et de validation peuvent être téléchargés à partir du projet OSF suivant: https://osf.io/thxzn/
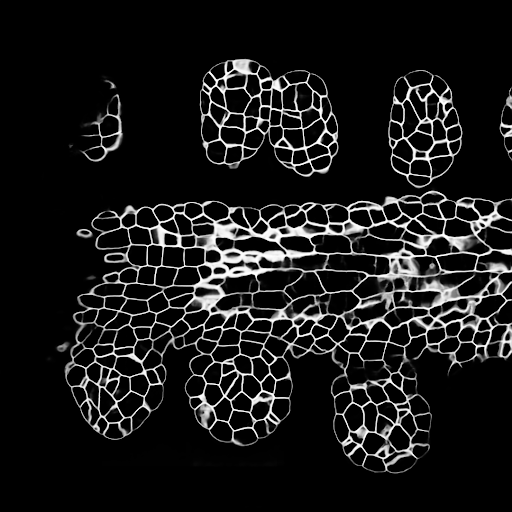
Échantillon de prédictions de lileur z sur l'ensemble de tests (en haut: entrée brute, en bas: prédictions des noyaux):
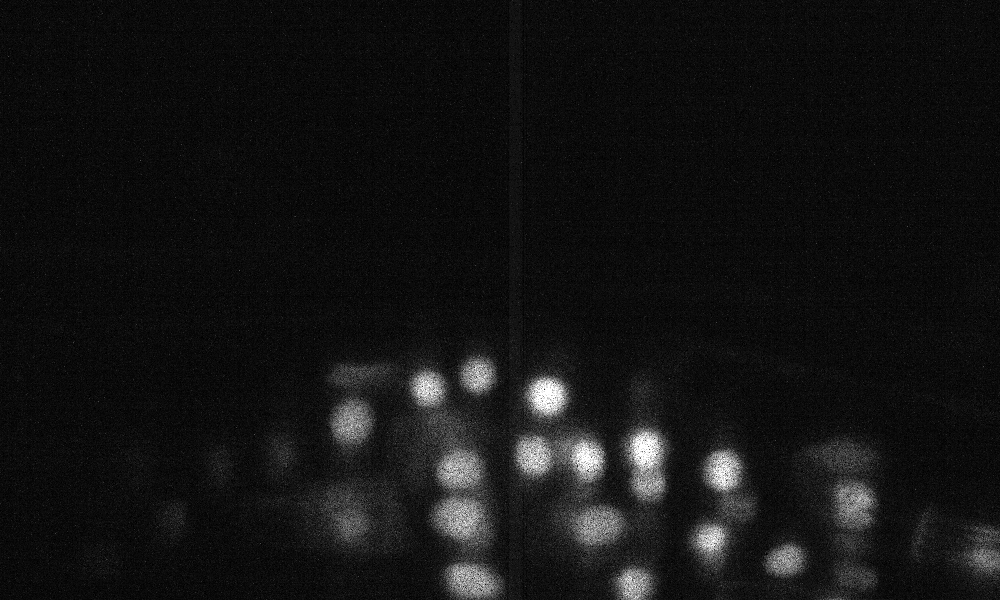

Les données peuvent être téléchargées à partir de: https://www.kaggle.com/c/data-science-bowl-2018/data
Les configurations de formation / prédictions peuvent être trouvées dans 2DUNET_DSB2018.
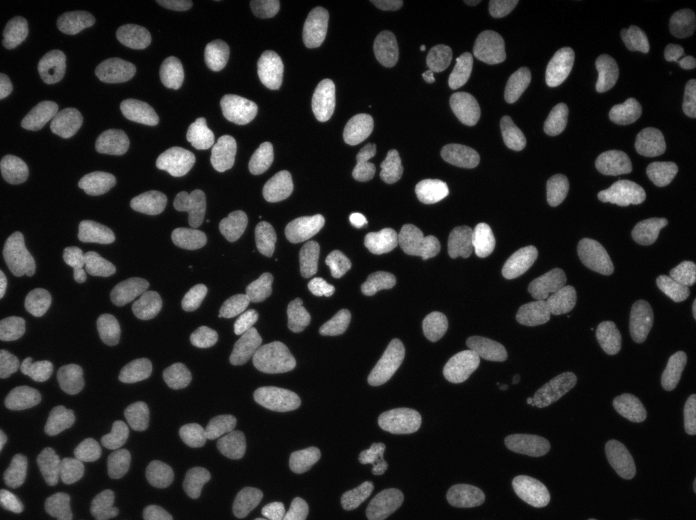
Exemples de prédictions sur l'image de test (en haut: entrée brute, en bas: prédictions des noyaux):

Si vous souhaitez contribuer, veuillez faire une demande de traction.
Si vous utilisez ce code pour vos recherches, veuillez citer comme:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}