Реализация Pytorch 3D U-Net и его варианты:
UNet3D Стандартный 3D U-Net на основе 3D U-Net: Learning Плотная объемная сегментация от разреженной аннотации
ResidualUNet3D 3D-сеть остаточных 3D на основе сверхчеловеческой точности на вызове SNEMI3D Connectomics Challenge
ResidualUNetSE3D аналогичный ResidualUNet3D , с добавлением сжимания и возбуждения, основанных на семантической сегментации глубокого обучения для медицинских объемов с высоким разрешением. Оригинальная сжимания и экипированная бумага: сетки сжимания и сжимания
Код позволяет обучать U-Net как: семантическую сегментацию (бинарную и многоклассу), так и проблемы регрессии (например, деконволюции обучения).
2D U-NET также поддерживается, см. 2DUNET_CONFOCAL или 2DUNET_DSB2018, например, конфигурация. Просто убедитесь, что сохраните одноэлектрон Z-измерение в вашем наборе данных H5 (то есть (1, Y, X) вместо (Y, X) ), потому что загрузка данных / увеличение данных требует тензоров ранга 3. Сам 2D U-Net использует стандартные 2D-слои, а не из 3D-совет с размером ядра (1, 3, 3) для производительности.
Входные данные должны храниться в файлах HDF5. Файлы HDF5 для обучения должны содержать два набора данных: raw и label . Необязательно, при обучении с PixelWiseCrossEntropyLoss следует предоставить набор данных weight . Набор данных raw должен содержать входные данные, в то время как набор данных label на метках наземных меток истины. Необязательный набор данных weight должен содержать значения для взвешивания функции потерь в разных областях ввода и должен быть одинакового размера, что и набор данных label . Формат наборов данных raw / label зависит от того, является ли проблема 2D или 3D и является ли данные одноканальным или многоканальным, см. В таблице ниже:
| 2d | 3d | |
|---|---|---|
| одноканальный | (1, y, x) | (Z, y, x) |
| многоканальный | (C, 1, Y, X) | (C, Z, Y, X) |
pytorch-3dunet -это кроссплатформенный пакет, который также работает в Windows и OS X.
pytorch-3dunet -через Conda/Mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
После установки следующие команды доступны в среде Conda: train3dunet для обучения сети и predict3dunet для прогнозирования (см. Ниже).
python setup.py install
Убедитесь, что установленная pytorch совместима с вашей версией CUDA, в противном случае обучение/прогноз не будет работать на GPU.
Учитывая, что пакет pytorch-3dunet был установлен через Conda, как описано выше, можно обучить сеть, просто вызывая:
train3dunet --config <CONFIG>
где CONFIG - это путь к файлу конфигурации YAML, который указывает все аспекты процедуры обучения.
Для обучения на своих собственных данных просто предоставьте пути к вашим наборам данных обучения и проверки HDF5 в конфигурации.
Можно отслеживать прогресс обучения с помощью Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (вам нужен tensorflow установленная в вашей конфинировании), где checkpoint_dir -это путь к каталогу контрольной точки, указанной в конфигурации.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : целевые данные должны быть 4D (одна целевая бинарная маска на канал). При тренировке с WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss целевой набор данных должен быть 3d, см. Также документацию Pytorch для потери CE: https://pytorch.org/docs/master/generated/torch.nn.crossentropyloss.htmlfinal_sigmoid в разделе конфигурации model применяется только к времени вывода (валидация, тест):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) установите final_sigmoid=False , чтобы нормализация Softmax применяется к выходу. Учитывая, что пакет pytorch-3dunet был установлен через Conda, как описано выше, можно запустить прогноз через:
predict3dunet --config <CONFIG>
Чтобы предсказать на своих собственных данных, просто предоставьте путь к вашей модели, а также пути к тестовым файлам HDF5 (см. Пример test_config_segmation.yaml).
LazyHDF5Dataset и LazyPredictor в конфигурации. Это сохранит память, загрузив данные на лету за счет более медленного времени прогноза. См. Test_config_lazy для примера config.save_segmentation: true в разделе predictor конфигурации» (см. Test_config_multiclass). По умолчанию, если несколько графических процессоров будут доступны для обучения/прогнозирования на всех графических процессорах, используя DataParallel. Если обучение/прогноз по всем доступным графическим процессорам не желательна, ограничить количество графических процессоров с помощью CUDA_VISIBLE_DEVICES , например,
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >или
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (бинарная перекрестная энтропия)DiceLoss (стандартный DiceLoss определяемый как 1 - DiceCoefficient используемый для бинарной семантической сегментации; когда в наземной истине присутствует более 2 классов, он вычисляет DiceLoss на канал и усредняет значения)BCEDiceLoss (линейная комбинация потерь BCE и DICE, то есть alpha * BCE + beta * Dice , alpha, beta может быть указана в разделе loss конфигурации)CrossEntropyLoss (можно указать веса класса с помощью weight: [w_1, ..., w_k] в разделе loss конфигурации)PixelWiseCrossEntropyLoss (можно указать веса для пикселя, чтобы придать больший градиент для важных/недостаточно представленных областей в наземной истине; набор weight должен быть предоставлен в файлах H5 для обучения и валидации; см. Пример конфигурации в Train_config.yml.WeightedCrossEntropyLoss (см. «Взвешенная перекрестная энтропия (WCE)» в приведенной ниже бумаге для подробного объяснения)GeneralizedDiceLoss (см. «Обобщенная потеря кости (GDL)» в приведенной ниже статье для подробного объяснения). Примечание: используйте эту функцию потерь только в том случае, если этикетки в наборе обучения очень дисбалансированы, например, один класс, имеющий как минимум на 3 порядка больше вокселей, чем другие. В противном случае используйте стандартный DiceLoss .Для получения подробного объяснения некоторых поддерживаемых функций потерь см.: Обобщенное перекрытие костей в качестве глубокой функции потери обучения для сильно несбалансированных сегментаций.
MSELoss (средняя потери ошибок в квадрате)L1Loss (средняя абсолютная потеря ошибок)SmoothL1Loss (менее чувствительный к выбросам, чем Mseloss)WeightedSmoothL1Loss (расширение SmoothL1Loss , которое позволяет взвесить значения вокселя выше/ниже данного порога по -разному) MeanIoU (среднее пересечение над союзом)DiceCoefficient (вычисляет коэффициент на канал коэффициент кости и возвращает среднее). Если 3D U-NET был обучен для прогнозирования границ ячеек, можно использовать следующие показатели сегментации семантических экземпляров (приведенные ниже показатели вычисляются путем запуска подключенных компонентов на карте пороговой границы и сравнивая полученные экземпляры с сегментацией основы истины):BoundaryAveragePrecision (средняя точность, применяемая к картам вероятности границ: пороговые значения вывод из сети, запускает подключенные компоненты, чтобы получить сегментацию и вычислять AP между результирующей сегментацией и основой истиной)AdaptedRandError (см. Http://brainiac2.mit.edu/snemi3d/evaluation для подробного объяснения)AveragePrecision (см. Если не указано, MeanIoU будет использоваться по умолчанию.
PSNR (пиковое отношение сигнала к шуму)MSE (средняя квадратная ошибка) Обучение/прогнозы конфигурации можно найти в 3dunet_lightsheet_boundary. Предварительно обученные веса модели доступны здесь. Чтобы использовать предварительно обученную модель на ваших собственных данных:
best_checkpoint.pytorch по ссылке вышеpredict3dunet --config test_config.ymlpre_trained атрибут в конфигурации YAML, чтобы указать на путь best_checkpoint.pytorchДанные, используемые для обучения, могут быть загружены из следующего проекта OSF:
Образец z-Slice прогнозы на тестовом наборе (вверху: необработанный вход, внизу: граничные прогнозы):
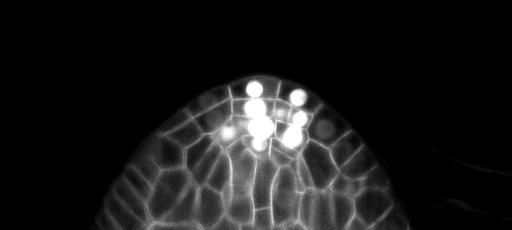

Конфигурации обучения/прогнозов можно найти в 3dunet_confocal_boundary. Предварительно обученные веса модели доступны здесь. Чтобы использовать предварительно обученную модель на ваших собственных данных:
best_checkpoint.pytorch по ссылке вышеpredict3dunet --config test_config.ymlpre_trained атрибут в конфигурации YAML, чтобы указать на путь best_checkpoint.pytorchДанные, используемые для обучения, могут быть загружены из следующего проекта OSF:
Образец z-Slice прогнозы на тестовом наборе (вверху: необработанный вход, внизу: граничные прогнозы):
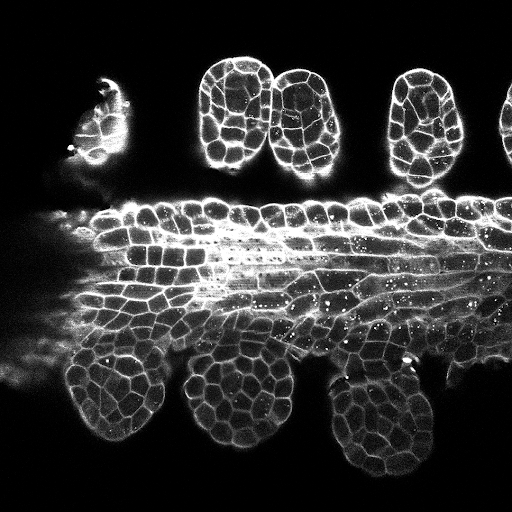
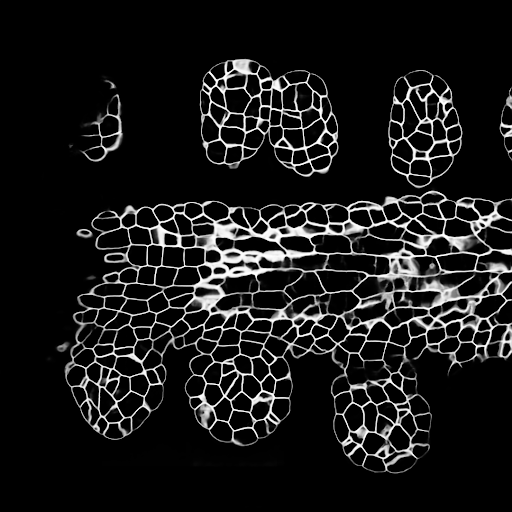
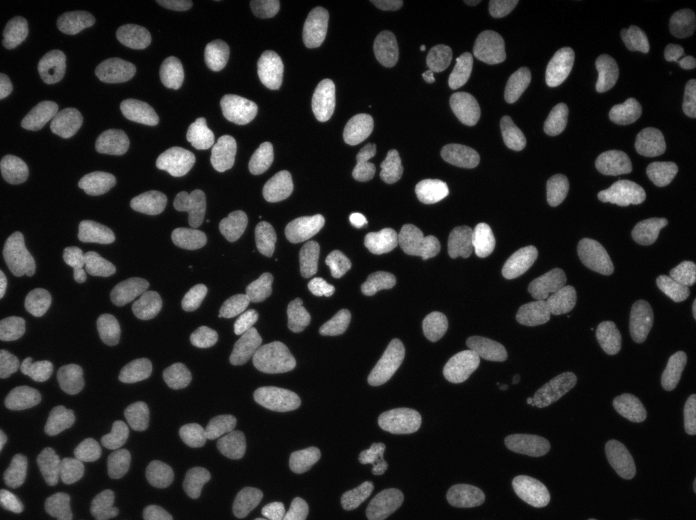
Конфигурации обучения/прогнозов можно найти в 3dunet_lightsheet_nuclei. Предварительно обученные веса модели доступны здесь. Чтобы использовать предварительно обученную модель на ваших собственных данных:
best_checkpoint.pytorch по ссылке вышеpredict3dunet --config test_config.ymlpre_trained атрибут в конфигурации YAML, чтобы указать на путь best_checkpoint.pytorchНаборы обучения и проверки могут быть загружены из следующего проекта OSF: https://osf.io/thxzn/
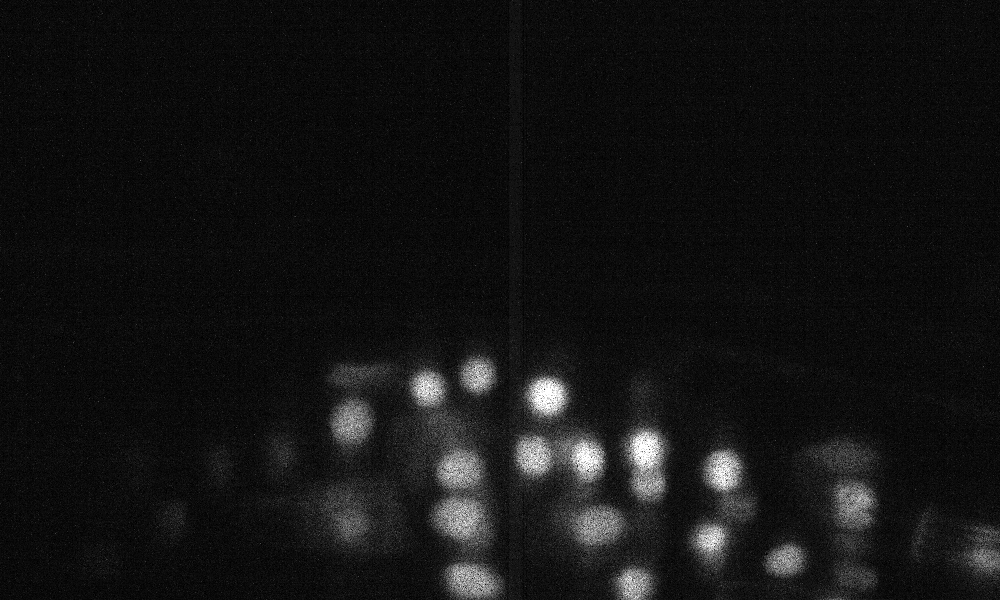
Образец z-Slice прогнозы на наборе тестирования (вверху: необработанный вход, внизу: прогнозы ядер):

Данные можно загрузить с: https://www.kaggle.com/c/data-comance-bowl-2018/data
Конфигурации обучения/прогнозов можно найти в 2DUNET_DSB2018.
Пример прогнозы на тестовом изображении (вверху: необработанный вход, внизу: прогнозы ядер):

Если вы хотите внести свой вклад, пожалуйста, сделайте запрос на тягу.
Если вы используете этот код для своего исследования, пожалуйста, цитируйте как:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}