Implementação de Pytorch da rede U 3D e suas variantes:
Net 3D padrão UNet3D Baseado na rede U 3D: Aprendendo segmentação volumétrica densa da anotação esparsa
ResidualUNet3D residual 3D-NET baseado na precisão sobre-humana no SNEMI3D Connectomics Challenge
ResidualUNetSE3D semelhante ao ResidualUNet3D com a adição de bloqueios de aperto e excitação com base na segmentação semântica de aprendizado profundo para volumes médicos de alta resolução. Squeeze e papel excitado: redes de aperto e excitação
O código permite treinar a rede U para ambos: segmentação semântica (problemas binários e multi-classes) e de regressão (por exemplo, desmontagem, desconvoluções de aprendizado).
O 2D U-Net também é suportado, consulte 2dunet_confocal ou 2dunet_dsb2018, por exemplo, configuração. Apenas certifique-se de manter a dimensão z Singleton no seu conjunto de dados H5 (ou seja (1, Y, X) em vez de (Y, X) ), porque o carregamento / aumento de dados requer tensores da classificação 3. O próprio 2D-NET usa as camadas convolucionais 2D padrão em vez de Convolutions 3D com tamanho de kernel (1, 3, 3) .
Os dados de entrada devem ser armazenados nos arquivos HDF5. Os arquivos HDF5 para treinamento devem conter dois conjuntos de dados: raw e label . Opcionalmente, ao treinar com PixelWiseCrossEntropyLoss , deve -se fornecer conjunto de dados weight . O conjunto de dados raw deve conter os dados de entrada, enquanto o conjunto de dados label dos rótulos da verdade do solo. O conjunto de dados weight opcional deve conter os valores para ponderar a função de perda em diferentes regiões da entrada e deve ter o mesmo tamanho do conjunto de dados label . O formato dos conjuntos de dados raw / label depende se o problema é 2D ou 3D e se os dados são de canal único ou multicanal, consulte a tabela abaixo:
| 2d | 3d | |
|---|---|---|
| canal único | (1, y, x) | (Z, Y, X) |
| multicanal | (C, 1, y, x) | (C, Z, Y, X) |
pytorch-3dunet é um pacote cruzado e é executado no Windows e OS X também.
pytorch-3dunet é via conda/mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
Após a instalação, os seguintes comandos estão acessíveis no ambiente do CONDA: train3dunet para treinar a rede e predict3dunet para previsão (veja abaixo).
python setup.py install
Certifique -se de que o pytorch instalado seja compatível com sua versão CUDA; caso contrário, a treinamento/previsão não será executada na GPU.
Dado que o pacote pytorch-3dunet foi instalado via CONDA, conforme descrito acima, pode-se treinar a rede simplesmente invocando:
train3dunet --config <CONFIG>
Onde CONFIG é o caminho para um arquivo de configuração da YAML, que especifica todos os aspectos do procedimento de treinamento.
Para treinar seus próprios dados, forneça os caminhos aos seus conjuntos de dados de treinamento e validação em HDF5 na configuração.
Pode -se monitorar o progresso do treinamento com o Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (você precisa tensorflow instalado no seu CONDA ENV), onde checkpoint_dir é o caminho para o diretório do ponto de verificação especificado na configuração.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : os dados de destino devem ser 4D (uma máscara binária alvo por canal). Ao treinar com WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss , o conjunto de dados de destino deve ser 3D, consulte também documentação de pytorch para perda de CE: https://pytorch.org/docs/master/generated/torch.nn.crossentropylishish.htmlosfinal_sigmoid na seção de configuração model se aplica apenas ao tempo de inferência (validação, teste):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) Definir final_sigmoid=False para que a normalização Softmax seja aplicada à saída. Dado que o pacote pytorch-3dunet foi instalado via CONDA, como descrito acima, pode-se executar a previsão via:
predict3dunet --config <CONFIG>
Para prever seus próprios dados, basta fornecer o caminho para o seu modelo, bem como os caminhos para os arquivos de teste HDF5 (consulte o exemplo test_config_segmentation.yaml).
LazyHDF5Dataset e LazyPredictor na configuração. Isso salvará a memória carregando dados em tempo real à custa do tempo de previsão mais lento. Consulte Test_Config_Lazy para obter um exemplo de configuração.save_segmentation: true na seção predictor da configuração (consulte TEST_CONFIG_MULTICLASS). Por padrão, se várias GPUs estiverem disponíveis treinamento/previsão será executado em todas as GPUs usando o DataParallelel. Se o treinamento/previsão em todas as GPUs disponíveis não for desejável, restrinja o número de GPUs usando CUDA_VISIBLE_DEVICES , por exemplo,
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >ou
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (binário entre entropia)DiceLoss ( DiceLoss padrão definido como 1 - DiceCoefficient usado para segmentação semântica binária; quando mais de 2 classes estão presentes na verdade do solo, ele calcula o DiceLoss por canal e calcula a média dos valores)BCEDiceLoss (combinação linear de perdas de BCE e dados, ou seja, alpha * BCE + beta * Dice , alpha, beta pode ser especificado na seção loss da configuração)CrossEntropyLoss (pode -se especificar pesos de classe através do weight: [w_1, ..., w_k] na seção de loss da configuração)PixelWiseCrossEntropyLoss (pode-se especificar pesos por pixel para dar mais gradiente às regiões importantes/sub-representadas na verdade do solo; o conjunto de dados weight deve ser fornecido nos arquivos H5 para treinamento e validação; consulte a amostra Config em tren_config.ymlWeightedCrossEntropyLoss (consulte 'entropia cruzada ponderada (WCE)' no papel abaixo para uma explicação detalhada)GeneralizedDiceLoss (consulte 'Perda de dados generalizados (GDL)' no artigo abaixo para uma explicação detalhada) NOTA: Use essa função de perda apenas se os rótulos no conjunto de dados de treinamento forem muito desequilibrados, por exemplo, uma classe com pelo menos 3 ordens de magnitude mais voxels que os outros. Caso contrário, use DiceLoss padrão.Para uma explicação detalhada de algumas das funções de perda suportadas, consulte: Dados generalizados se sobrepõem como uma função de perda de aprendizado profunda para segmentações altamente desequilibradas.
MSELoss (perda média de erro ao quadrado)L1Loss (perda média de erro absoluto)SmoothL1Loss (menos sensível a outliers que mseloss)WeightedSmoothL1Loss (extensão do SmoothL1Loss , que permite ponderar os valores do voxel acima/abaixo de um determinado limiar de maneira diferente) MeanIoU (interseção média sobre Union)DiceCoefficient (calcula o coeficiente de dados por canal e retorna a média) Se uma rede U 3D foi treinada para prever os limites das células, pode-se usar as seguintes métricas de segmentação de instâncias semânticas (as métricas abaixo são calculadas pela execução de componentes conectados no limite do mapa e comparando as instâncias resultantes à segmentação da segmentação da verdade):BoundaryAveragePrecision (precisão média aplicada aos mapas de probabilidade de limite: limiar a saída da rede, executa componentes conectados para obter a segmentação e calcula AP entre a segmentação resultante e a verdade do solo)AdaptedRandError (consulte http://brainiac2.mit.edu/snemi3d/avaluation para uma explicação detalhada)AveragePrecision (consulte https://www.kaggle.com/stkbailey/step-by-tep-explanation-of-scoring-métrico) Se não for especificado, MeanIoU será usado por padrão.
PSNR (Razão de sinal de pico / ruído)MSE (erro médio ao quadrado) As configurações de treinamento/previsões podem ser encontradas em 3dunet_lightsheet_boundery. Pesos do modelo pré-treinado disponíveis aqui. Para usar o modelo pré-treinado em seus próprios dados:
best_checkpoint.pytorch do link acimapredict3dunet --config test_config.ymlpre_trained na configuração YAML para apontar para o caminho best_checkpoint.pytorchOs dados usados para treinamento podem ser baixados do seguinte projeto OSF:
Exemplo de previsões de fatia Z no conjunto de testes (superior: entrada bruta, inferior: previsões de limite):

As configurações de treinamento/previsões podem ser encontradas em 3dunet_confocal_boundery. Pesos do modelo pré-treinado disponíveis aqui. Para usar o modelo pré-treinado em seus próprios dados:
best_checkpoint.pytorch do link acimapredict3dunet --config test_config.ymlpre_trained na configuração YAML para apontar para o caminho best_checkpoint.pytorchOs dados usados para treinamento podem ser baixados do seguinte projeto OSF:
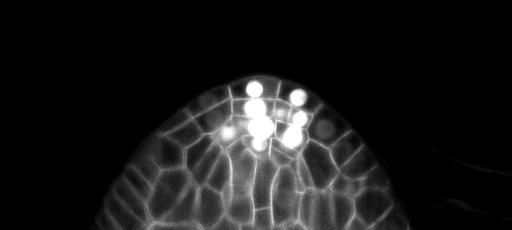
Exemplo de previsões de fatia Z no conjunto de testes (superior: entrada bruta, inferior: previsões de limite):
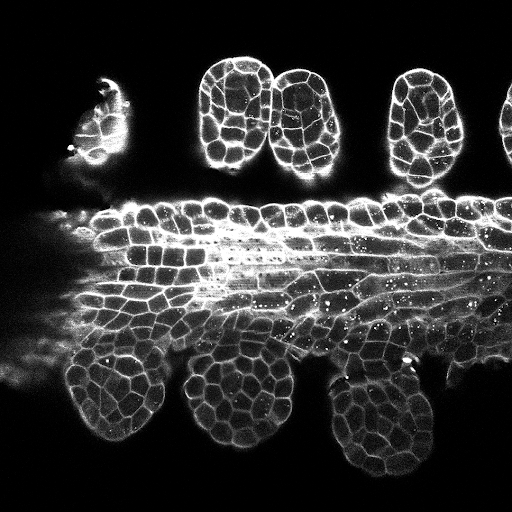
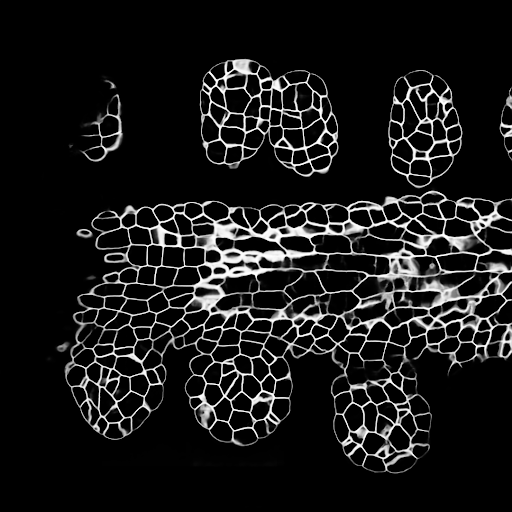
As configurações de treinamento/previsões podem ser encontradas em 3dunet_lightsheet_nuclei. Pesos do modelo pré-treinado disponíveis aqui. Para usar o modelo pré-treinado em seus próprios dados:
best_checkpoint.pytorch do link acimapredict3dunet --config test_config.ymlpre_trained na configuração YAML para apontar para o caminho best_checkpoint.pytorchOs conjuntos de treinamento e validação podem ser baixados do seguinte projeto OSF: https://osf.io/thxzn/
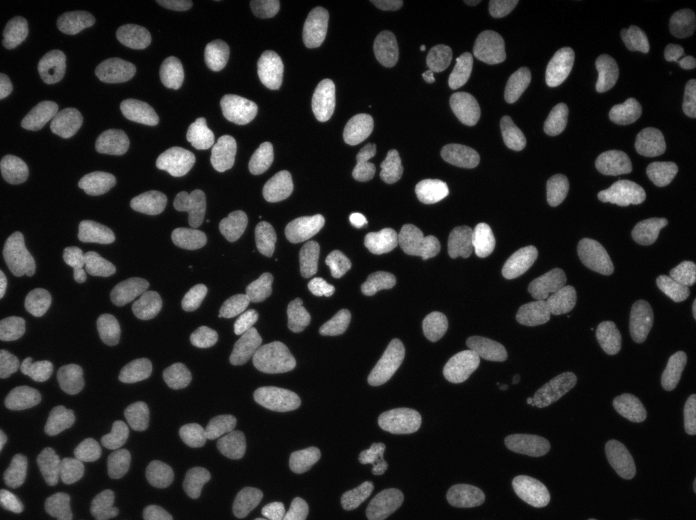
Exemplo de previsões de fatia Z no conjunto de testes (superior: entrada bruta, inferior: previsões de núcleos):
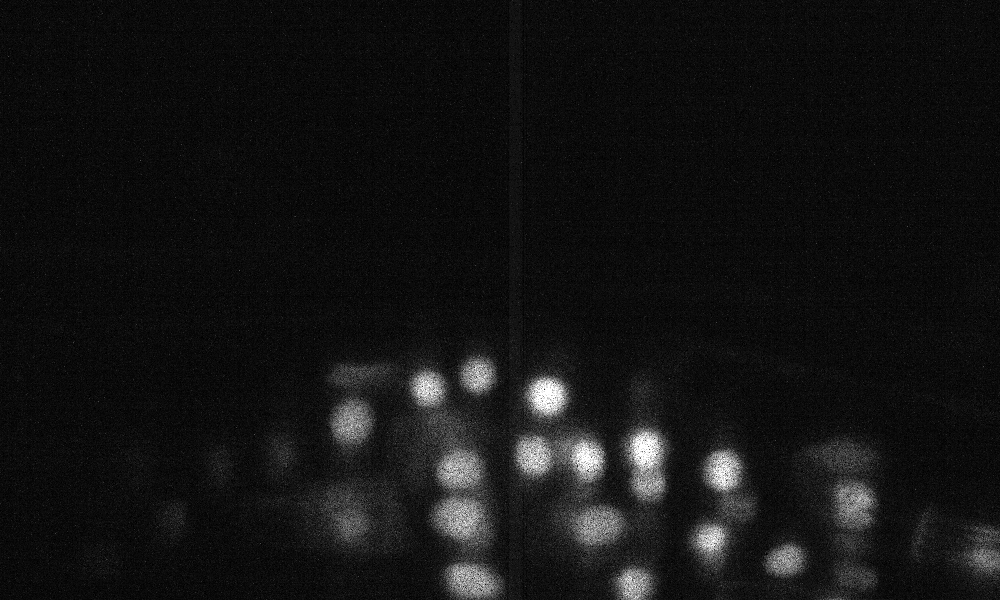

Os dados podem ser baixados de: https://www.kaggle.com/c/data-science-bowl-2018/data
As configurações de treinamento/previsões podem ser encontradas em 2dunet_dsb2018.
Previsões de amostra na imagem de teste (superior: entrada bruta, inferior: previsões de núcleos):

Se você deseja contribuir de volta, faça uma solicitação de tração.
Se você usar este código para sua pesquisa, cite como:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}