3D U-Net 및 그 변형의 Pytorch 구현 :
3D U-Net을 기반으로 한 UNet3D 표준 3D U-Net : 스파 스 주석으로부터 조밀 한 체적 분할 학습
Snemi3D Connectomics Challenge의 초인간 정확도를 기반으로하는 ResidualUNet3D
ResidualUNetSE3D 고해상도 의료 부피에 대한 딥 러닝 시맨틱 세분화를 기반으로 스퀴즈 및 여기 블록의 추가와 함께 잔류 ResidualUNet3D 과 유사합니다. 원래 압박 및 흥분 종이 : 스퀴즈 및 발행 네트워크
이 코드는 시맨틱 세분화 (바이너리 및 멀티 클래스) 및 회귀 문제 (예 : 노이즈, 학습 대충)에 대한 U-Net을 훈련시킬 수 있습니다.
2d U-Net도 지원됩니다 (예 : 구성은 2dunet_confocal 또는 2dunet_dsb2018을 참조하십시오. 데이터 로딩 / 데이터 증강에는 순위 3의 텐서가 필요하기 때문에 H5 데이터 세트 ( (1, Y, X) (Y, X) (1, 3, 3) 에 싱글 톤 z 차원을 유지하십시오.
입력 데이터는 HDF5 파일에 저장해야합니다. 교육용 HDF5 파일에는 raw 와 label 두 가지 데이터 세트가 포함되어야합니다. 선택적으로, PixelWiseCrossEntropyLoss 로 훈련 할 때 weight 데이터 세트를 제공해야합니다. raw 데이터 세트에는 입력 데이터가 포함되어야하며 label 데이터 세트는 Ground Truth 레이블입니다. 선택적 weight 데이터 세트에는 입력의 다른 영역에서 손실 함수를 가중시키는 값이 포함되어야하며 label 데이터 세트와 동일한 크기 여야합니다. raw / label 데이터 세트의 형식은 문제가 2D 또는 3D인지 여부와 데이터가 단일 채널 또는 멀티 채널인지 여부에 따라 다릅니다. 아래 표를 참조하십시오.
| 2d | 3d | |
|---|---|---|
| 단일 채널 | (1, y, x) | (Z, y, x) |
| 다 채널 | (c, 1, y, x) | (c, z, y, x) |
pytorch-3dunet 은 크로스 플랫폼 패키지이며 Windows 및 OS X에서도 실행됩니다.
pytorch-3dunet 패키지를 설치하는 가장 쉬운 방법은 Conda/Mamba를 통한 것입니다. conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
설치 후 Conda 환경 내에서 다음 명령에 액세스 할 수 있습니다. 네트워크를 훈련시키기위한 train3dunet 및 예측을 predict3dunet (아래 참조).
python setup.py install
설치된 pytorch CUDA 버전과 호환되는지 확인하십시오. 그렇지 않으면 교육/예측이 GPU에서 실행되지 않습니다.
pytorch-3dunet 패키지가 위에서 설명한대로 Conda를 통해 설치되었다는 점을 감안할 때, 단순히 다음을 호출하여 네트워크를 훈련시킬 수 있습니다.
train3dunet --config <CONFIG>
여기서 CONFIG 훈련 절차의 모든 측면을 지정하는 YAML 구성 파일의 경로입니다.
자신의 데이터를 훈련시키기 위해 구성에서 HDF5 교육 및 유효성 검사 데이터 세트의 경로를 제공하십시오.
Tensorboard tensorboard --logdir <checkpoint_dir>/logs/ (Conda Env에 설치 해야하는 텐서 tensorflow 필요합니다). 여기서 checkpoint_dir 구성에 지정된 체크 포인트 디렉토리의 경로입니다.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : 대상 데이터는 4D (채널 당 하나의 대상 바이너리 마스크) 여야합니다. WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss 로 훈련 할 때 대상 데이터 세트는 3D 여야합니다. CE 손실에 대한 pytorch 문서 : https://pytorch.org/docs/master/generated/torch.n.crossentropyloss.htmlfinal_sigmoid model 구성 섹션의 추론 시간에만 적용됩니다 (유효성 검사, 테스트) :BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss Set final_sigmoid=True 교육 할 때WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss )으로 훈련 할 때 final_sigmoid=False 설정하여 Softmax 정규화가 출력에 적용되도록합니다. pytorch-3dunet 패키지가 위에서 설명한대로 Conda를 통해 설치되었다는 점을 감안할 때 다음을 통해 예측을 실행할 수 있습니다.
predict3dunet --config <CONFIG>
자신의 데이터를 예측하려면 HDF5 테스트 파일로가는 경로뿐만 아니라 모델로가는 경로를 제공하십시오 (예 : test_config_segmentation.yaml 참조).
LazyHDF5Dataset 및 LazyPredictor 사용하는 것을 고려하십시오. 이렇게하면 예측 시간이 느려지면서 데이터를 즉시로드하여 메모리를 절약 할 수 있습니다. 예제 구성은 test_config_lazy를 참조하십시오.predictor 섹션에서 save_segmentation: true 설정하십시오 (test_config_multiclass 참조). 기본적으로 여러 GPU를 사용할 수있는 경우 DataparAllel을 사용하여 모든 GPU에서 교육/예측이 실행됩니다. 사용 가능한 모든 GPU에 대한 교육/예측이 바람직하지 않은 경우 CUDA_VISIBLE_DEVICES 사용하여 GPU 수를 제한하십시오.
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >또는
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (Binary Cross-Entropy)DiceLoss (표준 DiceLoss 1 - DiceCoefficient ; 2 개 이상의 클래스가 지상 진실에 존재하면 채널당 DiceLoss 계산하고 평균 값을 평균화합니다).BCEDiceLoss (BCE 및 주사위 손실의 선형 조합, 즉 alpha * BCE + beta * Dice , alpha, beta 구성의 loss 섹션에 지정할 수 있습니다)CrossEntropyLoss (구성의 loss 섹션에서 weight: [w_1, ..., w_k] 통해 클래스 가중치를 지정할 수 있습니다)PixelWiseCrossEntropyLoss (지상 진실의 중요/표현 영역에 더 많은 그라디언트를 제공하기 위해 픽셀 당 가중치를 지정할 수 있습니다. 훈련 및 검증을 위해 H5 파일에 weight 데이터 세트를 제공해야합니다. Train_config.yml의 샘플 구성을 참조하십시오.WeightedCrossEntropyLoss (자세한 설명을 위해 아래 논문의 '가중치 크로스-엔트로피 (WCE)'참조)GeneralizedDiceLoss 주사위 손실 (GDL)'참조) 참고 : 훈련 데이터 세트의 레이블이 다른 클래스보다 3 배 이상 더 많은 복스를 가진 한 클래스가 다른 클래스가 다른 클래스를 가진 경우에만이 손실 기능을 사용하십시오. 그렇지 않으면 표준 DiceLoss 사용하십시오.지원되는 손실 함수 중 일부에 대한 자세한 설명은 다음을 참조하십시오. 일반화 된 주사위는 균형 잡힌 분할을위한 딥 러닝 손실 함수로서 중첩됩니다.
MSELoss (평균 제곱 오류 손실)L1Loss (평균 절대 오류 손실)SmoothL1Loss (mseloss보다 이상치에 덜 민감함)WeightedSmoothL1Loss (주어진 임계 값 위/이하의 복셀 값을 가중치로 가중치를 줄 수있는 SmoothL1Loss 의 확장) MeanIoU (노동 조합에 대한 평균 교차)DiceCoefficient (채널 주사위 계수 당 계산 및 평균을 반환) 3D U-NET가 셀 경계를 예측하도록 훈련 된 경우 다음 시맨틱 인스턴스 세분화 메트릭을 사용할 수 있습니다 (아래의 메트릭은 임계 값 경계 맵에서 연결된 구성 요소를 실행하여 계산 된 인스턴스와 비교하여 계산됩니다).BoundaryAveragePrecision (경계 확률 맵에 적용되는 평균 정밀도 : 네트워크의 출력 임계 값, 연결된 구성 요소를 실행하여 세분화를 가져오고 결과 분할과 접지 진실 사이의 AP를 계산합니다).AdaptedRandError (자세한 설명은 http://brainiac2.mit.edu/snemi3d/evaluation 참조)AveragePrecision (https://www.kaggle.com/stkbailey/step-by-step-explanation-of-scoring-metric 참조) 지정되지 않은 경우 MeanIoU 기본적으로 사용됩니다.
PSNR (피크 신호 대 노이즈 비율)MSE (평균 제곱 오류) 교육/예측 구성은 3dunet_lightsheet_boundary에서 찾을 수 있습니다. 미리 훈련 된 모델 가중치는 여기에서 사용할 수 있습니다. 자신의 데이터에서 미리 훈련 된 모델을 사용하기 위해 :
best_checkpoint.pytorch 다운로드하십시오predict3dunet --config test_config.yml 실행하십시오pre_trained 속성을 best_checkpoint.pytorch Path를 가리 키게하여 사전 훈련 된 모델을 자신의 데이터로 미세 조정하십시오.교육에 사용되는 데이터는 다음 OSF 프로젝트에서 다운로드 할 수 있습니다.
테스트 세트의 샘플 Z-Slice 예측 (상단 : 원시 입력, 하단 : 경계 예측) :

교육/예측 구성은 3dunet_confocal_boundary에서 찾을 수 있습니다. 미리 훈련 된 모델 가중치는 여기에서 사용할 수 있습니다. 자신의 데이터에서 미리 훈련 된 모델을 사용하기 위해 :
best_checkpoint.pytorch 다운로드하십시오predict3dunet --config test_config.yml 실행하십시오pre_trained 속성을 best_checkpoint.pytorch Path를 가리 키게하여 사전 훈련 된 모델을 자신의 데이터로 미세 조정하십시오.교육에 사용되는 데이터는 다음 OSF 프로젝트에서 다운로드 할 수 있습니다.
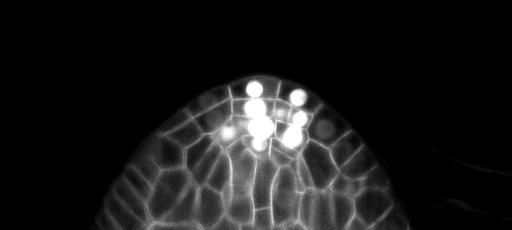
테스트 세트의 샘플 Z-Slice 예측 (상단 : 원시 입력, 하단 : 경계 예측) :
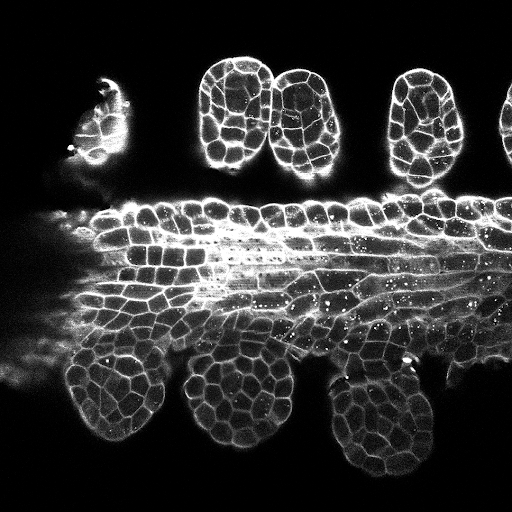
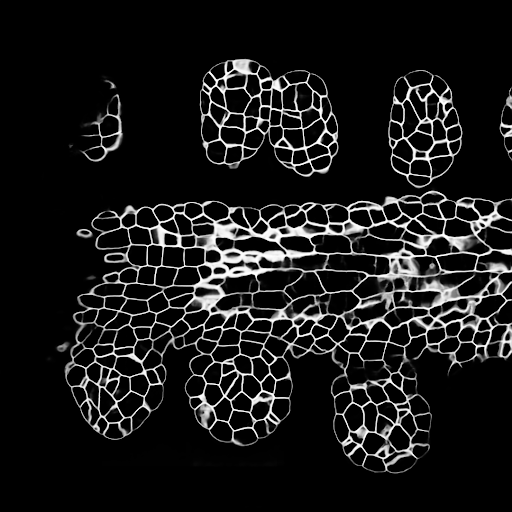
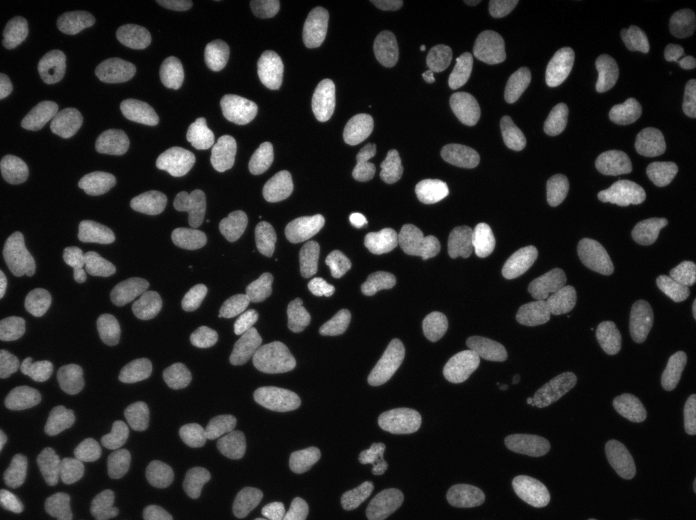
교육/예측 구성은 3dunet_lightsheet_nuclei에서 찾을 수 있습니다. 미리 훈련 된 모델 가중치는 여기에서 사용할 수 있습니다. 자신의 데이터에서 미리 훈련 된 모델을 사용하기 위해 :
best_checkpoint.pytorch 다운로드하십시오predict3dunet --config test_config.yml 실행하십시오pre_trained 속성을 best_checkpoint.pytorch Path를 가리 키게하여 사전 훈련 된 모델을 자신의 데이터로 미세 조정하십시오.교육 및 검증 세트는 다음 OSF 프로젝트에서 다운로드 할 수 있습니다 : https://osf.io/thxzn/
테스트 세트의 Z- 슬라이스 예측 (상단 : 원시 입력, 하단 : 핵 예측) :
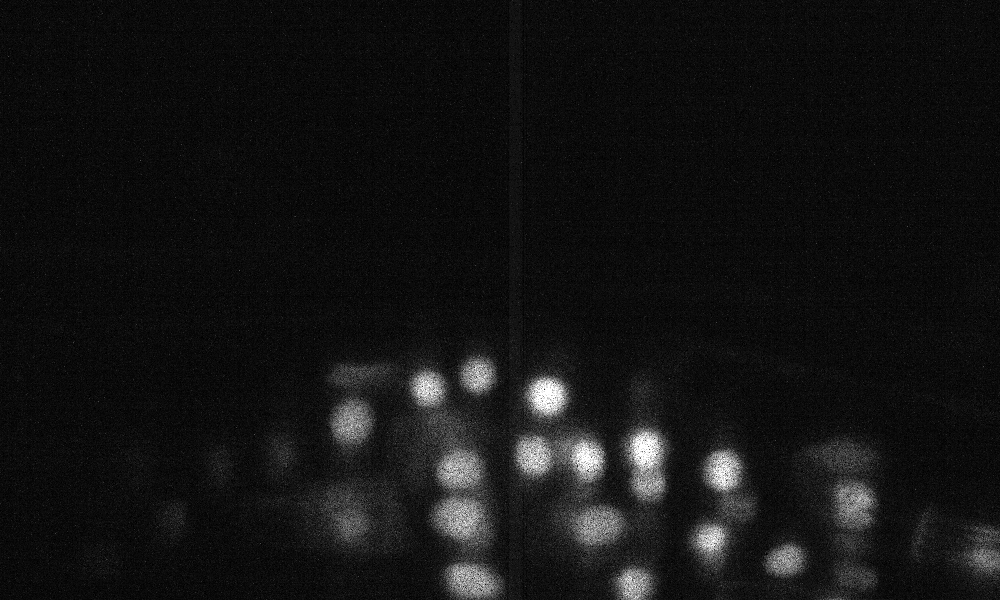

데이터는 https://www.kaggle.com/c/data-science-bowl-2018/data에서 다운로드 할 수 있습니다
교육/예측 구성은 2dunet_dsb2018에서 찾을 수 있습니다.
테스트 이미지의 샘플 예측 (상단 : 원시 입력, 하단 : 핵 예측) :

다시 기여하려면 풀 요청을하십시오.
이 코드를 연구에 사용하는 경우 다음과 같이 인용하십시오.
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}