Implementación de Pytorch de 3D U-Net y sus variantes:
UNet3D estándar 3D U-Net basado en 3D U-Net: aprendizaje de segmentación volumétrica densa de anotación dispersa
ResidualUNet3D residual 3D U-red basado en la precisión sobrehumana en el desafío Snemi3d Connectomics
ResidualUNetSE3D similar a ResidualUNet3D con la adición de bloques de apretón y excitación basados en la segmentación semántica de aprendizaje profundo para volúmenes médicos de alta resolución. Squeeze original y papel de excite: redes de exposición y excitación
El código permite capacitar a la red U para ambos: segmentación semántica (problemas de regresión binarios y de clase múltiple) y de regresión (p. Ej., Des-deconvolutiones de aprendizaje).
También es compatible con 2D U-Net, ver 2Dunet_Confocal o 2Dunet_DSB2018, por ejemplo, la configuración. Solo asegúrese de mantener la dimensión Z Singleton en su conjunto de datos H5 (es decir, (1, Y, X) en lugar de (Y, X) ), porque la carga de datos / aumento de datos requiere tensores de rango 3. La sola-red 2D en sí usa las capas convolucionales 2D estándar en lugar de convoluciones 3D con tamaño de kernel (1, 3, 3) para razones de rendimiento.
Los datos de entrada deben almacenarse en archivos HDF5. Los archivos HDF5 para capacitación deben contener dos conjuntos de datos: raw y label . Opcionalmente, cuando el entrenamiento con PixelWiseCrossEntropyLoss uno debe proporcionar un conjunto de datos weight . El conjunto de datos raw debe contener los datos de entrada, mientras que el conjunto de datos label las etiquetas de la verdad del suelo. El conjunto de datos weight opcional debe contener los valores para ponderar la función de pérdida en diferentes regiones de la entrada y debe ser del mismo tamaño que el conjunto de datos label . El formato de los conjuntos de datos raw / label depende de si el problema es 2D o 3D y si los datos son de canal único o multicanal, consulte la siguiente tabla:
| 2D | 3D | |
|---|---|---|
| un solo canal | (1, y, x) | (Z, y, x) |
| multicanal | (C, 1, y, x) | (C, Z, Y, X) |
pytorch-3dunet es un paquete multiplataforma y también se ejecuta en Windows y OS X.
pytorch-3dunet es a través de Conda/Mamba: conda install -c conda-forge mamba
mamba create -n pytorch-3dunet -c pytorch -c nvidia -c conda-forge pytorch pytorch-cuda=12.1 pytorch-3dunet
conda activate pytorch-3dunet
Después de la instalación, se pueden acceder a los siguientes comandos dentro del entorno CondA: train3dunet para capacitar a la red y predict3dunet para su predicción (ver más abajo).
python setup.py install
Asegúrese de que el pytorch instalado sea compatible con su versión CUDA, de lo contrario, la capacitación/predicción no se ejecutará en GPU.
Dado que el paquete pytorch-3dunet se instaló a través de Conda como se describió anteriormente, uno puede entrenar la red simplemente invocando:
train3dunet --config <CONFIG>
donde CONFIG es la ruta a un archivo de configuración YAML, que especifica todos los aspectos del procedimiento de capacitación.
Para entrenar en sus propios datos, solo proporcione las rutas a sus conjuntos de datos de capacitación y validación de HDF5 en la configuración.
Uno puede monitorear el progreso de la capacitación con TensorBoard tensorboard --logdir <checkpoint_dir>/logs/ (necesita tensorflow instalado en su env envado), donde checkpoint_dir es la ruta al directorio de punto de control especificado en la configuración.
BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss : los datos objetivo deben ser 4D (una máscara binaria objetivo por canal). Al entrenar con Crossentropilos WeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss , el conjunto de datos de destino debe ser 3D, consulte también la documentación de Pytorch para la pérdida de CE: https://pytorch.org/docs/master/generated/torch.nn.cossentropyloss.htmfinal_sigmoid en la sección de configuración model se aplica solo al tiempo de inferencia (validación, prueba):BCEWithLogitsLoss , DiceLoss , BCEDiceLoss , GeneralizedDiceLoss set final_sigmoid=TrueWeightedCrossEntropyLoss , CrossEntropyLoss , PixelWiseCrossEntropyLoss ) establece final_sigmoid=False de modo que la normalización Softmax se aplique a la salida. Dado que el paquete pytorch-3dunet se instaló a través de Conda como se describió anteriormente, se puede ejecutar la predicción a través de:
predict3dunet --config <CONFIG>
Para predecir en sus propios datos, solo proporcione la ruta a su modelo, así como rutas a los archivos de prueba HDF5 (consulte el ejemplo test_config_segmentation.yaml).
LazyHDF5Dataset y LazyPredictor en la configuración. Esto ahorrará memoria cargando datos sobre la marcha a costa del tiempo de predicción más lento. Consulte test_config_lazy para una configuración de ejemplo.save_segmentation: true en la sección predictor de la configuración (ver test_config_multiclass). De manera predeterminada, si hay varias GPU disponibles, la capacitación/predicción se ejecutará en todas las GPU utilizando DataParAllel. Si no es deseable la capacitación/predicción en todas las GPU disponibles, restringir el número de GPU usando CUDA_VISIBLE_DEVICES , por ejemplo
CUDA_VISIBLE_DEVICES=0,1 train3dunet --config < CONFIG >o
CUDA_VISIBLE_DEVICES=0,1 predict3dunet --config < CONFIG > BCEWithLogitsLoss (entropía cruzada binaria)DiceLoss ( DiceLoss estándar definido como 1 - DiceCoefficient UTILIZADO PARA SEGMIGACIÓN SEMANTICA BINARIA; Cuando hay más de 2 clases presentes en la verdad del suelo, calcula el DiceLoss por canal y promedia los valores)BCEDiceLoss (combinación lineal de pérdidas BCE y dados, es decir, alpha * BCE + beta * Dice , alpha, beta se puede especificar en la sección loss de la configuración)CrossEntropyLoss (se puede especificar pesos de clase a través del weight: [w_1, ..., w_k] en la sección loss de la configuración)PixelWiseCrossEntropyLoss (se puede especificar pesos por píxel para dar más gradiente a las regiones importantes/subrepresentadas en la verdad del suelo; el conjunto de datos weight debe proporcionarse en los archivos H5 para capacitación y validación; vea la configuración de muestra en Train_config.ymlWeightedCrossEntropyLoss (ver 'Entropía cruzada ponderada (WCE)' en el siguiente documento para una explicación detallada)GeneralizedDiceLoss (ver 'Pérdida de dados generalizada (GDL)' en el siguiente documento para una explicación detallada) Nota: Use esta función de pérdida solo si las etiquetas en el conjunto de datos de entrenamiento son muy desequilibrados, por ejemplo, una clase que tiene al menos 3 órdenes de magnitud más vóxeles que los otros. De lo contrario, use DiceLoss estándar.Para obtener una explicación detallada de algunas de las funciones de pérdida respaldadas, ver: dados generalizados superposición como una función de pérdida de aprendizaje profundo para segmentaciones altamente desequilibradas.
MSELoss (pérdida de error al cuadrado medio)L1Loss (pérdida media de error absoluto)SmoothL1Loss (menos sensible a los valores atípicos que MSELOSS)WeightedSmoothL1Loss (Extensión del SmoothL1Loss que permite pescar los valores de Voxel por encima/por debajo de un umbral dado de manera diferente) MeanIoU (intersección media sobre la unión)DiceCoefficient (calculos por coeficiente de dados de canal y devuelve el promedio) Si se entrenó una red U 3D para predecir los límites de las celdas, se pueden usar las siguientes métricas de segmentación de instancias semánticas (las métricas a continuación se calculan ejecutando componentes conectados en el mapa de límite de umbral y las casos comparados con la segmentación de la instancia de la verdad de tierra)BoundaryAveragePrecision (precisión promedio aplicada a los mapas de probabilidad de límite: umbral de la salida de la red, ejecuta componentes conectados para obtener la segmentación y calcula AP entre la segmentación resultante y la verdad del suelo)AdaptedRandError (ver http://brainiac2.mit.edu/snemi3d/evaluation para una explicación detallada)AveragePrecision (ver https://www.kaggle.com/stkbailey/step-by-step-explanation-of-scoring-metric) Si no se especifica, MeanIoU se usará de forma predeterminada.
PSNR (relación de señal pico a ruido)MSE (error cuadrado medio) Las configuraciones de entrenamiento/predicciones se pueden encontrar en 3Dunet_lightSheet_Boundary. Pesos de modelo previamente capacitados disponibles aquí. Para usar el modelo previamente capacitado en sus propios datos:
best_checkpoint.pytorch desde el enlace anteriorpredict3dunet --config test_config.ymlpre_trained en la configuración YAML para apuntar a la ruta best_checkpoint.pytorchLos datos utilizados para la capacitación se pueden descargar del siguiente proyecto OSF:
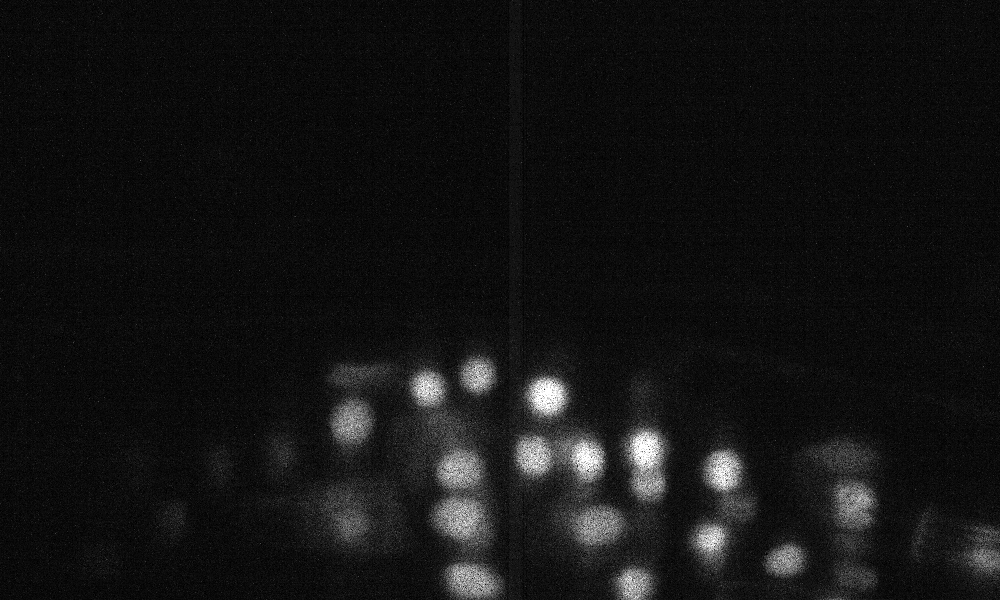
Muestra de predicciones de deslizamiento Z en el conjunto de pruebas (arriba: entrada sin procesar, abajo: predicciones límite):

Las configuraciones de entrenamiento/predicciones se pueden encontrar en 3Dunet_Confocal_Boundary. Pesos de modelo previamente capacitados disponibles aquí. Para usar el modelo previamente capacitado en sus propios datos:
best_checkpoint.pytorch desde el enlace anteriorpredict3dunet --config test_config.ymlpre_trained en la configuración YAML para apuntar a la ruta best_checkpoint.pytorchLos datos utilizados para la capacitación se pueden descargar del siguiente proyecto OSF:
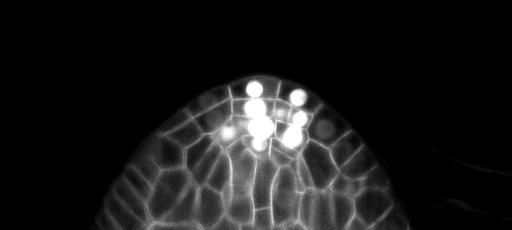
Muestra de predicciones de deslizamiento Z en el conjunto de pruebas (arriba: entrada sin procesar, abajo: predicciones límite):
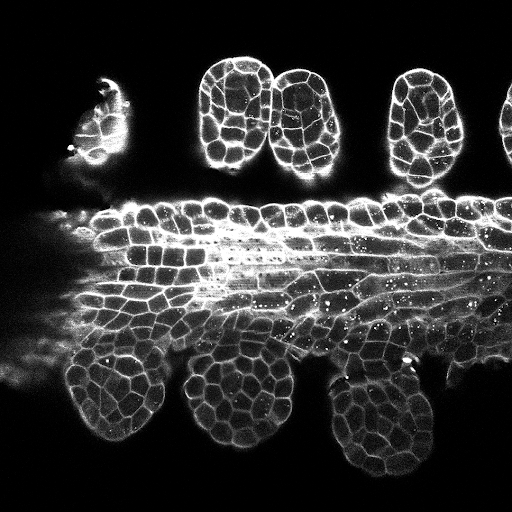
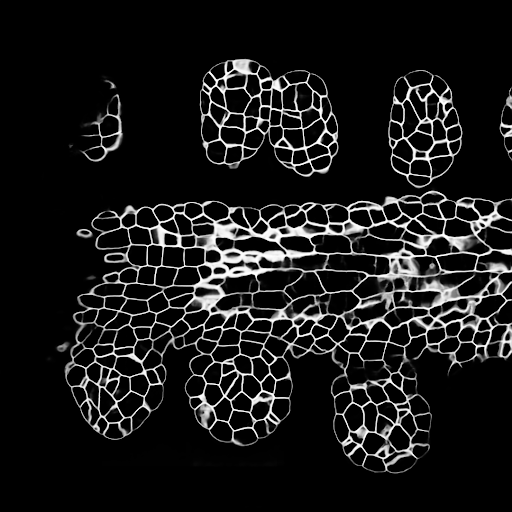
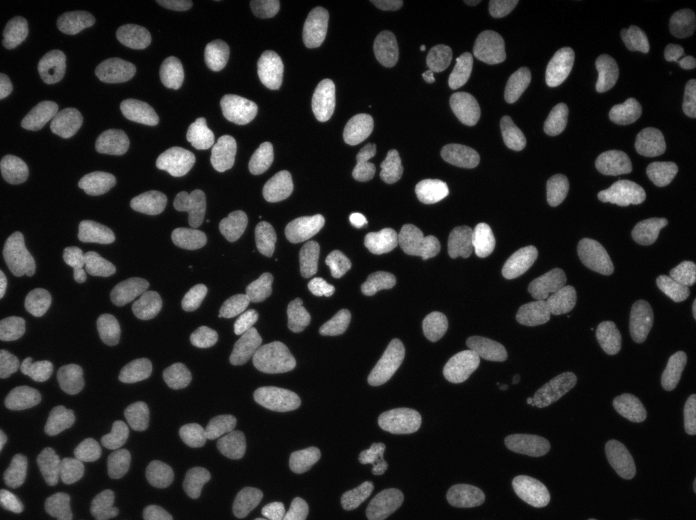
Las configuraciones de entrenamiento/predicciones se pueden encontrar en 3Dunet_lightSheet_nuclei. Pesos de modelo previamente capacitados disponibles aquí. Para usar el modelo previamente capacitado en sus propios datos:
best_checkpoint.pytorch desde el enlace anteriorpredict3dunet --config test_config.ymlpre_trained en la configuración YAML para apuntar a la ruta best_checkpoint.pytorchLos conjuntos de capacitación y validación se pueden descargar del siguiente proyecto OSF: https://osf.io/thxzn/
Muestra de predicciones de deslizamiento Z en el conjunto de pruebas (arriba: entrada en bruto, abajo: predicciones de núcleos):

Los datos se pueden descargar desde: https://www.kaggle.com/c/data-science-bowl-2018/data
Las configuraciones de capacitación/predicciones se pueden encontrar en 2Dunet_DSB2018.
Predicciones de muestra en la imagen de prueba (arriba: entrada sin procesar, abajo: predicciones de núcleos):

Si desea contribuir, haga una solicitud de extracción.
Si usa este código para su investigación, cite como:
@article {10.7554/eLife.57613,
article_type = {journal},
title = {Accurate and versatile 3D segmentation of plant tissues at cellular resolution},
author = {Wolny, Adrian and Cerrone, Lorenzo and Vijayan, Athul and Tofanelli, Rachele and Barro, Amaya Vilches and Louveaux, Marion and Wenzl, Christian and Strauss, Sören and Wilson-Sánchez, David and Lymbouridou, Rena and Steigleder, Susanne S and Pape, Constantin and Bailoni, Alberto and Duran-Nebreda, Salva and Bassel, George W and Lohmann, Jan U and Tsiantis, Miltos and Hamprecht, Fred A and Schneitz, Kay and Maizel, Alexis and Kreshuk, Anna},
editor = {Hardtke, Christian S and Bergmann, Dominique C and Bergmann, Dominique C and Graeff, Moritz},
volume = 9,
year = 2020,
month = {jul},
pub_date = {2020-07-29},
pages = {e57613},
citation = {eLife 2020;9:e57613},
doi = {10.7554/eLife.57613},
url = {https://doi.org/10.7554/eLife.57613},
keywords = {instance segmentation, cell segmentation, deep learning, image analysis},
journal = {eLife},
issn = {2050-084X},
publisher = {eLife Sciences Publications, Ltd},
}