A resposta a perguntas (controle de qualidade) é um campo no processamento de linguagem natural (PNL) e recuperação de informações (IR). A tarefa de controle de qualidade basicamente pretende fornecer respostas precisas e rápidas à determinada pergunta em idiomas naturais usando dados ou bancos de dados. Neste projeto, abordamos o problema da resposta de perguntas em documentos médicos. Existem muitos modelos de idiomas publicados e disponíveis para uso para tarefas de resposta a perguntas. Neste projeto, queríamos desenvolver um modelo de idioma, treinado especificamente no campo médico. Nosso objetivo é desenvolver um modelo de idioma específico do contexto em documentos médicos, tem um desempenho melhor que os modelos de idiomas em geral. Usamos o Electra-Small como nosso modelo básico e o treinamos usando o conjunto de dados de papel médico e depois ajustado no conjunto de dados de controle de qualidade médico. Treinamos três modelos diferentes e comparamos seus resultados nas tarefas a jusante da PNL.
Você pode acessar nossos modelos aqui:
MED-ELECTRA Small Model 17GB-64K Vocab https://huggingface.co/enelpi/med-electra-small-discriminator
MED-ELECTRA Small Model 39GB-30,5K Vocab https://huggingface.co/enelpi/med-electra-small-30k-discriminator
MED-ELECTRA Small Model 39GB-64K Vocab https://huggingface.co/enelpi/med-electra-small-64k-discriminator
Utilizamos documentos médicos S2ORC. Filizamos o banco de dados S2ORC usando o campo de estudo e tomamos documentos médicos. Utilizamos dois conjuntos de dados diferentes, consistem em fragmentos, levamos 11 fragmentos para o conjunto de dados de 17 GB e usamos 26 fragmentos para dados de 39 GB. Depois disso, levamos os publicados no PubMed e PubMedCentral. Utilizamos apenas os PDF_PARSEs desses documentos, pois as frases no PDF_PARSEs contêm mais informações.
{
"section": "Introduction",
"text": "Dogs are happier cats [13, 15]. See Figure 3 for a diagram.",
"cite_spans": [
{"start": 22, "end": 25, "text": "[13", "ref_id": "BIBREF11"},
{"start": 27, "end": 30, "text": "15]", "ref_id": "BIBREF30"},
...
],
"ref_spans": [
{"start": 36, "end": 44, "text": "Figure 3", "ref_id": "FIGREF2"},
]
}
{
...,
"BIBREF11": {
"title": "Do dogs dream of electric humans?",
"authors": [
{"first": "Lucy", "middle": ["Lu"], "last": "Wang", "suffix": ""},
{"first": "Mark", "middle": [], "last": "Neumann", "suffix": "V"}
],
"year": "",
"venue": "barXiv",
"link": null
},
...
}
{
"TABREF4": {
"text": "Table 5. Clearly, we achieve SOTA here or something.",
"type": "table"
}
...,
"FIGREF2": {
"text": "Figure 3. This is the caption of a pretty figure.",
"type": "figure"
},
...
}
}
Resumo dos dados do corpus para 17 GB
| Frase | Palavras únicas | Tamanho | Tamanho do token | |
|---|---|---|---|---|
| Trem | 111537350 | 27609654 | 16,9 GB | 2538210492 |
Resumo dos dados do corpus para 39 GB
| Frase | Palavras únicas | Tamanho | Tamanho do token | |
|---|---|---|---|---|
| Trem | 263134203 | 52206886 | 39.9 GB | 6000436472 |
Usando o corpus gerado, pré-treinado modelo Electra-pequeno do zero. O modelo é treinado na GPU RTX 2080 TI.
| Modelo | Camadas | Tamanho oculto | Parâmetros |
|---|---|---|---|
| Electra-small | 12 | 256 | 14m |
Para 17 GB
Número de linhas: 111332331
Número de palavras (tokens): 2538210492
Este modelo levou 6 dias 12 horas para treinar
| Métrica | Valor |
|---|---|
| disco_accuracy | 0,9456 |
| disco_auc | 0,9256 |
| disco_loss | 0,154 |
| disco_precision | 0,7832 |
| Disc_recall | 0,4545 |
| perda | 10.45 |
| Masked_LM_Accuracy | 0,5168 |
| Masked_lm_loss | 2.776 |
| sampled_masked_lm_accuracy | 0,4135 |
Para 39 GB com tamanho de vocabulário de 30,5k
Número de linhas: 263134203
Número de palavras (tokens): 6000436472
Este modelo levou 5 dias 9 horas para treinar
| Métrica | Valor |
|---|---|
| disco_accuracy | 0,943 |
| disco_auc | 0,9184 |
| disco_loss | 0.1609 |
| disco_precision | 0,7718 |
| Disc_recall | 0,4153 |
| perda | 10.72 |
| Masked_LM_Accuracy | 0,5218 |
| Masked_lm_loss | 2.7 |
| sampled_masked_lm_accuracy | 0,4177 |
Para 39 GB com tamanho de vocabulário de 64k
Número de linhas: 263134203
Número de palavras (tokens): 6000436472
Este modelo levou 6 dias 12 horas para treinar
| Métrica | Valor |
|---|---|
| disco_accuracy | 0,9453 |
| disco_auc | 0,9278 |
| disco_loss | 0,1534 |
| disco_precision | 0,7788 |
| Disc_recall | 0,4655 |
| perda | 10.48 |
| Masked_LM_Accuracy | 0,5095 |
| Masked_lm_loss | 2.82 |
| sampled_masked_lm_accuracy | 0,4066 |
Para
| Modelo/Hyperparameters | TRIN_STEPS | vocab_size | batch_size |
|---|---|---|---|
| Electra-small | 1m | 64000 | 128 |
Os resultados do treinamento podem ser acessados aqui:
https://tensorboard.dev/experiment/g9pkbfzaqeacr7dgw2uljq/#scalars https://tensorboard.dev/experiment/qu1bq0mirgocgqbzhqs2ta/#scalars
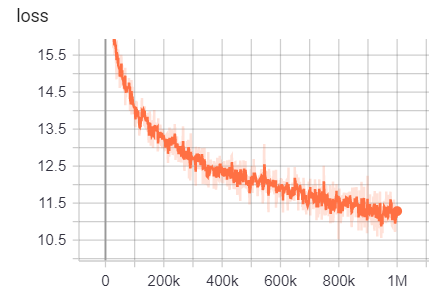
Para
| Modelo/Hyperparameters | TRIN_STEPS | vocab_size | batch_size |
|---|---|---|---|
| Electra-small | 1m | 30522 | 128 |
Os resultados do treinamento podem ser acessados aqui:
https://tensorboard.dev/experiment/npyu6mkhrmgoyd8kdsqw5w/#scalars https://tensorboard.dev/experiment/zqbeq7zjsdyijs5jb8ov3g/#scalars
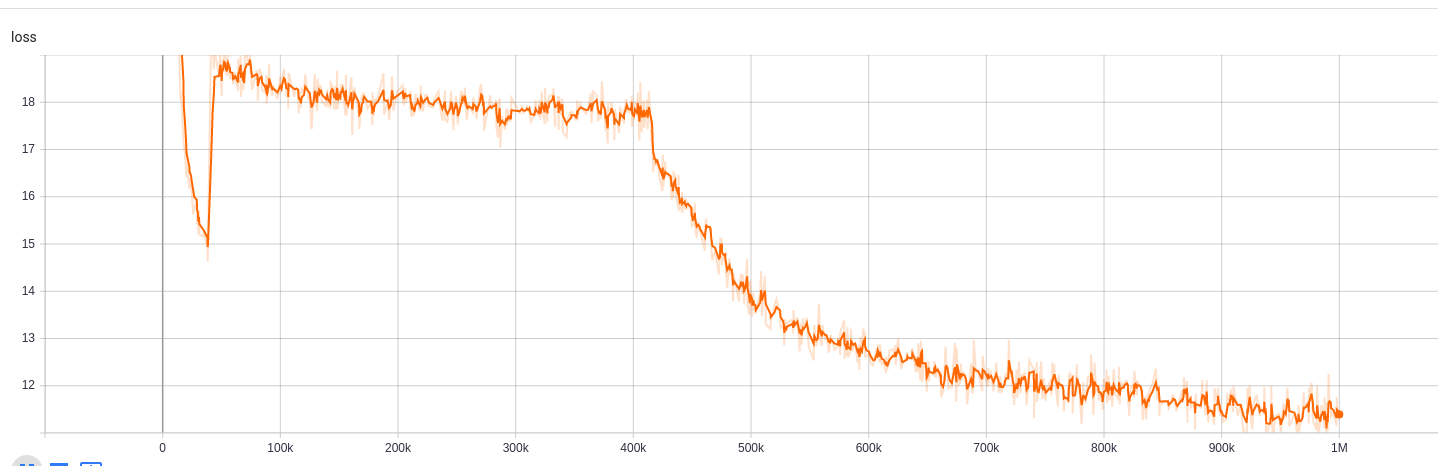
Para
| Modelo/Hyperparameters | TRIN_STEPS | vocab_size | batch_size |
|---|---|---|---|
| Electra-small | 1m | 64000 | 128 |
Os resultados do treinamento podem ser acessados aqui:
https://tensorboard.dev/experiment/gc51rmhdtgmj7eq0uyuavw/#scalars https://tensorboard.dev/experiment/q66kfo3lqtwk1kykgjcyyg/#scalars
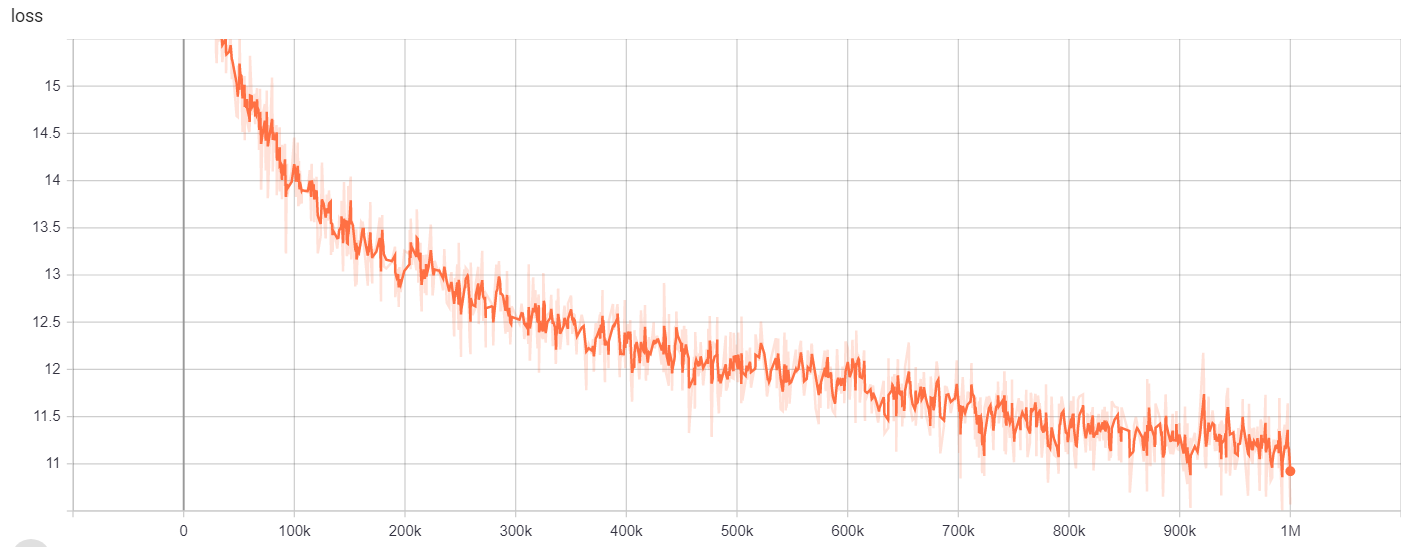
Para reconhecimento de entidade nomeado, usamos o corpus do NCBI-DISEASE, que é um recurso para o reconhecimento de nomes de doenças publicado no PubMed.
| Modelo | F1 | Perda | precisão | precisão | lembrar |
|---|---|---|---|---|---|
| ENELPI/MED-ELECTRA-MELA-DISCRIMINADOR | 0,8462 | 0,0545 | 0,9827 | 0,8052 | 0,8462 |
| Google/Electra-Small-Discriminator | 0,8294 | 0,0640 | 0,9806 | 0,7998 | 0,8614 |
| Google/Electra-Base-Discriminador | 0,8580 | 0,0675 | 0,9835 | 0,8446 | 0,8718 |
| Distilbert-Base-ANSed | 0,8348 | 0,0832 | 0,9815 | 0,8126 | 0,8583 |
| DistilroBerta-Base | 0,8416 | 0,0828 | 0,9808 | 0,8207 | 0,8635 |
| Modelo | F1 | Perda | precisão | precisão | lembrar |
|---|---|---|---|---|---|
| ENELPI/MED-ELECTRA-MELA-DISCRIMINADOR | 0,8425 | 0,0545 | 0,9824 | 0,8028 | 0,8864 |
| Google/Electra-Small-Discriminator | 0,8280 | 0,0642 | 0,9807 | 0,7961 | 0,8625 |
| Google/Electra-Base-Discriminador | 0,8648 | 0,0682 | 0,9838 | 0,8442 | 0,8864 |
| Distilbert-Base-ANSed | 0,8373 | 0.0806 | 0,9814 | 0,8153 | 0,8604 |
| DistilroBerta-Base | 0,8329 | 0.0811 | 0,9801 | 0,8100 | 0,8572 |
| Modelo | F1 | Perda | precisão | precisão | lembrar |
|---|---|---|---|---|---|
| ENELPI/MED-ELECTRA-MELA-DISCRIMINADOR | 0,8463 | 0,0559 | 0,9823 | 0,8071 | 0,8895 |
| Google/Electra-Small-Discriminator | 0,8280 | 0,0691 | 0,9806 | 0,8025 | 0,8552 |
| Google/Electra-Base-Discriminador | 0,8542 | 0,0645 | 0,9840 | 0.8307 | 0,8791 |
| Distilbert-Base-ANSed | 0,8424 | 0,0799 | 0,9822 | 0,8251 | 0,8604 |
| DistilroBerta-Base | 0,8339 | 0,0924 | 0,9806 | 0,8136 | 0,8552 |
Para tarefas de resposta a perguntas, usamos o conjunto de dados de perguntas do Bioasq.
| Modelo | SACC | LACC |
|---|---|---|
| ENELPI/MED-ELECTRA-MELA-DISCRIMINATOR-128 | 0,2821 | 0,4359 |
| Google/Electra-Discriminator-128 | 0,3077 | 0,5128 |
| ENELPI/MED-ELECTRA-SMALL-Discriminator-512 | 0,1538 | 0,3590 |
| Google/Electra-Small-Discriminator-512 | 0,2564 | 0,5128 |
Você pode acessar o vídeo de apresentação do YouTube. https://www.youtube.com/watch?v=fao9clyfldc&list=plhnxo6hzwbglge_iywgyxnmpz-3pgpggt&index=2
https://github.com/google-research/electra https://chriskhanhtran.github.io/_posts/2020-06-11-electra-spanish/ https://github.com/allenai/s2orc https://github.com/allenai/scibert https://github.com/abachaa/medquad https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5530755/ https://pubmed.ncbi.nlm.nih.gov/24377777777 https://github.com/lasseregin/medical-question-answer-data https://huggingface.co/blog/how-train https://arxiv.org/abs/1909.06146 https://www.nlm.nih.gov/databases/download/pubmed_medline.html