La respuesta a las preguntas (QA) es un campo en el procesamiento del lenguaje natural (PNL) y la recuperación de información (IR). La tarea de control de calidad básicamente tiene como objetivo dar respuestas precisas y rápidas a la pregunta dada en idiomas naturales mediante el uso de datos o bases de datos dadas. En este proyecto, abordamos el problema de la respuesta de las preguntas en los documentos médicos. Hay muchos modelos de idiomas publicados y disponibles para usar para la tarea de respuesta de preguntas. En este proyecto, queríamos desarrollar un modelo de idioma, específicamente capacitado en el campo de la medicina. Nuestro objetivo es desarrollar un modelo de lenguaje específico del contexto en documentos médicos, funciona mejor que los modelos de idiomas generales. Utilizamos Electra-Small como nuestro modelo base y lo capacitamos utilizando un conjunto de datos de papel médico, luego ajustados en el conjunto de datos de control de control médico. Entrenamos tres modelos diferentes y comparamos sus resultados en las tareas de NLP aguas abajo.
Puede acceder a nuestros modelos aquí:
MED-Electra Small Model 17GB-64K Vocab https://huggingface.co/enelpi/med-electra-small-discriminator
Modelo pequeño Med-Electra 39GB-30.5k Vocab https://huggingface.co/enelpi/med-electra-small-30k-discriminator
MED-Electra Small Model 39GB-64K Vocab https://huggingface.co/enelpi/med-electra-small-64k-discriminator
Usamos documentos médicos S2ORC. Filtramos la base de datos S2ORC usando el campo de estudio y tomamos documentos médicos. Utilizamos dos conjuntos de datos diferentes, consisten en fragmentos, tomamos 11 fragmentos para el conjunto de datos de 17 GB y utilizamos 26 fragmentos para el conjunto de datos de 39 GB. Después de eso, tomamos los que se publican en PubMed y PubMedcentral. Utilizamos solo los PDF_Parses de esos documentos, ya que las oraciones en PDF_PARSES contienen más información.
{
"section": "Introduction",
"text": "Dogs are happier cats [13, 15]. See Figure 3 for a diagram.",
"cite_spans": [
{"start": 22, "end": 25, "text": "[13", "ref_id": "BIBREF11"},
{"start": 27, "end": 30, "text": "15]", "ref_id": "BIBREF30"},
...
],
"ref_spans": [
{"start": 36, "end": 44, "text": "Figure 3", "ref_id": "FIGREF2"},
]
}
{
...,
"BIBREF11": {
"title": "Do dogs dream of electric humans?",
"authors": [
{"first": "Lucy", "middle": ["Lu"], "last": "Wang", "suffix": ""},
{"first": "Mark", "middle": [], "last": "Neumann", "suffix": "V"}
],
"year": "",
"venue": "barXiv",
"link": null
},
...
}
{
"TABREF4": {
"text": "Table 5. Clearly, we achieve SOTA here or something.",
"type": "table"
}
...,
"FIGREF2": {
"text": "Figure 3. This is the caption of a pretty figure.",
"type": "figure"
},
...
}
}
Resumen de datos del corpus para 17 GB
| Oración | Palabras únicas | Tamaño | Tamaño del token | |
|---|---|---|---|---|
| Tren | 111537350 | 27609654 | 16.9GB | 2538210492 |
Resumen de datos del corpus para 39 GB
| Oración | Palabras únicas | Tamaño | Tamaño del token | |
|---|---|---|---|---|
| Tren | 263134203 | 52206886 | 39.9GB | 6000436472 |
Usando el corpus generado, previamente entrenamos el modelo de electricidad desde cero. El modelo está entrenado en RTX 2080 TI GPU.
| Modelo | Capas | Tamaño oculto | Parámetros |
|---|---|---|---|
| Electro-pequeña | 12 | 256 | 14m |
Por 17 GB
Número de líneas: 111332331
Número de palabras (tokens): 2538210492
Este modelo tardó 6 días en 12 horas en entrenar
| Métrico | Valor |
|---|---|
| disco | 0.9456 |
| disco | 0.9256 |
| disco_loss | 0.154 |
| disc_precision | 0.7832 |
| disco | 0.4545 |
| pérdida | 10.45 |
| Masked_lm_accuracy | 0.5168 |
| Masked_lm_loss | 2.776 |
| Sampled_masked_lm_accuracy | 0.4135 |
Para 39GB con 30.5k de tamaño de vocabulario
Número de líneas: 263134203
Número de palabras (tokens): 6000436472
Este modelo tardó 5 días en 9 horas en entrenar
| Métrico | Valor |
|---|---|
| disco | 0.943 |
| disco | 0.9184 |
| disco_loss | 0.1609 |
| disc_precision | 0.7718 |
| disco | 0.4153 |
| pérdida | 10.72 |
| Masked_lm_accuracy | 0.5218 |
| Masked_lm_loss | 2.7 |
| Sampled_masked_lm_accuracy | 0.4177 |
Para 39 GB con 64k de tamaño de vocabulario
Número de líneas: 263134203
Número de palabras (tokens): 6000436472
Este modelo tardó 6 días en 12 horas en entrenar
| Métrico | Valor |
|---|---|
| disco | 0.9453 |
| disco | 0.9278 |
| disco_loss | 0.1534 |
| disc_precision | 0.7788 |
| disco | 0.4655 |
| pérdida | 10.48 |
| Masked_lm_accuracy | 0.5095 |
| Masked_lm_loss | 2.82 |
| Sampled_masked_lm_accuracy | 0.4066 |
Para
| Modelo/hiperparámetros | Train_steps | VOCAB_SIZE | lote_size |
|---|---|---|---|
| Electro-pequeña | 1M | 64000 | 128 |
Se puede acceder a los resultados de la capacitación aquí:
https://tensorboard.dev/experiment/g9pkbfzaqeacr7dgw2uljq/#scalars https://tensorboard.dev/experiment/qu1bq0mirgocgqbzhqs2ta/#scalars
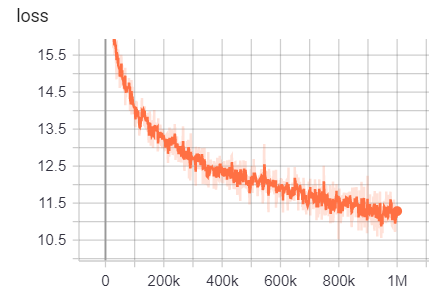
Para
| Modelo/hiperparámetros | Train_steps | VOCAB_SIZE | lote_size |
|---|---|---|---|
| Electro-pequeña | 1M | 30522 | 128 |
Se puede acceder a los resultados de la capacitación aquí:
https://tensorboard.dev/experiment/npyu6mkhrmgoyd8kdsqw5w/#scalars https://tensorboard.dev/experiment/zqbeq7zjjsdyijs5jb8ov3g/#scalars
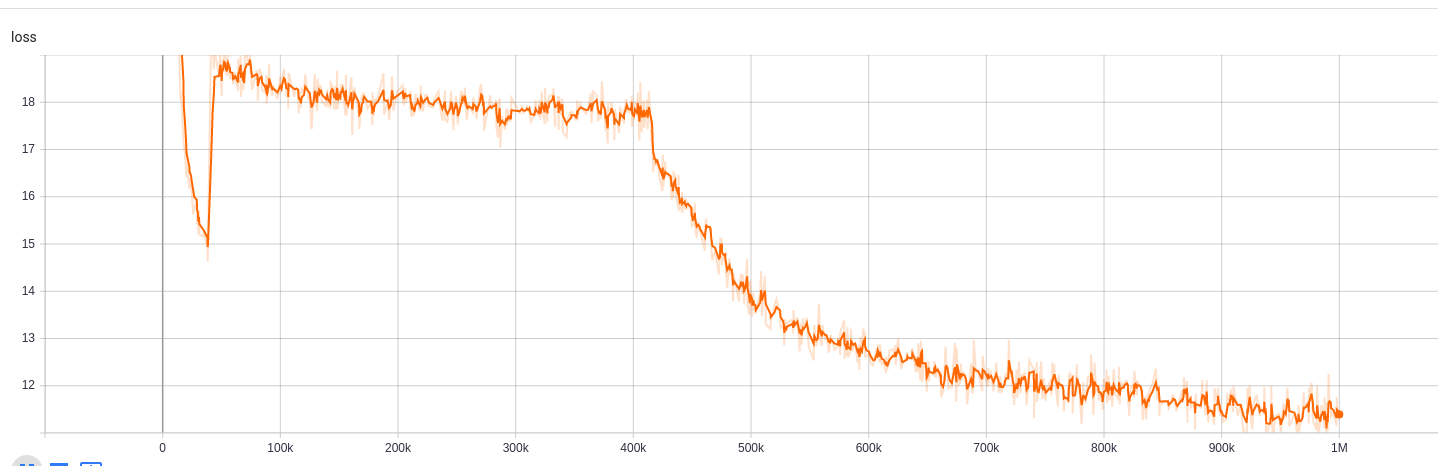
Para
| Modelo/hiperparámetros | Train_steps | VOCAB_SIZE | lote_size |
|---|---|---|---|
| Electro-pequeña | 1M | 64000 | 128 |
Se puede acceder a los resultados de la capacitación aquí:
https://tensorboard.dev/experiment/gc51rmhdtgmj7eq0uyuavw/#scalars https://tensorboard.dev/experiment/q66kfo3lqtwk1kykgjcyyg/#scalars
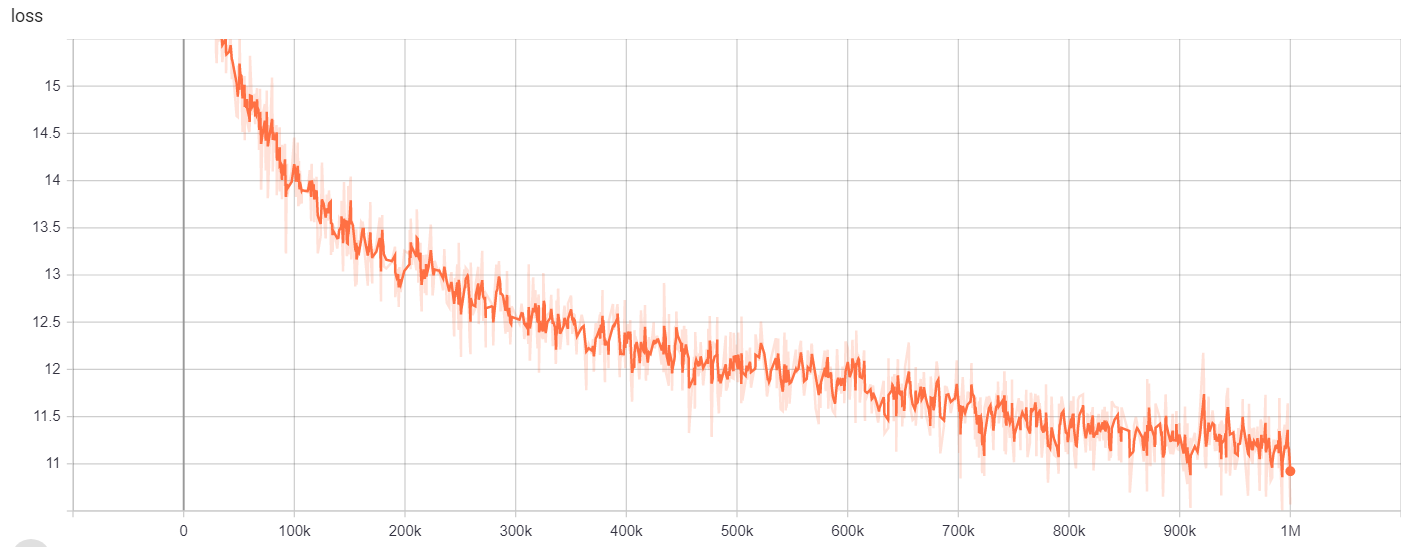
Para el reconocimiento de entidades con nombre, utilizamos el Corpus NCBI-Disease, que es un recurso para el reconocimiento de nombre de la enfermedad publicado en PubMed.
| Modelo | F1 | Pérdida | exactitud | precisión | recordar |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0.8462 | 0.0545 | 0.9827 | 0.8052 | 0.8462 |
| Google/Electra-Small-Discriminator | 0.8294 | 0.0640 | 0.9806 | 0.7998 | 0.8614 |
| Google/electra-base-discriminator | 0.8580 | 0.0675 | 0.9835 | 0.8446 | 0.8718 |
| disimitado | 0.8348 | 0.0832 | 0.9815 | 0.8126 | 0.8583 |
| base de la base | 0.8416 | 0.0828 | 0.9808 | 0.8207 | 0.8635 |
| Modelo | F1 | Pérdida | exactitud | precisión | recordar |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0.8425 | 0.0545 | 0.9824 | 0.8028 | 0.8864 |
| Google/Electra-Small-Discriminator | 0.8280 | 0.0642 | 0.9807 | 0.7961 | 0.8625 |
| Google/electra-base-discriminator | 0.8648 | 0.0682 | 0.9838 | 0.8442 | 0.8864 |
| disimitado | 0.8373 | 0.0806 | 0.9814 | 0.8153 | 0.8604 |
| base de la base | 0.8329 | 0.0811 | 0.9801 | 0.8100 | 0.8572 |
| Modelo | F1 | Pérdida | exactitud | precisión | recordar |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0.8463 | 0.0559 | 0.9823 | 0.8071 | 0.8895 |
| Google/Electra-Small-Discriminator | 0.8280 | 0.0691 | 0.9806 | 0.8025 | 0.8552 |
| Google/electra-base-discriminator | 0.8542 | 0.0645 | 0.9840 | 0.8307 | 0.8791 |
| disimitado | 0.8424 | 0.0799 | 0.9822 | 0.8251 | 0.8604 |
| base de la base | 0.8339 | 0.0924 | 0.9806 | 0.8136 | 0.8552 |
Para la tarea de respuesta de preguntas, utilizamos el conjunto de datos de preguntas BioAsq.
| Modelo | SACC | Lacc |
|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator-128 | 0.2821 | 0.4359 |
| Google/Electra-Small-Discriminator-128 | 0.3077 | 0.5128 |
| Enelpi/Med-Electra-Small-Discriminator-512 | 0.1538 | 0.3590 |
| Google/Electra-Small-Discriminator-512 | 0.2564 | 0.5128 |
Puede acceder al video de presentación desde YouTube. https://www.youtube.com/watch?v=fao9clyfldc&list=plhnxo6hzwbglge_iywgyxnmpz-3pgpggt&index=2
https://github.com/google-research/electra https://chriskhanhtran.github.io/_posts/2020-06-11-electra-spanish/ https://github.com/allenai/s2orc https://github.com/allenai/sciberT https://github.com/abachaa/medquad https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5530755/ https://pubmed.ncbi.nlm.nih.gov/24393765/ https://github.com/lasseregin/medical-question-answer-data https://huggingface.co/blog/how-to-train https://arxiv.org/abs/1909.06146 https:/www.nl.m.nih.gov/databases