La réponse à la question (QA) est un domaine du traitement du langage naturel (PNL) et de la recherche d'informations (IR). La tâche QA vise essentiellement à donner des réponses précises et rapides à la question donnée dans les langages naturels en utilisant des données ou des bases de données données. Dans ce projet, nous avons abordé le problème de la réponse aux questions sur les documents médicaux. Il existe de nombreux modèles de langage publiés et disponibles à utiliser pour la tâche de réponse aux questions. Dans ce projet, nous voulions développer un modèle de langue, spécialement formé sur le domaine médical. Notre objectif est de développer un modèle linguistique spécifique au contexte sur les documents médicaux, fonctionne mieux que les modèles de langage général. Nous avons utilisé Electra-Small comme modèle de base, et l'avons formé à l'aide d'un ensemble de données de papier médical, puis affinés sur un ensemble de données QA médical. Nous avons formé trois modèles différents et comparé leurs résultats sur les tâches en aval de la PNL.
Vous pouvez accéder à nos modèles ici:
Med-Electra Small Model 17 Go - 64K Vocab https://huggingface.co/enelpi/med-electra-small-discriminator
Med-Electra Small Model 39 Go - 30,5k Vocab https://huggingface.co/enelpi/med-electra-small-30k-discriminator
Med-Electra Small Model 39 Go - 64K Vocab https://huggingface.co/enelpi/med-electra-small-64k-discriminator
Nous avons utilisé des documents médicaux S2ORC. Nous avons filtré la base de données S2ORC à l'aide d'un domaine d'étude et pris des documents médicaux. Nous avons utilisé deux ensembles de données différents, constitué des éclats, nous avons pris 11 éclats pour l'ensemble de données de 17 Go et utilisé 26 éclats pour un ensemble de données de 39 Go. Après cela, nous avons pris ceux qui sont publiés sur PubMed et PubMedcentral. Nous avons utilisé uniquement les PDF_PARSES de ces articles, car les phrases dans PDF_PARSES contient plus d'informations.
{
"section": "Introduction",
"text": "Dogs are happier cats [13, 15]. See Figure 3 for a diagram.",
"cite_spans": [
{"start": 22, "end": 25, "text": "[13", "ref_id": "BIBREF11"},
{"start": 27, "end": 30, "text": "15]", "ref_id": "BIBREF30"},
...
],
"ref_spans": [
{"start": 36, "end": 44, "text": "Figure 3", "ref_id": "FIGREF2"},
]
}
{
...,
"BIBREF11": {
"title": "Do dogs dream of electric humans?",
"authors": [
{"first": "Lucy", "middle": ["Lu"], "last": "Wang", "suffix": ""},
{"first": "Mark", "middle": [], "last": "Neumann", "suffix": "V"}
],
"year": "",
"venue": "barXiv",
"link": null
},
...
}
{
"TABREF4": {
"text": "Table 5. Clearly, we achieve SOTA here or something.",
"type": "table"
}
...,
"FIGREF2": {
"text": "Figure 3. This is the caption of a pretty figure.",
"type": "figure"
},
...
}
}
Résumé des données du corpus pour 17 Go
| Phrase | Mots uniques | Taille | Token | |
|---|---|---|---|---|
| Former | 111537350 | 27609654 | 16,9 Go | 2538210492 |
Résumé des données du corpus pour 39 Go
| Phrase | Mots uniques | Taille | Token | |
|---|---|---|---|---|
| Former | 263134203 | 52206886 | 39,9 Go | 6000436472 |
En utilisant le corpus généré, nous avons pré-formé un modèle électro-petit à partir de zéro. Le modèle est formé sur le GPU RTX 2080.
| Modèle | Couches | Taille cachée | Paramètres |
|---|---|---|---|
| Électra-petit | 12 | 256 | 14m |
Pour 17 Go
Nombre de lignes: 111332331
Nombre de mots (jetons): 2538210492
Ce modèle a pris 6 jours 12 heures pour s'entraîner
| Métrique | Valeur |
|---|---|
| disque | 0,9456 |
| disque_auc | 0,9256 |
| disque_loss | 0,154 |
| disque | 0,7832 |
| disque_recal | 0,4545 |
| perte | 10.45 |
| masquée_lm_acultation | 0,5168 |
| masqué_lm_loss | 2.776 |
| échantillon | 0,4135 |
Pour 39 Go avec une taille de vocabulaire de 30,5k
Nombre de lignes: 263134203
Nombre de mots (jetons): 6000436472
Ce modèle a pris 5 jours 9 heures pour s'entraîner
| Métrique | Valeur |
|---|---|
| disque | 0,943 |
| disque_auc | 0,9184 |
| disque_loss | 0.1609 |
| disque | 0,7718 |
| disque_recal | 0,4153 |
| perte | 10.72 |
| masquée_lm_acultation | 0,5218 |
| masqué_lm_loss | 2.7 |
| échantillon | 0,4177 |
Pour 39 Go avec une taille de vocabulaire 64k
Nombre de lignes: 263134203
Nombre de mots (jetons): 6000436472
Ce modèle a pris 6 jours 12 heures pour s'entraîner
| Métrique | Valeur |
|---|---|
| disque | 0,9453 |
| disque_auc | 0,9278 |
| disque_loss | 0,1534 |
| disque | 0,7788 |
| disque_recal | 0,4655 |
| perte | 10.48 |
| masquée_lm_acultation | 0,5095 |
| masqué_lm_loss | 2.82 |
| échantillon | 0,4066 |
Pour
| Modèle / hyperparamètres | Train_steps | vocab_size | batch_size |
|---|---|---|---|
| Électra-petit | 1m | 64000 | 128 |
Les résultats de la formation sont accessibles ici:
https://tensorboard.dev/experiment/g9pkbfzaqEcr7dgw2uljq/#scalars https://tensorboard.dev/experiment/qu1bq0Mirgocgqbzhqs2ta/#scalars
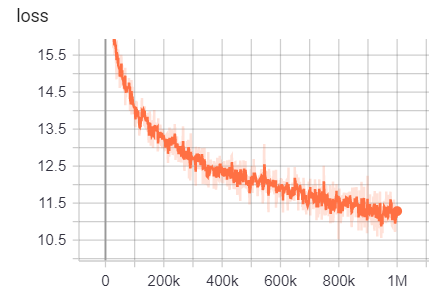
Pour
| Modèle / hyperparamètres | Train_steps | vocab_size | batch_size |
|---|---|---|---|
| Électra-petit | 1m | 30522 | 128 |
Les résultats de la formation sont accessibles ici:
https://tensorboard.dev/experiment/npyu6mkhrmgoyd8kdsqw5w/#calars https://tensorboard.dev/experiment/zqbeq7zjsdyijs5jb8ov3g/#scalars
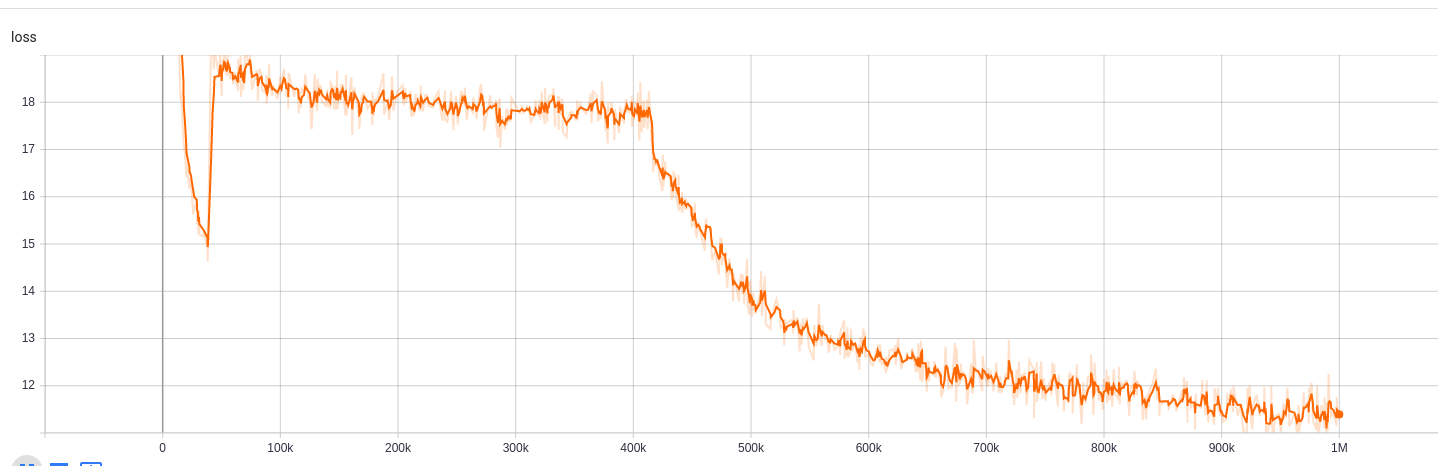
Pour
| Modèle / hyperparamètres | Train_steps | vocab_size | batch_size |
|---|---|---|---|
| Électra-petit | 1m | 64000 | 128 |
Les résultats de la formation sont accessibles ici:
https://tensorboard.dev/experiment/gc51rmhdtgmj7eq0uyuavw/#calars https://tensorboard.dev/#scalars/q66kfo3lqtwk1kykgjcyyg/#scalars
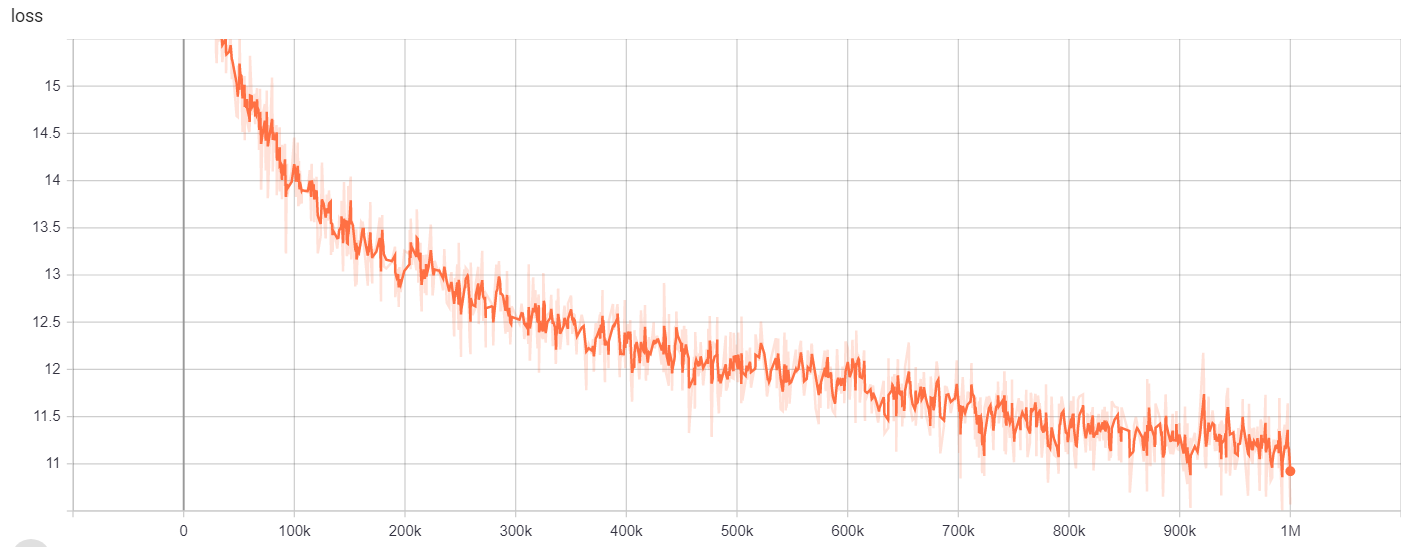
Pour la reconnaissance des entités nommées, nous avons utilisé NCBI-Dishease Corpus, qui est une ressource pour la reconnaissance du nom de la maladie publiée dans PubMed.
| Modèle | F1 | Perte | précision | précision | rappel |
|---|---|---|---|---|---|
| Enelpi / Med-Electra-Small-Discriminateur | 0,8462 | 0,0545 | 0,9827 | 0,8052 | 0,8462 |
| Google / Electra-Small-Discriminateur | 0,8294 | 0,0640 | 0,9806 | 0,7998 | 0,8614 |
| Google / Electra-Base-Discriminateur | 0,8580 | 0,0675 | 0,9835 | 0,8446 | 0,8718 |
| Distilbert-base-basée | 0,8348 | 0,0832 | 0,9815 | 0,8126 | 0,8583 |
| Distilroberta-base | 0,8416 | 0,0828 | 0,9808 | 0,8207 | 0,8635 |
| Modèle | F1 | Perte | précision | précision | rappel |
|---|---|---|---|---|---|
| Enelpi / Med-Electra-Small-Discriminateur | 0,8425 | 0,0545 | 0,9824 | 0,8028 | 0,8864 |
| Google / Electra-Small-Discriminateur | 0,8280 | 0,0642 | 0,9807 | 0,7961 | 0,8625 |
| Google / Electra-Base-Discriminateur | 0,8648 | 0,0682 | 0,9838 | 0,8442 | 0,8864 |
| Distilbert-base-basée | 0,8373 | 0,0806 | 0,9814 | 0,8153 | 0,8604 |
| Distilroberta-base | 0,8329 | 0,0811 | 0,9801 | 0,8100 | 0,8572 |
| Modèle | F1 | Perte | précision | précision | rappel |
|---|---|---|---|---|---|
| Enelpi / Med-Electra-Small-Discriminateur | 0,8463 | 0,0559 | 0,9823 | 0,8071 | 0,8895 |
| Google / Electra-Small-Discriminateur | 0,8280 | 0,0691 | 0,9806 | 0,8025 | 0,8552 |
| Google / Electra-Base-Discriminateur | 0,8542 | 0,0645 | 0,9840 | 0,8307 | 0,8791 |
| Distilbert-base-basée | 0,8424 | 0,0799 | 0,9822 | 0,8251 | 0,8604 |
| Distilroberta-base | 0,8339 | 0,0924 | 0,9806 | 0,8136 | 0,8552 |
Pour la tâche de réponse aux questions, nous avons utilisé un ensemble de données de questions Bioasq.
| Modèle | SACC | LACC |
|---|---|---|
| enelpi / med-electra-small-discriminator-128 | 0,2821 | 0,4359 |
| Google / Electra-Small-Discriminator-128 | 0,3077 | 0,5128 |
| enelpi / med-electra-small-discriminator-512 | 0,1538 | 0,3590 |
| Google / Electra-Small-Discriminator-512 | 0,2564 | 0,5128 |
Vous pouvez accéder à la vidéo de présentation depuis YouTube. https://www.youtube.com/watch?v=fao9clyfldc&list=plhnxo6hzwbglge_iywgyxnmpz-3pgpggt&index=2
https://github.com/google-research/electra https://chriskhanhtran.github.io/_posts/2020-06-11-electra-spanish/ https://github.com/allenai/s2orc https://github.com/github https://github.com/abachaa/medquad https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5530755/ https://pubmed.ncbi.nlm.nih.gov/24393765/ https://github.com/LasseRegin/medical-question-answer-data https://huggingface.co/blog/how-to-train https://arxiv.org/abs/1909.06146 https://www.nlm.nih.gov/databases/download/pubmed_medline.html