Fragenbeantwortung (QA) ist ein Feld in der natürlichen Sprachverarbeitung (NLP) und des Informationsabrufs (IR). Die QA -Aufgabe zielt im Grunde genommen darauf ab, in den natürlichen Sprachen genaue und schnelle Antworten in den natürlichen Sprachen zu geben, indem angegebene Daten oder Datenbanken verwendet werden. In diesem Projekt haben wir uns mit dem Problem der Beantwortung von Fragen zur Beantwortung von medizinischen Papieren befasst. Es gibt viele Sprachmodelle, die für die Beantwortungsaufgabe verwendet werden können. In diesem Projekt wollten wir ein Sprachmodell entwickeln, das speziell auf dem medizinischen Bereich geschult wurde. Unser Ziel ist es, ein kontextspezifisches Sprachmodell für medizinische Papiere zu entwickeln und besser als allgemeine Sprachmodelle zu erzielen. Wir haben Electra-Small als Basismodell verwendet und es mit einem medizinischen Papierdatensatz trainiert und dann auf dem medizinischen QA-Datensatz fein abgestimmt. Wir haben drei verschiedene Modelle ausgebildet und ihre Ergebnisse mit den nachgeschalteten NLP -Aufgaben verglichen.
Hier können Sie auf unsere Modelle zugreifen:
Med-Electra kleines Modell 17GB-64K Vocab https://huggingface.co/enelpi/med-electra-small-discriminator
Med-Electra kleines Modell 39GB-30,5K Vocab https://huggingface.co/enelpi/med-electra-small-30k-discriminator
Med-Electra kleines Modell 39GB-64K VOCAB https://huggingface.co/enelpi/med-electra-small-64k-discriminator
Wir haben medizinische Papiere S2ORC verwendet. Wir haben die S2ORC -Datenbank unter Verwendung des Studienfeldes gefiltert und medizinische Papiere übernommen. Wir haben zwei verschiedene Datensätze verwendet, bestehen aus Scherben, haben 11 Scherben für den 17 -GB -Datensatz genommen und 26 Scherben für 39 GB Datensatz verwendet. Danach haben wir diejenigen genommen, die auf PubMed und PubMedCentral veröffentlicht werden. Wir haben nur die PDF_Parse dieser Papiere verwendet, da die Sätze in der PDF_Parse weitere Informationen enthalten.
{
"section": "Introduction",
"text": "Dogs are happier cats [13, 15]. See Figure 3 for a diagram.",
"cite_spans": [
{"start": 22, "end": 25, "text": "[13", "ref_id": "BIBREF11"},
{"start": 27, "end": 30, "text": "15]", "ref_id": "BIBREF30"},
...
],
"ref_spans": [
{"start": 36, "end": 44, "text": "Figure 3", "ref_id": "FIGREF2"},
]
}
{
...,
"BIBREF11": {
"title": "Do dogs dream of electric humans?",
"authors": [
{"first": "Lucy", "middle": ["Lu"], "last": "Wang", "suffix": ""},
{"first": "Mark", "middle": [], "last": "Neumann", "suffix": "V"}
],
"year": "",
"venue": "barXiv",
"link": null
},
...
}
{
"TABREF4": {
"text": "Table 5. Clearly, we achieve SOTA here or something.",
"type": "table"
}
...,
"FIGREF2": {
"text": "Figure 3. This is the caption of a pretty figure.",
"type": "figure"
},
...
}
}
Corpus -Datenzusammenfassung für 17 GB
| Satz | Einzigartige Worte | Größe | Tokengröße | |
|---|---|---|---|---|
| Zug | 111537350 | 27609654 | 16,9 GB | 2538210492 |
Corpus -Datenzusammenfassung für 39 GB
| Satz | Einzigartige Worte | Größe | Tokengröße | |
|---|---|---|---|---|
| Zug | 263134203 | 52206886 | 39,9GB | 6000436472 |
Mit dem erzeugten Corpus haben wir das elektra-mall-mall-Modell von Grund auf neu ausgebildet. Das Modell ist auf RTX 2080 Ti GPU ausgebildet.
| Modell | Schichten | Versteckte Größe | Parameter |
|---|---|---|---|
| Electra-Small | 12 | 256 | 14m |
Für 17 GB
Anzahl der Zeilen: 111332331
Anzahl der Wörter (Token): 2538210492
Dieses Modell dauerte 6 Tage 12 Stunden, um zu trainieren
| Metrisch | Wert |
|---|---|
| disc_accuracy | 0,9456 |
| disc_auc | 0,9256 |
| disc_loss | 0,154 |
| disc_precision | 0,7832 |
| disc_recall | 0,4545 |
| Verlust | 10.45 |
| masked_lm_accuracy | 0,5168 |
| masked_lm_loss | 2.776 |
| Sampled_masked_lm_accuracy | 0,4135 |
Für 39 GB mit 30,5.000 Vokabulargröße
Anzahl der Zeilen: 263134203
Anzahl der Wörter (Token): 6000436472
Dieses Modell dauerte 5 Tage 9 Stunden, um zu trainieren
| Metrisch | Wert |
|---|---|
| disc_accuracy | 0,943 |
| disc_auc | 0,9184 |
| disc_loss | 0,1609 |
| disc_precision | 0,7718 |
| disc_recall | 0,4153 |
| Verlust | 10.72 |
| masked_lm_accuracy | 0,5218 |
| masked_lm_loss | 2.7 |
| Sampled_masked_lm_accuracy | 0,4177 |
Für 39 GB mit 64K -Wortschatzgröße
Anzahl der Zeilen: 263134203
Anzahl der Wörter (Token): 6000436472
Dieses Modell dauerte 6 Tage 12 Stunden, um zu trainieren
| Metrisch | Wert |
|---|---|
| disc_accuracy | 0,9453 |
| disc_auc | 0,9278 |
| disc_loss | 0,1534 |
| disc_precision | 0,7788 |
| disc_recall | 0,4655 |
| Verlust | 10.48 |
| masked_lm_accuracy | 0,5095 |
| masked_lm_loss | 2.82 |
| Sampled_masked_lm_accuracy | 0,4066 |
Für
| Modell/Hyperparameter | train_steps | vocab_size | batch_size |
|---|---|---|---|
| Electra-Small | 1m | 64000 | 128 |
Die Trainingsergebnisse können hier zugegriffen werden:
https://tensorboard.dev/experiment/g9pkbfzaqeaccr7dgw2uljq/#scalars https://tensorboard.dev/experiment/qu1bq0mirgocgqbzhqs2ta/#scalars
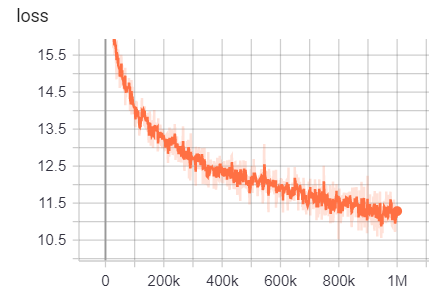
Für
| Modell/Hyperparameter | train_steps | vocab_size | batch_size |
|---|---|---|---|
| Electra-Small | 1m | 30522 | 128 |
Die Trainingsergebnisse können hier zugegriffen werden:
https://tensorboard.dev/experiment/npyu6mkhrmgoyd8kdsqw5w/#scalars https://tensorboard.dev/experiment/zqbeq7zjsdyijs5jb8ov3g/#scalars
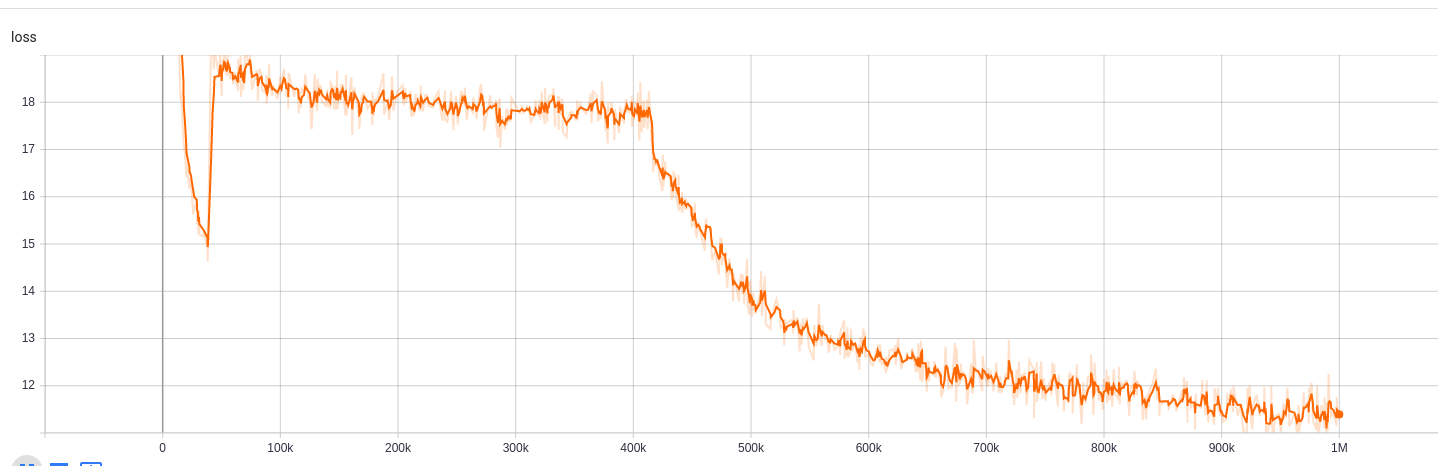
Für
| Modell/Hyperparameter | train_steps | vocab_size | batch_size |
|---|---|---|---|
| Electra-Small | 1m | 64000 | 128 |
Die Trainingsergebnisse können hier zugegriffen werden:
https://tensorboard.dev/experiment/gc51rmhdtgmj7eq0uyuavw/#scalars https://tensorboard.dev/experiment/q66kfo3lqtwk1kykgjyg/#scalars
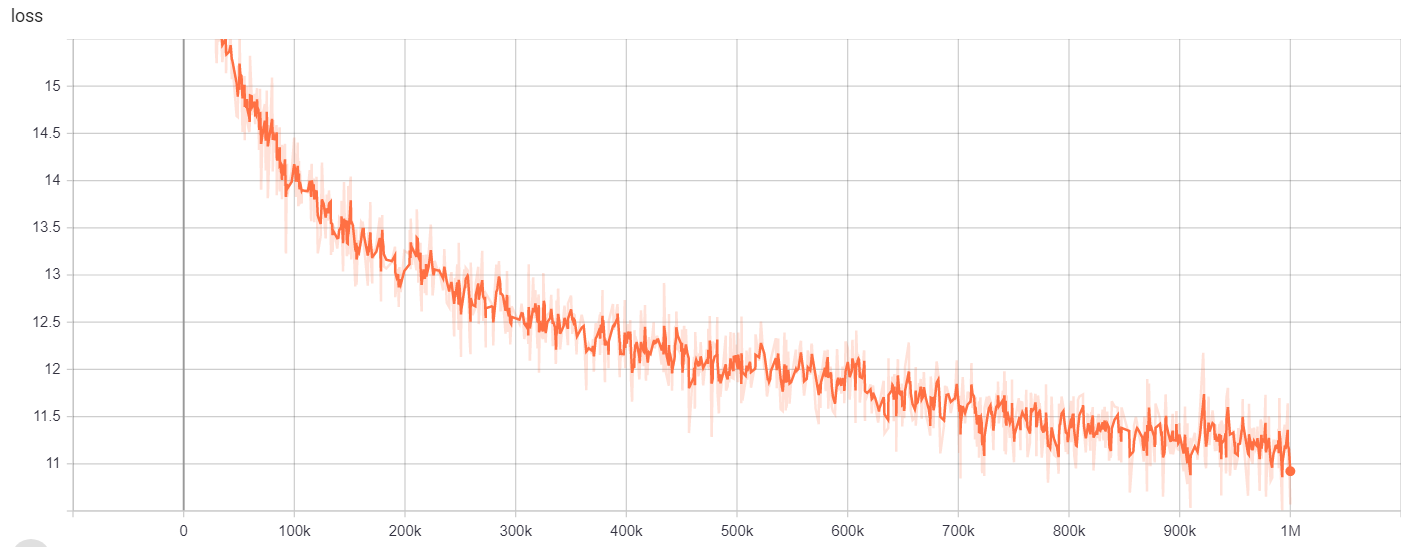
Für die genannte Entitätserkennung verwendeten wir NCBI-Disease Corpus, eine Ressource für die Erkennung von Krankheiten, die in PubMed veröffentlicht wurde.
| Modell | F1 | Verlust | Genauigkeit | Präzision | abrufen |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0,8462 | 0,0545 | 0,9827 | 0,8052 | 0,8462 |
| Google/Electra-Small-Discriminator | 0,8294 | 0,0640 | 0,9806 | 0,7998 | 0,8614 |
| Google/Electra-Base-Discriminator | 0,8580 | 0,0675 | 0,9835 | 0,8446 | 0,8718 |
| Distilbert-Base-Unbekannt | 0,8348 | 0,0832 | 0,9815 | 0,8126 | 0,8583 |
| Distilroberta-Base | 0,8416 | 0,0828 | 0,9808 | 0,8207 | 0,8635 |
| Modell | F1 | Verlust | Genauigkeit | Präzision | abrufen |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0,8425 | 0,0545 | 0,9824 | 0,8028 | 0,8864 |
| Google/Electra-Small-Discriminator | 0,8280 | 0,0642 | 0,9807 | 0,7961 | 0,8625 |
| Google/Electra-Base-Discriminator | 0,8648 | 0,0682 | 0,9838 | 0,8442 | 0,8864 |
| Distilbert-Base-Unbekannt | 0,8373 | 0,0806 | 0,9814 | 0,8153 | 0,8604 |
| Distilroberta-Base | 0,8329 | 0,0811 | 0,9801 | 0,8100 | 0,8572 |
| Modell | F1 | Verlust | Genauigkeit | Präzision | abrufen |
|---|---|---|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator | 0,8463 | 0,0559 | 0,9823 | 0,8071 | 0,8895 |
| Google/Electra-Small-Discriminator | 0,8280 | 0,0691 | 0,9806 | 0,8025 | 0,8552 |
| Google/Electra-Base-Discriminator | 0,8542 | 0,0645 | 0,9840 | 0,8307 | 0,8791 |
| Distilbert-Base-Unbekannt | 0,8424 | 0,0799 | 0,9822 | 0,8251 | 0,8604 |
| Distilroberta-Base | 0,8339 | 0,0924 | 0,9806 | 0,8136 | 0,8552 |
Für die Beantwortungsaufgabe haben wir den Bioasq -Frage -Datensatz verwendet.
| Modell | Sacc | Lacc |
|---|---|---|
| Enelpi/Med-Electra-Small-Discriminator-128 | 0,2821 | 0,4359 |
| Google/Electra-Small-Discriminator-128 | 0,3077 | 0,5128 |
| Enelpi/Med-Electra-Small-Discriminator-512 | 0,1538 | 0,3590 |
| Google/Electra-Small-Discriminator-512 | 0,2564 | 0,5128 |
Sie können von YouTube auf Präsentationsvideo zugreifen. https://www.youtube.com/watch?v=fao9clyfldc&list=plhnxo6hzwbglge_iywgyxnmpz-3pgpggt&index=2
https://github.com/google-research/electra https://chriskhanhtran.github.io/_posts/2020-06-11-electra-speanish/ https://github.com/allenai/s2orc https https://github.com/abachaa/medquad https://www.ncbi.nlm.nih.gov/pmc/articles/pmc5530755/ https://pubmed.ncbi.nlm.nih.gov/24393765/ https://github.com/lasseregin/medical-question-answer-data https://huggingface.co/blog/how-to--train https://arxiv.org/abs/1909.06146 https://www.nlm.nih.gov/databases/download/pubmed_medline.html