使用對齊算法比較二進製文件。
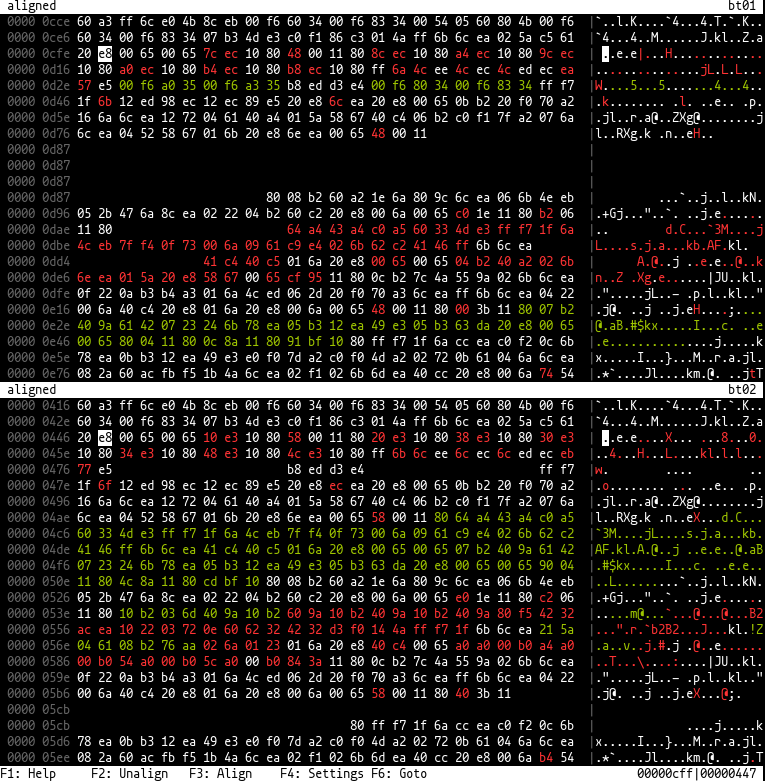
這是二進制擴散的工具。
該工具能夠並排顯示兩個二進製文件,因此在兩側的相同位置將處於相同的位置,並且一側缺少字節。它使用“ WFA2”或rust-bio庫(通常用於DNA序列比對)的生物信息學算法。用於配置的對話框是使用cursive進行的。
[ , ] , 0在終端中執行biodiff file_a file_b ,您應將您並排顯示兩個文件顯示到十六進制視圖中。最初,文件將不會對齊和顯示,而沒有每一側的空白。通過將光標和視圖移動到左側和右側相似的地方,並按下F3 (或3 ),它們可以對齊。這是按塊在標準配置中按塊完成的,這意味著首先對光標附近的字節對齊,然後在後來兩側顯示進一步的對齊塊。
也可以通過使用F4更改設置(請務必就參數諮詢幫助),對整個文件進行全局對齊。通常,由於需要二次的時間和空間,因此全局對齊對大於64kb的文件無法正常工作。還有一種“帶”的算法,該算法速度更快,但準確率略低。
您還可以在一個文件上選擇一個區域,並按下F3,對齊算法將使用選定的字節作為模式進行半光盤對齊,以在另一個文件上找到相應的字節。
也可以使用biodiff --print file_a file_b直接將差異直接打印到終端。在這種情況下(如果文件足夠小,以至於不需要太長時間),則可以添加-gglobal標誌來執行全局對齊(而不是塊置的標誌,它更適合互動使用)。
如果幸運的話,您的主要軟件包管理器中將有一個包裹,請參閱“重複”頁面。在版本頁面下的某些環境中應該有可下載的二進製文件。另外,您也可以通過cargo install biodiff來使用cargo進行安裝。您將需要安裝CMAKE,以進行wfa2功能進行編譯。請注意,如果您使用Windows,則需要使用x86_64-unknown-linux-gnu目標,如果您想獲得wfa2支持。
您也可以通過執行cargo run --release -- file_a file_b直接使用此存儲庫中的代碼執行。請注意,僅保證在標記版本之間保持兼容配置文件。
默認情況下,設置存儲在特定於平台的用戶目錄中。要使用自定義設置目錄,請在運行biodiff之前將BIODIFF_CONFIG_DIR環境變量設置為所需的目錄路徑。如果目錄不存在,它將自動創建。
該項目已根據MIT許可獲得許可。