Compare archivos binarios utilizando algoritmos de alineación.
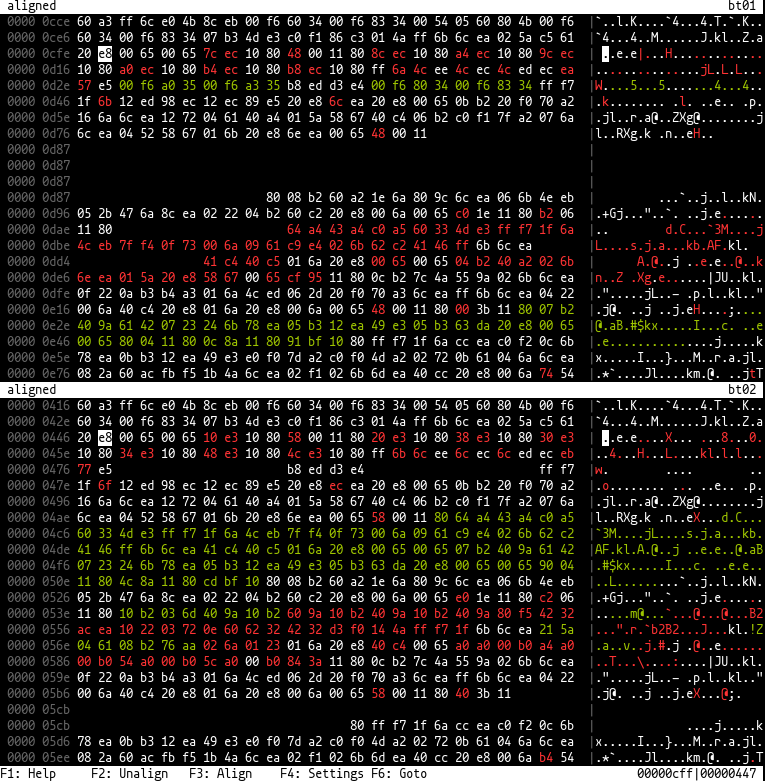
Esta es una herramienta para la difusión binaria.
La herramienta puede mostrar dos archivos binarios uno al lado del otro para que los lugares similares estén en la misma posición en ambos lados y los bytes que faltan desde un lado están acolchados. Utiliza algoritmos bioinformáticos de la biblioteca 'WFA2' o rust-bio (típicamente utilizada para la alineación de secuencia de ADN) para eso. Los cuadros de diálogo para la configuración se realizan con cursive .
[ , ] , 0 Ejecute biodiff file_a file_b en un terminal y debe ser deja caer en una vista hexadecimal que muestre dos archivos uno al lado del otro. Inicialmente, los archivos no se alinearán y mostrarán sin huecos en cada lado. Al mover el cursor y las vistas a un lugar donde el lado izquierdo y el lado derecho son similares y apremiantes F3 (o 3 ), se pueden alinear. Esto se realiza el bloque por bloque en la configuración estándar, lo que significa que los bytes cerca del cursor están alineados primero y los bloques alineados adicionales se muestran más adelante en ambos lados.
También es posible hacer una alineación global (de los archivos completos a la vez) cambiando la configuración utilizando F4 (asegúrese de consultar la ayuda en los parámetros). En general, dado que lleva tiempo y espacio cuadrático, la alineación global no funcionará bien para archivos más grandes de 64 kb. También hay un algoritmo "con bandas" que es más rápido, pero un poco menos preciso.
También puede seleccionar una región en un archivo y presionando F3, el algoritmo de alineación hará una alineación semiglóbica utilizando los bytes seleccionados como patrón para encontrar los bytes correspondientes en el otro archivo.
También es posible imprimir el DIFF directamente en el terminal usando biodiff --print file_a file_b . En ese caso (si los archivos son lo suficientemente pequeños como para que no tome demasiado tiempo), puede agregar la bandera -gglobal para hacer una alineación global (a diferencia de una blockwise, que es más adecuada para su uso interactivo).
Si tiene suerte, habrá un paquete disponible en su administrador de paquetes principales, consulte la página de Repología. Debe haber archivos binarios descargables para algunos entornos en la página de lanzamientos. Alternativamente, también puede instalar esto usando cargo haciendo cargo install biodiff . Necesitará CMAKE instalado para que la función wfa2 compile. Tenga en cuenta que en caso de que use Windows, debe usar el objetivo x86_64-unknown-linux-gnu si desea tener soporte wfa2 .
También puede ejecutar directamente usando el código de este repositorio ejecutando cargo run --release -- file_a file_b . Tenga en cuenta que los archivos de configuración solo están garantizados para mantenerse compatibles entre las versiones etiquetadas.
De forma predeterminada, la configuración se almacenan en un directorio de usuario específico de la plataforma. Para usar un directorio de configuración personalizado, establezca la variable de entorno BIODIFF_CONFIG_DIR en la ruta de directorio deseada antes de ejecutar biodiff . Si el directorio no existe, se creará automáticamente.
Este proyecto tiene licencia bajo la licencia MIT.