使用对齐算法比较二进制文件。
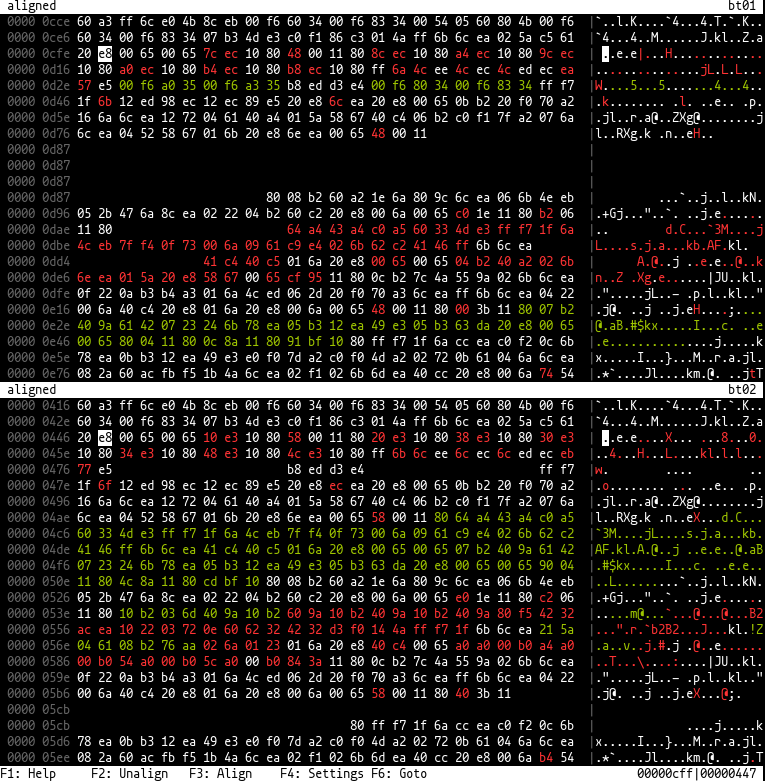
这是二进制扩散的工具。
该工具能够并排显示两个二进制文件,因此在两侧的相同位置将处于相同的位置,并且一侧缺少字节。它使用“ WFA2”或rust-bio库(通常用于DNA序列比对)的生物信息学算法。用于配置的对话框是使用cursive进行的。
[ , ] , 0在终端中执行biodiff file_a file_b ,您应将您并排显示两个文件显示到十六进制视图中。最初,文件将不会对齐和显示,而没有每一侧的空白。通过将光标和视图移动到左侧和右侧相似的地方,并按下F3 (或3 ),它们可以对齐。这是按块在标准配置中按块完成的,这意味着首先对光标附近的字节对齐,然后在后来两侧显示进一步的对齐块。
也可以通过使用F4更改设置(请务必就参数咨询帮助),对整个文件进行全局对齐。通常,由于需要二次的时间和空间,因此全局对齐对大于64kb的文件无法正常工作。还有一种“带”的算法,该算法速度更快,但准确率略低。
您还可以在一个文件上选择一个区域,并按下F3,对齐算法将使用选定的字节作为模式进行半光盘对齐,以在另一个文件上找到相应的字节。
也可以使用biodiff --print file_a file_b直接将差异直接打印到终端。在这种情况下(如果文件足够小,以至于不需要太长时间),则可以添加-gglobal标志来执行全局对齐(而不是块置的标志,它更适合互动使用)。
如果幸运的话,您的主要软件包管理器中将有一个包裹,请参阅“重复”页面。在版本页面下的某些环境中应该有可下载的二进制文件。另外,您也可以通过cargo install biodiff来使用cargo进行安装。您将需要安装CMAKE,以进行wfa2功能进行编译。请注意,如果您使用Windows,则需要使用x86_64-unknown-linux-gnu目标,如果您想获得wfa2支持。
您也可以通过执行cargo run --release -- file_a file_b直接使用此存储库中的代码执行。请注意,仅保证在标记版本之间保持兼容配置文件。
默认情况下,设置存储在特定于平台的用户目录中。要使用自定义设置目录,请在运行biodiff之前将BIODIFF_CONFIG_DIR环境变量设置为所需的目录路径。如果目录不存在,它将自动创建。
该项目已根据MIT许可获得许可。