Compare arquivos binários usando algoritmos de alinhamento.
Esta é uma ferramenta para difusão binária.
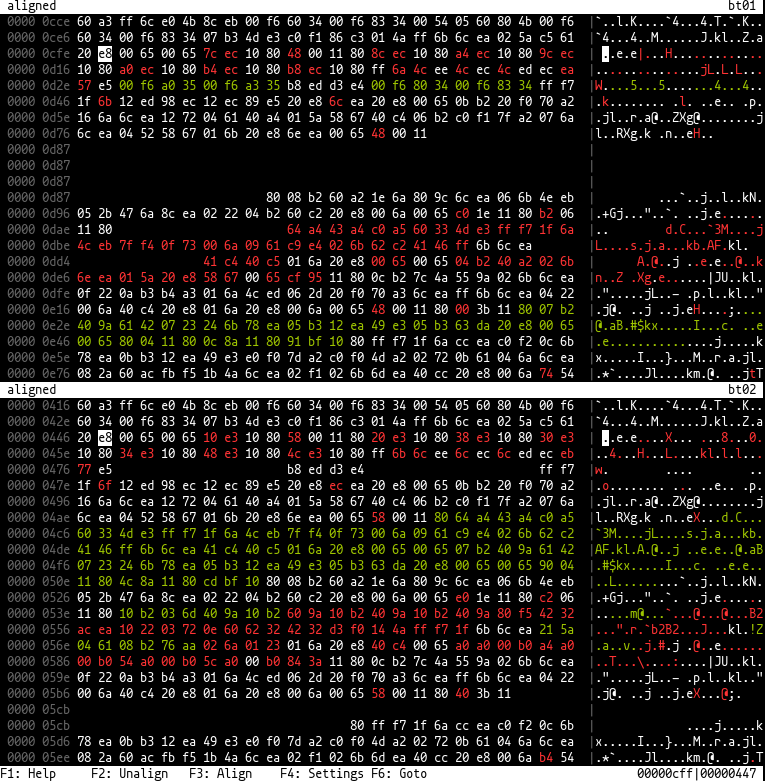
A ferramenta é capaz de mostrar dois arquivos binários lado a lado, para que locais semelhantes estejam na mesma posição nos dois lados e bytes ausentes de um lado são acolchoados. Ele usa algoritmos bio-informáticos da biblioteca 'WFA2' ou rust-bio (normalmente usada para o alinhamento da sequência de DNA) para isso. As caixas de diálogo para configuração são feitas usando cursive .
[ , ] , 0 Execute biodiff file_a file_b em um terminal e você deve ser retirado em uma visualização hexadecimal mostrando dois arquivos lado a lado. Inicialmente, os arquivos não serão alinhados e exibidos sem lacunas de cada lado. Ao mover o cursor e as vistas para um local onde o lado esquerdo e o lado direito são semelhantes e pressionando F3 (ou 3 ), eles podem ser alinhados. Isso é feito bloco a bloco na configuração padrão, o que significa que os bytes próximos ao cursor estão alinhados primeiro e adicionais blocos alinhados são exibidos posteriormente nos dois lados.
Também é possível fazer o alinhamento global (de todos os arquivos de uma só vez) alterando as configurações usando F4 (consulte a ajuda nos parâmetros). Geralmente, uma vez que leva tempo e espaço quadrático, o alinhamento global não funciona bem para arquivos maiores que 64kb. Há também um algoritmo de "faixas" que é mais rápido, mas um pouco menos preciso.
Você também pode selecionar uma região em um arquivo e pressionando F3, o algoritmo de alinhamento fará um alinhamento semiglobal usando os bytes selecionados como um padrão para encontrar os bytes correspondentes no outro arquivo.
Também é possível imprimir o diferencial diretamente para o terminal usando biodiff --print file_a file_b . Nesse caso (se os arquivos forem pequenos o suficiente para não demorar muito), você poderá adicionar o sinalizador -gglobal para fazer um alinhamento global (em oposição a um Blockwise One, que é mais adequado para uso interativo).
Se você tiver sorte, haverá um pacote disponível no seu gerenciador de pacotes primário, consulte a página de repologia. Deve haver arquivos binários para download para alguns ambientes na página de lançamentos. Como alternativa, você também pode instalar isso usando cargo fazendo cargo install biodiff . Você precisará de cmake instalado para o recurso wfa2 para compilar. Observe que, caso você use o Windows, você precisará usar o destino x86_64-unknown-linux-gnu se desejar ter suporte wfa2 .
Você também pode executar diretamente usando o código deste repositório executando a execução cargo run --release -- file_a file_b . Observe que os arquivos de configuração são garantidos apenas para permanecer compatíveis entre as versões marcadas.
Por padrão, as configurações são armazenadas em um diretório de usuário específico da plataforma. Para usar um diretório de configurações personalizado, defina a variável de ambiente BIODIFF_CONFIG_DIR no caminho do diretório desejado antes de executar biodiff . Se o diretório não existir, ele será criado automaticamente.
Este projeto está licenciado sob a licença do MIT.