정렬 알고리즘을 사용하여 이진 파일을 비교하십시오.
이것은 이진 차이를위한 도구입니다.
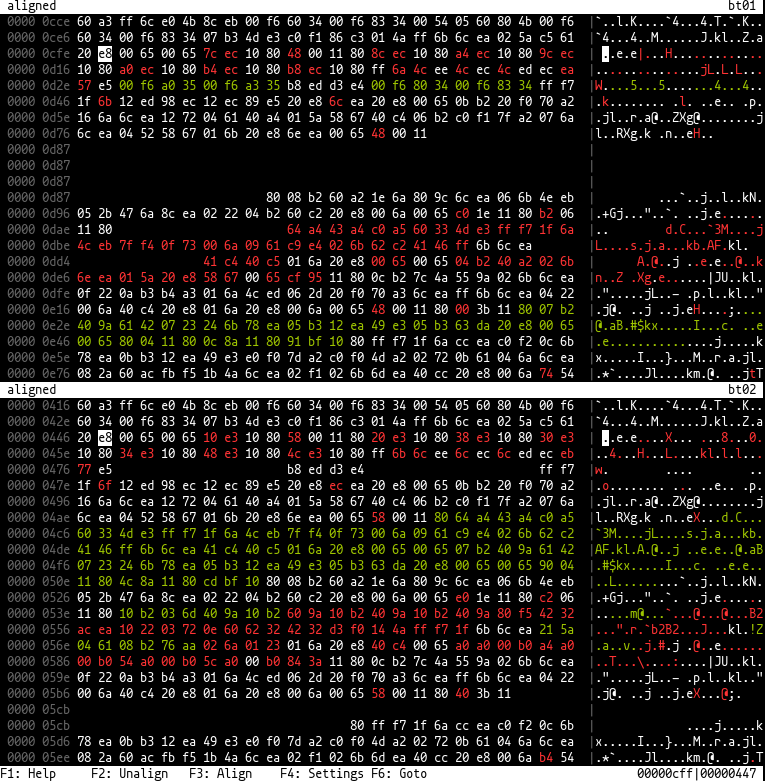
이 도구는 두 개의 이진 파일을 나란히 보여줄 수 있으므로 비슷한 장소가 양쪽에서 동일한 위치에 있고 한쪽에서 누락 된 바이트가 패딩됩니다. 이를 위해 'WFA2'또는 rust-bio 라이브러리 (일반적으로 DNA 서열 정렬에 사용)의 생체 정보 알고리즘을 사용합니다. 구성을위한 대화 상자는 cursive 사용하여 수행됩니다.
[ , ] , 0 눌러 조정 가능 터미널에서 biodiff file_a file_b 실행하면 두 개의 파일을 나란히 보여주는 16 진 뷰로 삭제해야합니다. 처음에는 파일이 각 측면에 간격이 없으면 정렬되지 않고 표시되지 않습니다. 커서와 뷰를 왼쪽과 오른쪽이 비슷하고 F3 (또는 3 )을 누르는 장소로 이동함으로써 정렬 될 수 있습니다. 이것은 표준 구성에서 블록별로 블록으로 수행되므로 커서 근처의 바이트가 먼저 정렬되고 추가 정렬 된 블록이 나중에 양쪽에 표시됩니다.
F4 사용하여 설정을 변경하여 (한 번에 전체 파일의) 글로벌 정렬을 수행 할 수도 있습니다 (매개 변수의 도움말을 참조하십시오). 일반적으로 2 차 시간과 공간이 필요하기 때문에 64KB보다 큰 파일의 경우 글로벌 정렬이 잘 작동하지 않습니다. 더 빠르지 만 약간 덜 정확한 "밴드"알고리즘도 있습니다.
한 파일에서 영역을 선택할 수도 있고 F3을 눌러 정렬 알고리즘은 선택한 바이트를 패턴으로 사용하여 다른 파일의 해당 바이트를 찾는 반 글로벌 정렬을 수행합니다.
biodiff --print file_a file_b 사용하여 Diff를 터미널에 직접 인쇄 할 수도 있습니다. 이 경우 (파일이 너무 오래 걸리지 않기에 충분히 작다면) -gglobal 플래그를 추가하여 글로벌 정렬을 수행 할 수 있습니다 (대화식 사용에 더 적합한 블록과는 반대로).
운이 좋으면 기본 패키지 관리자에 사용할 수있는 패키지가 있습니다. Repology 페이지를 참조하십시오. 릴리스 페이지의 일부 환경에 대한 다운로드 가능한 이진 파일이 있어야합니다. 또는 cargo install biodiff 사용하여 cargo 을 사용 하여이 정보를 설치할 수도 있습니다. 컴파일하려면 wfa2 기능을 위해 CMAKE를 설치해야합니다. Windows를 사용하는 경우 wfa2 지원하려면 x86_64-unknown-linux-gnu 대상을 사용해야합니다.
cargo run --release -- file_a file_b 실행 하여이 저장소에서 코드를 사용하여 직접 사용하여 직접 실행할 수도 있습니다. 구성 파일은 태그 릴리스간에 호환성을 유지하도록 보장됩니다.
기본적으로 설정은 플랫폼 별 사용자 디렉토리에 저장됩니다. 사용자 정의 설정 디렉토리를 사용하려면 biodiff 실행하기 전에 BIODIFF_CONFIG_DIR 환경 변수를 원하는 디렉토리 경로로 설정하십시오. 디렉토리가 존재하지 않으면 자동으로 생성됩니다.
이 프로젝트는 MIT 라이센스에 따라 라이센스가 부여됩니다.