Vergleichen Sie Binärdateien mithilfe von Alignment -Algorithmen.
Dies ist ein Werkzeug für binäre Differenzierung.
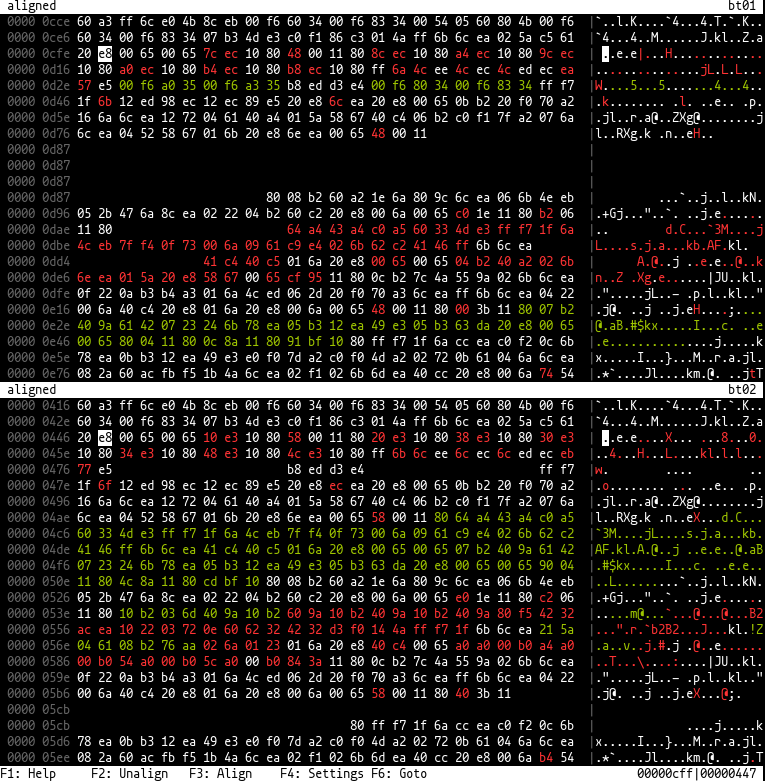
Das Tool kann zwei binäre Dateien nebeneinander anzeigen, so dass ähnliche Orte auf beiden Seiten an derselben Position liegen und von einer Seite fehlt von Bytes gepolstert werden. Dafür verwendet Bio-Informatics-Algorithmen aus der Bibliothek "WFA2" oder rust-bio (typischerweise für die DNA-Sequenzausrichtung verwendet). Die Dialogfelder für die Konfiguration werden mit cursive durchgeführt.
[ , ] , 0 Führen Sie biodiff file_a file_b in einem Terminal aus und Sie sollten in eine Hex -Ansicht fallen, die zwei Dateien nebeneinander angezeigt wird. Zunächst werden die Dateien nicht ohne Lücken auf jeder Seite ausgerichtet und angezeigt. Durch Bewegen des Cursors und der Ansichten an einen Ort, an dem die linke und rechte Seite ähnlich ist und F3 (oder 3 ) drückt, können sie ausgerichtet werden. Dies geschieht nach Block in der Standardkonfiguration, was bedeutet, dass Bytes in der Nähe des Cursors zuerst ausgerichtet sind und später auf beiden Seiten weiter ausgerichtete Blöcke angezeigt werden.
Es ist auch möglich, eine globale Ausrichtung (der gesamten Dateien auf einmal) durchzuführen, indem die Einstellungen mit F4 geändert werden (wenden Sie sich an die Hilfe in den Parametern). Im Allgemeinen funktioniert die globale Ausrichtung, da es quadratische Zeit und Raum benötigt, für Dateien, die größer als 64 KB sind, nicht gut. Es gibt auch einen "gebundenen" Algorithmus, der schneller, aber etwas weniger genau ist.
Sie können auch eine Region in einer Datei auswählen und durch Drücken von F3 den Ausrichtungsalgorithmus eine semiglobale Ausrichtung unter Verwendung der ausgewählten Bytes als Muster durchführen, um die entsprechenden Bytes in der anderen Datei zu ermitteln.
Es ist auch möglich, den Diff direkt in das Terminal zu drucken, indem Sie biodiff --print file_a file_b verwenden. In diesem Fall (wenn die Dateien klein genug sind, um nicht zu lange zu dauern) können Sie das -gglobal -Flag hinzufügen, um eine globale Ausrichtung durchzuführen (im Gegensatz zu einem blockweise, der besser für die interaktive Verwendung geeignet ist).
Wenn Sie Glück haben, gibt es in Ihrem Primärpaket -Manager ein Paket. Siehe die Seite Repology. Für einige Umgebungen auf der Seite "Releases" sollten es für einige Umgebungen herunterladbare Binärdateien vorhanden sein. Alternativ können Sie diese auch mit cargo durch die cargo install biodiff installieren. Sie benötigen CMake für die wfa2 -Funktion zum Kompilieren. Beachten Sie, dass Sie für den Fall, dass Sie Windows verwenden, das Ziel x86_64-unknown-linux-gnu verwenden müssen, wenn Sie wfa2 Unterstützung erhalten möchten.
Sie können auch direkt mit dem Code aus diesem Repository ausführen, indem Sie cargo run --release -- file_a file_b ausführen. Beachten Sie, dass Konfigurationsdateien nur garantiert zwischen Tagged -Releases kompatibel bleiben.
Standardmäßig werden Einstellungen in einem plattformspezifischen Benutzerverzeichnis gespeichert. Um ein benutzerdefiniertes Einstellungsverzeichnis zu verwenden, setzen Sie die Umgebungsvariable BIODIFF_CONFIG_DIR auf den gewünschten Verzeichnispfad, bevor Sie biodiff ausführen. Wenn das Verzeichnis nicht vorhanden ist, wird es automatisch erstellt.
Dieses Projekt ist unter der MIT -Lizenz lizenziert.