FLAG
1.0.0
靶蛋白與結構基序結合的分子產生
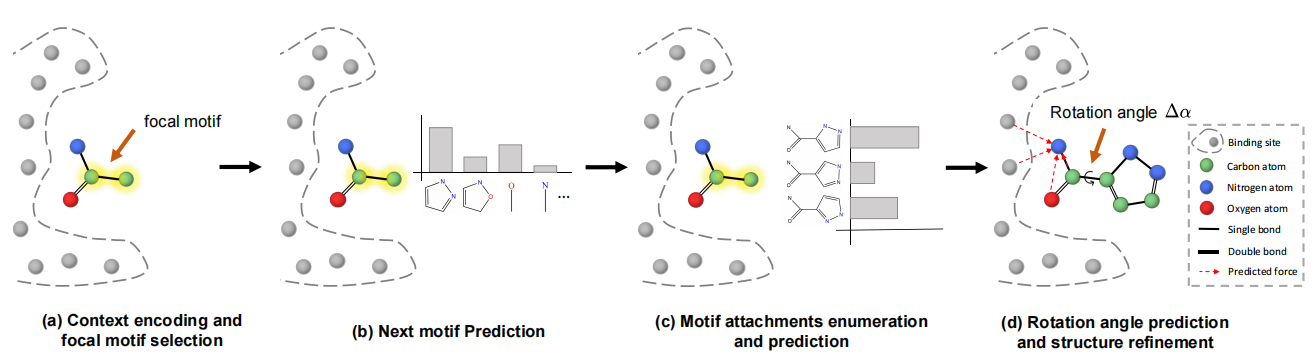
設計與特定蛋白質結合位點結合的配體分子是基於結構的藥物設計的基本問題。儘管深層生成模型和幾何深度學習在藥物設計方面取得了巨大進展,但現有的作品要么在2D圖空間中進行樣品,要么無法生成具有現實子結構的有效分子。為了解決這些問題,我們提出了一個基於侵犯的l ig和g end g勢框架( FLAG ),以生成具有有效且現實的子結構劃分的3D分子。在旗幟中,通過在數據集中提取常見的分子片段(IE,基序)來構建基序詞彙。在每個一代步驟中,首先使用3D圖神經網絡編碼中間上下文信息。然後,我們的模型選擇焦點基序,預測下一種基序類型,並附加新基序。鍵長/角可以通過化學信息素工具快速準確地確定。最後,根據預測的旋轉角度和結構細化,進一步調整了分子幾何形狀。我們的模型不僅可以在常規指標(例如結合親和力,QED和SA)上實現競爭性能,而且在產生具有逼真的子結構的分子方面,差距均優於基準。
conda env create -f flag_env.yaml
conda activate flag_env請參閱data文件夾中的README.md 。
python build_vocab.py
python train.py
python motif_sample.py
演示:https://huggingface.co/spaces/zaixi/iclr_flag
檢查點:https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp = drive_link
100個蛋白質靶標的生成的分子結構存儲在flag_gen.pt.pt中
索引文件是test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}