구조적 모티프와 표적 단백질 결합을위한 분자 생성
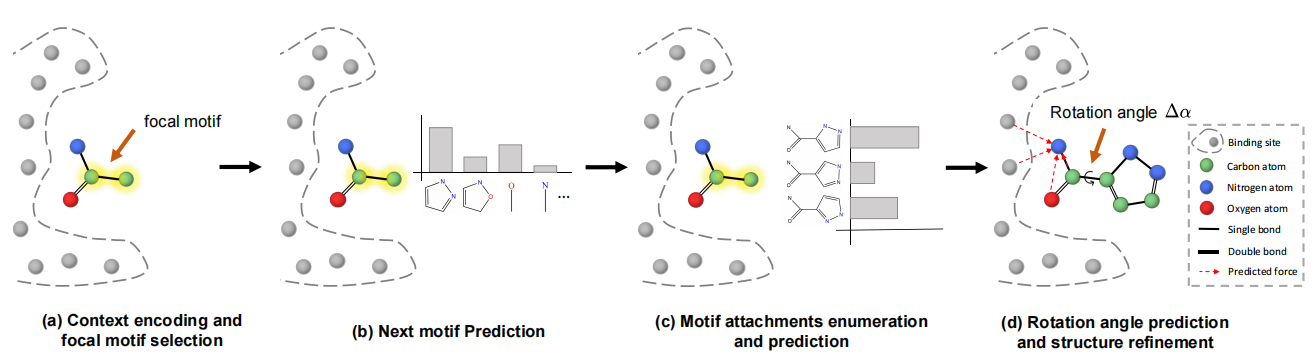
특정 단백질 결합 부위에 결합하는 리간드 분자 설계는 구조 기반 약물 설계에서 근본적인 문제입니다. 심층 생성 모델과 기하학적 딥 러닝은 약물 설계에서 큰 진전을 보였지만, 기존의 작품은 2D 그래프 공간의 샘플 또는 현실적인 하위 구조로 유효한 분자를 생성하지 못합니다. 이러한 문제를 해결하기 위해, 우리는 유효하고 현실적인 하위 구조 단편을 가진 3D 분자를 생성하기 위해 Fragment -based l ig a nd g eneration framework ( FLAG )를 제안합니다. 플래그에서, 모티프 어휘는 데이터 세트에서 일반적인 분자 단편 (즉, 모티프)을 추출하여 구성됩니다. 각 세대 단계에서 3D 그래프 신경망이 먼저 중간 컨텍스트 정보를 인코딩하기 위해 사용됩니다. 그런 다음 우리 모델은 초점 모티프를 선택하고 다음 주제 유형을 예측하고 새로운 모티프를 첨부합니다. 결합 길이/각도는 Cheminformatics 도구에 의해 빠르고 정확하게 결정될 수 있습니다. 마지막으로, 분자 형상은 예측 된 회전 각도 및 구조 정제에 따라 추가로 조정된다. 우리의 모델은 바인딩 친화력, QED 및 SA와 같은 기존의 메트릭에 대한 경쟁력있는 성능을 달성 할뿐만 아니라 현실적인 하위 구조로 분자를 생성하는 데 큰 마진으로 기준선을 능가합니다.
conda env create -f flag_env.yaml
conda activate flag_env data 폴더의 README.md 참조하십시오.
python build_vocab.py
python train.py
python motif_sample.py
데모 : https://huggingface.co/spaces/zaixi/iclr_flag
CheckPoints : https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp=drive_link
100 개의 단백질 표적에 대한 생성 된 분자 구조는 flag_gen.pt에 저장됩니다.
인덱스 파일은 test_index.pkl입니다
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}