構造モチーフとの標的タンパク質結合の分子生成
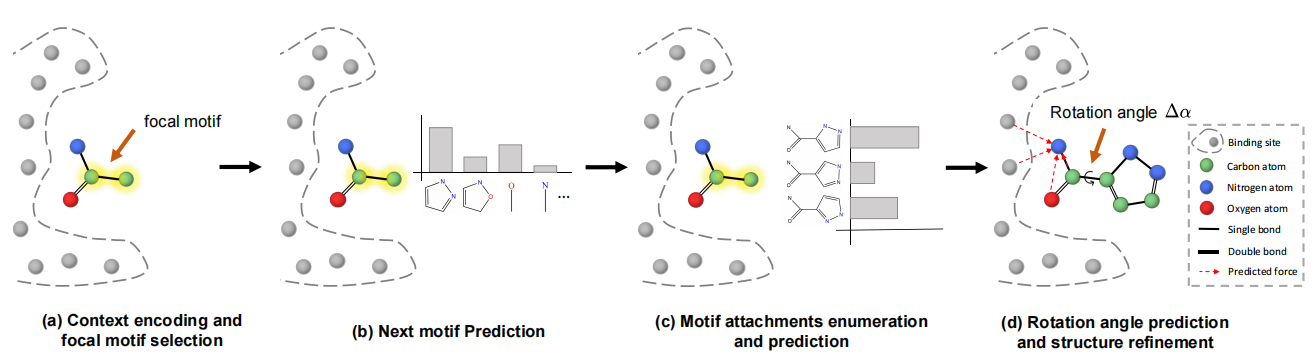
特定のタンパク質結合部位に結合するリガンド分子の設計は、構造ベースの薬物設計における根本的な問題です。深い生成モデルと幾何学的な深い学習は、薬物設計で大きな進歩を遂げていますが、既存の作業は2Dグラフ空間でサンプルするか、現実的なサブ構造を持つ有効な分子を生成できません。これらの問題に取り組むために、 fラグメントベースのl ig and g enerationフレームワーク( FLAG )を提案し、有効で現実的なサブ構造の断片ごとに3D分子を生成します。フラグでは、データセットに共通の分子フラグメント(つまり、モチーフ)を抽出することにより、モチーフ語彙が構築されます。各世代のステップで、3Dグラフニューラルネットワークが最初に採用され、中間コンテキスト情報をエンコードします。次に、モデルは焦点モチーフを選択し、次のモチーフタイプを予測し、新しいモチーフを取り付けます。結合長/角度は、化学情報ツールによって迅速かつ正確に決定できます。最後に、分子ジオメトリは、予測された回転角と構造の改良に従ってさらに調整されます。私たちのモデルは、結合親和性、QED、SAなどの従来のメトリックで競争力のあるパフォーマンスを達成するだけでなく、現実的な下部構造を持つ分子を生成する際に大きなマージンでベースラインを上回ります。
conda env create -f flag_env.yaml
conda activate flag_envdataフォルダーのREADME.mdを参照してください。
python build_vocab.py
python train.py
python motif_sample.py
デモ:https://huggingface.co/spaces/zaixi/iclr_flag
チェックポイント:https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp=drive_link
100のタンパク質ターゲットの生成された分子構造は、flag_gen.ptに保存されます
インデックスファイルはtest_index.pklです
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}