Génération de molécules pour la liaison des protéines cibles avec des motifs structurels
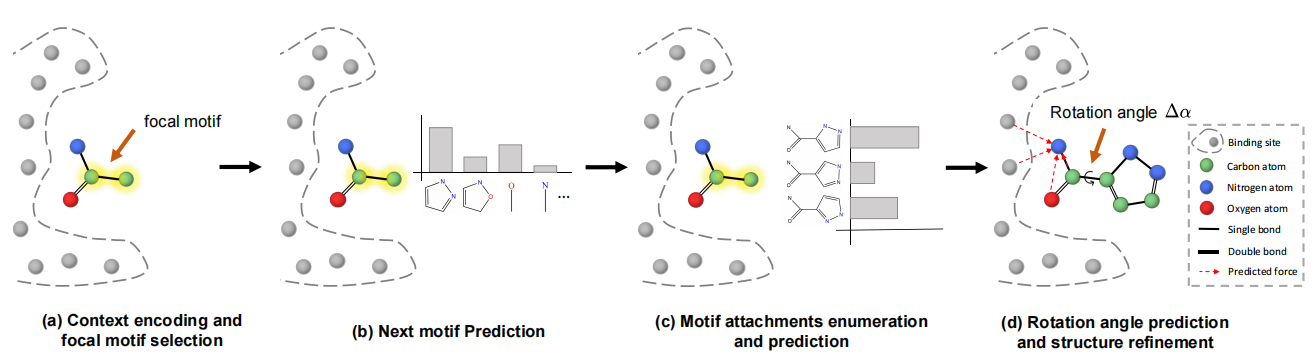
La conception de molécules de ligands qui se lient à des sites de liaison aux protéines spécifiques est un problème fondamental dans la conception de médicaments basée sur la structure. Bien que les modèles génératifs profonds et l'apprentissage en profondeur géométrique aient fait de grands progrès dans la conception de médicaments, les œuvres existantes échantillonnent dans l'espace graphique 2D ou ne parviennent pas à générer des molécules valides avec des sous-structures réalistes. Pour s'attaquer à ces problèmes, nous proposons un cadre l ig basé sur le ragnement et le cadre ( FLAG ), pour générer des molécules 3D avec des substances substantielles valides et réalistes fragment par fragment. Dans le drapeau, un vocabulaire du motif est construit en extraitant des fragments moléculaires courants (c.-à-d. Le motif) dans l'ensemble de données. À chaque étape de génération, un réseau de neurones graphiques 3D est d'abord utilisé pour coder les informations de contexte intermédiaire. Ensuite, notre modèle sélectionne le motif focal, prédit le type de motif suivant et attache le nouveau motif. Les longueurs / angles de liaison peuvent être déterminés rapidement et avec précision par les outils de chimiminformatique. Enfin, la géométrie moléculaire est encore ajustée en fonction de l'angle de rotation prévu et du raffinement de la structure. Notre modèle réalise non seulement des performances concurrentielles sur des mesures conventionnelles telles que l'affinité de liaison, le QED et la SA, mais surpasse également les lignes de base par une grande marge dans la génération de molécules avec des sous-structures réalistes.
conda env create -f flag_env.yaml
conda activate flag_env Veuillez vous référer à README.md dans le dossier data .
python build_vocab.py
python train.py
python motif_sample.py
Demo: https://huggingface.co/spaces/zaixi/iclr_flag
Points de contrôle: https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp=drive_link
Les structures moléculaires générées pour 100 cibles protéiques sont stockées dans Flag_gen.pt
Le fichier d'index est test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}