Moleküleerzeugung für Zielproteinbindung mit Strukturmotiven
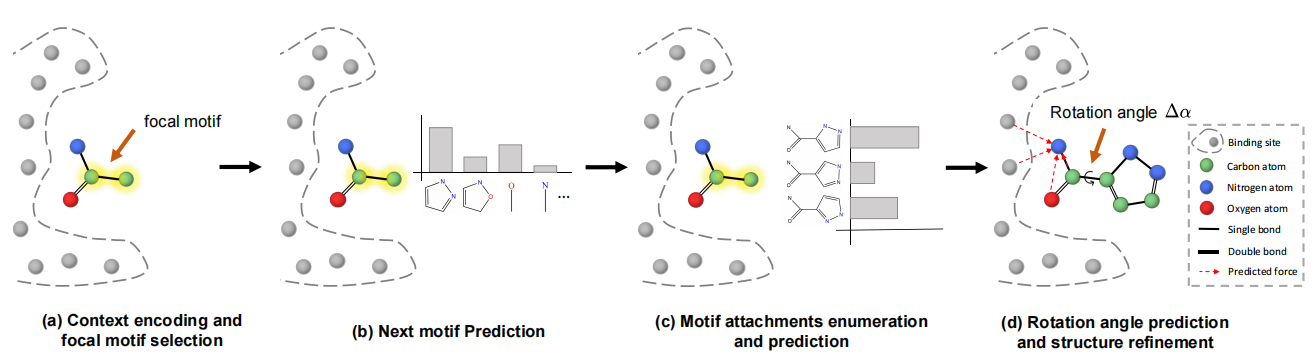
Das Design von Ligandenmolekülen, die an spezifische Proteinbindungsstellen binden, ist ein grundlegendes Problem im strukturbasierten Arzneimitteldesign. Obwohl tiefe generative Modelle und geometrisches Deep -Lernen große Fortschritte beim Arzneimitteldesign erzielt haben, arbeiten vorhandene Arbeiten entweder im 2D -Graphenraum oder erzeugen keine gültigen Moleküle mit realistischen Unterstrukturen. Um diese Probleme anzugehen, schlagen wir ein F- Ragment-basiertes LIG und G- Eneration-Framework ( FLAG ) vor, um 3D-Moleküle mit gültigen und realistischen Unterstrukturen zu erzeugen, die Fragment-by-Fragment-Fragments erzeugen. Im Flag wird ein Motivvokabular konstruiert, indem gemeinsame molekulare Fragmente (dh Motiv) im Datensatz extrahiert werden. Bei jedem Generationsschritt wird zunächst ein 3D -Diagramm -Netzwerk verwendet, um die intermediären Kontextinformationen zu codieren. Dann wählt unser Modell das Fokusmotiv aus, sagt den nächsten Motivtyp voraus und fügt das neue Motiv an. Die Bindungslängen/Winkel können durch Cheminformatikwerkzeuge schnell und genau bestimmt werden. Schließlich wird die molekulare Geometrie nach dem vorhergesagten Rotationswinkel und der Strukturverfeinerung weiter eingestellt. Unser Modell erzielt nicht nur wettbewerbsfähige Leistungen zu herkömmlichen Metriken wie Bindungsaffinität, QED und SA, sondern übertrifft auch die Baselines mit einem großen Rand bei der Erzeugung von Molekülen mit realistischen Unterstrukturen.
conda env create -f flag_env.yaml
conda activate flag_env Weitere Informationen finden Sie in README.md im data .
python build_vocab.py
python train.py
python motif_sample.py
Demo: https://huggingface.co/spaces/zaixi/iclr_flag
Kontrollpunkte: https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp=Drive_Link
Die erzeugten molekularen Strukturen für 100 Proteinziele werden in Flag_gen.PT gespeichert
Die Indexdatei ist test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}