การสร้างโมเลกุลสำหรับโปรตีนเป้าหมายผูกพันกับลวดลายโครงสร้าง
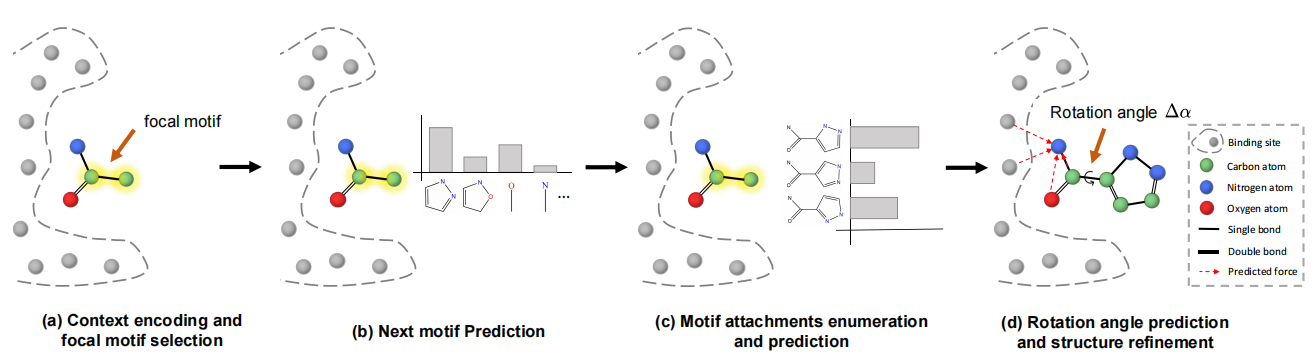
การออกแบบโมเลกุลแกนด์ที่ผูกกับไซต์ที่มีผลผูกพันโปรตีนเฉพาะเป็นปัญหาพื้นฐานในการออกแบบยาตามโครงสร้าง แม้ว่าแบบจำลองการกำเนิดที่ลึกและการเรียนรู้เชิงลึกทางเรขาคณิตมีความก้าวหน้าอย่างมากในการออกแบบยา แต่งานที่มีอยู่ตัวอย่างในพื้นที่กราฟ 2D หรือไม่สามารถสร้างโมเลกุลที่ถูกต้องด้วยโครงสร้างย่อยที่สมจริง เพื่อจัดการกับปัญหาเหล่านี้เราเสนอกรอบการ ใช้ พลังงาน ที่เป็นพื้นฐานของ การ ใช้ งาน ( FLAG ) เพื่อสร้างโมเลกุล 3 มิติที่มีโครงสร้างย่อยที่ถูกต้องและสมจริง ในแฟล็กคำศัพท์ motif ถูกสร้างขึ้นโดยการสกัดชิ้นส่วนโมเลกุลทั่วไป (เช่น motif) ในชุดข้อมูล ในแต่ละขั้นตอนเครือข่ายประสาทกราฟ 3 มิติจะถูกใช้เป็นครั้งแรกเพื่อเข้ารหัสข้อมูลบริบทระดับกลาง จากนั้นโมเดลของเราจะเลือกลวดลายโฟกัสทำนายประเภท motif ถัดไปและแนบลวดลายใหม่ ความยาวของพันธะ/มุมสามารถกำหนดได้อย่างรวดเร็วและแม่นยำโดยเครื่องมือเคมี ในที่สุดเรขาคณิตโมเลกุลจะถูกปรับเพิ่มเติมตามมุมการหมุนที่คาดการณ์ไว้และการปรับแต่งโครงสร้าง แบบจำลองของเราไม่เพียง แต่ประสบความสำเร็จในการแข่งขันในการวัดทั่วไปเช่นความสัมพันธ์ที่มีผลผูกพัน, QED และ SA เท่านั้น
conda env create -f flag_env.yaml
conda activate flag_env โปรดดู README.md ในโฟลเดอร์ data
python build_vocab.py
python train.py
python motif_sample.py
ตัวอย่าง: https://huggingface.co/spaces/zaixi/iclr_flag
จุดตรวจ: https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?USP=DRIVE_LINK
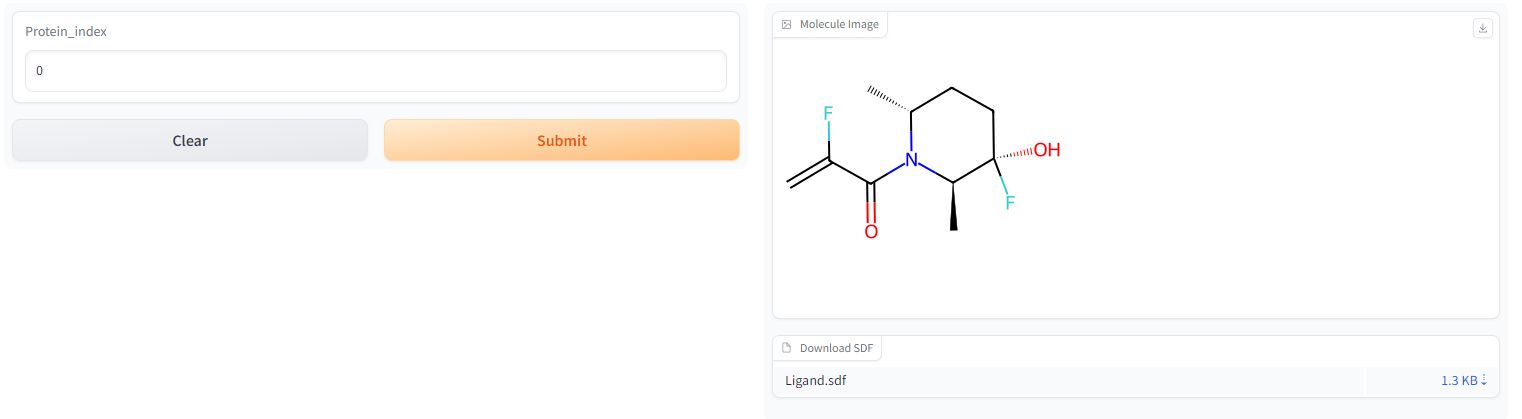
โครงสร้างโมเลกุลที่สร้างขึ้นสำหรับเป้าหมายโปรตีน 100 รายการจะถูกเก็บไว้ใน Flag_Gen.pt
ไฟล์ดัชนีคือ test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}