Geração de moléculas para ligação à proteína alvo com motivos estruturais
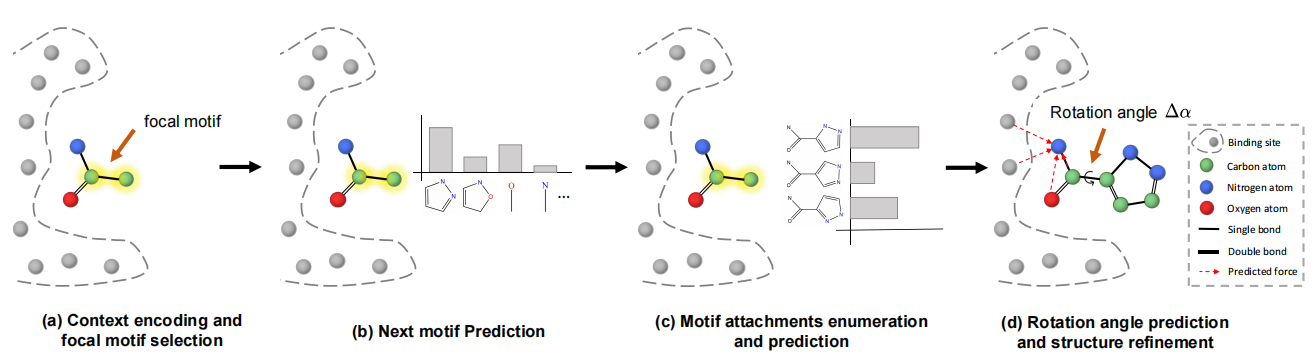
Projetar moléculas de ligante que se ligam a locais específicos de ligação a proteínas é um problema fundamental no design de medicamentos baseados em estrutura. Embora modelos generativos profundos e aprendizado geométrico profundo tenham feito um grande progresso no design de medicamentos, os trabalhos existentes amostram no espaço de gráficos 2D ou falham em gerar moléculas válidas com subestruturas realistas. Para resolver esses problemas, propomos uma estrutura de enriquecimento baseada em tragamento ( Flag ), para gerar moléculas 3D com subestruturas válidas e realistas fragmentação por fragmentos. Na bandeira, um vocabulário de motivos é construído extraindo fragmentos moleculares comuns (isto é, motivos) no conjunto de dados. Em cada etapa da geração, uma rede neural gráfica 3D é empregada pela primeira vez para codificar as informações de contexto intermediário. Em seguida, nosso modelo seleciona o motivo focal, prevê o próximo tipo de motivo e anexa o novo motivo. Os comprimentos/ângulos de ligação podem ser determinados com rapidez e precisão pelas ferramentas de quiminformática. Finalmente, a geometria molecular é ainda ajustada de acordo com o ângulo de rotação previsto e o refinamento da estrutura. Nosso modelo não apenas atinge performances competitivas em métricas convencionais, como afinidade de ligação, QED e SA, mas também supera as linhas de base por uma grande margem na geração de moléculas com subestruturas realistas.
conda env create -f flag_env.yaml
conda activate flag_env Consulte o README.md na pasta data .
python build_vocab.py
python train.py
python motif_sample.py
Demo: https://huggingface.co/spaces/zaixi/iclr_flag
Pontos de verificação: https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp=drive_link
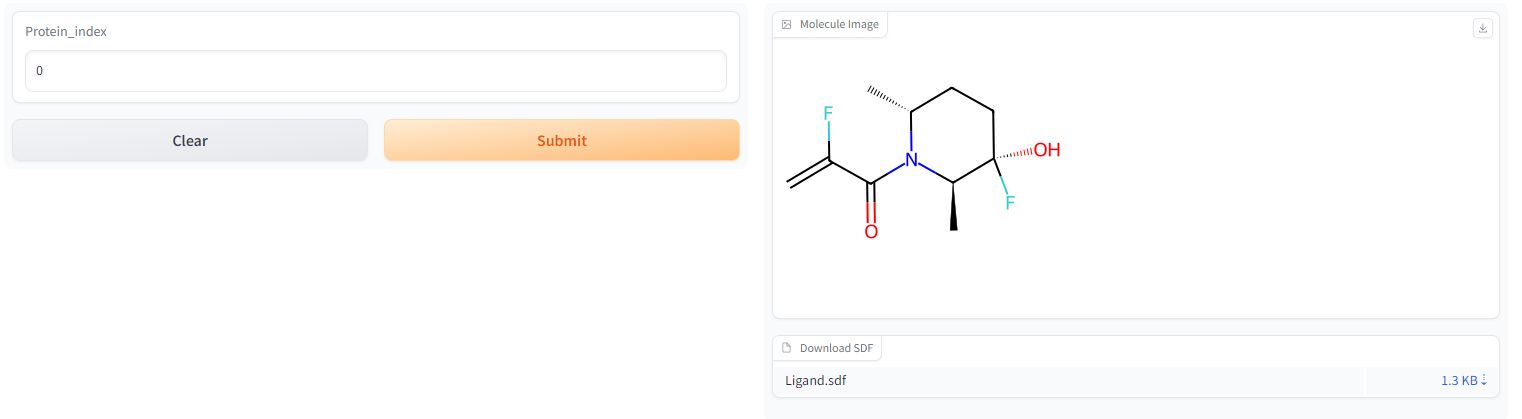
As estruturas moleculares geradas para 100 alvos de proteínas são armazenadas em flag_gen.pt
O arquivo de índice é test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}