Generación de moléculas para la unión de proteína diana con motivos estructurales
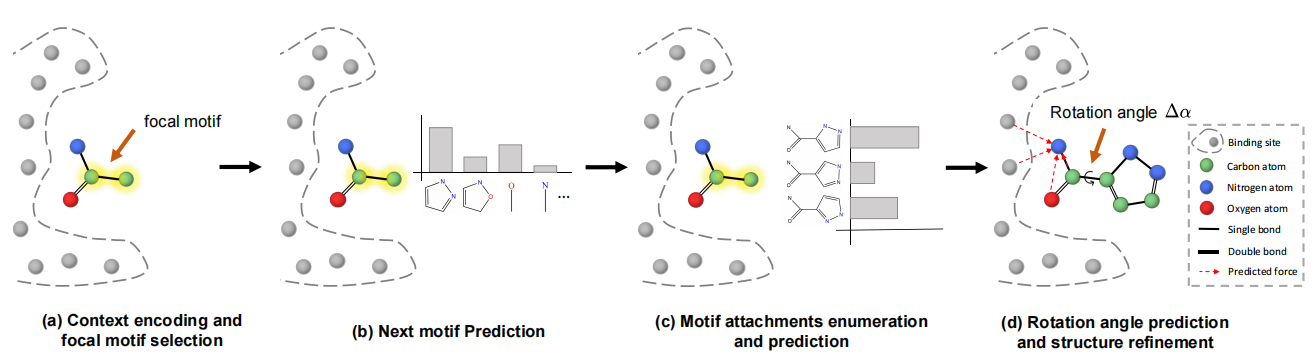
El diseño de moléculas de ligando que se unen a sitios específicos de unión de proteínas es un problema fundamental en el diseño de fármacos basados en la estructura. Aunque los modelos generativos profundos y el aprendizaje profundo geométrico han hecho un gran progreso en el diseño de fármacos, los trabajos existentes muestran en el espacio de gráficos 2D o no generan moléculas válidas con subestructuras realistas. Para abordar estos problemas, proponemos un marco de la Eneeration ( FLAG ) basado en el rago basado en ragmment para generar moléculas 3D con subestructuras válidas y realistas fragmento por fragmento. En la bandera, se construye un vocabulario de motivos extrayendo fragmentos moleculares comunes (es decir, motivo) en el conjunto de datos. En cada paso de generación, primero se emplea una red neuronal de gráfico 3D para codificar la información de contexto intermedio. Luego, nuestro modelo selecciona el motivo focal, predice el siguiente tipo de motivo y adjunta el nuevo motivo. Las longitudes/ángulos de enlace se pueden determinar rápida y precisión mediante herramientas químicas. Finalmente, la geometría molecular se ajusta aún más de acuerdo con el ángulo de rotación predicho y el refinamiento de la estructura. Nuestro modelo no solo logra actuaciones competitivas en métricas convencionales como la afinidad de unión, QED y SA, sino que también supera a las líneas de base por un gran margen en la generación de moléculas con subestructuras realistas.
conda env create -f flag_env.yaml
conda activate flag_env Consulte README.md en la carpeta data .
python build_vocab.py
python train.py
python motif_sample.py
Demostración: https://huggingface.co/spaces/zaixi/iclr_flag
Puntos de control: https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9??usp=drive_link
Las estructuras moleculares generadas para 100 objetivos de proteínas se almacenan en flag_gen.pt
El archivo de índice es test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}