靶蛋白与结构基序结合的分子产生
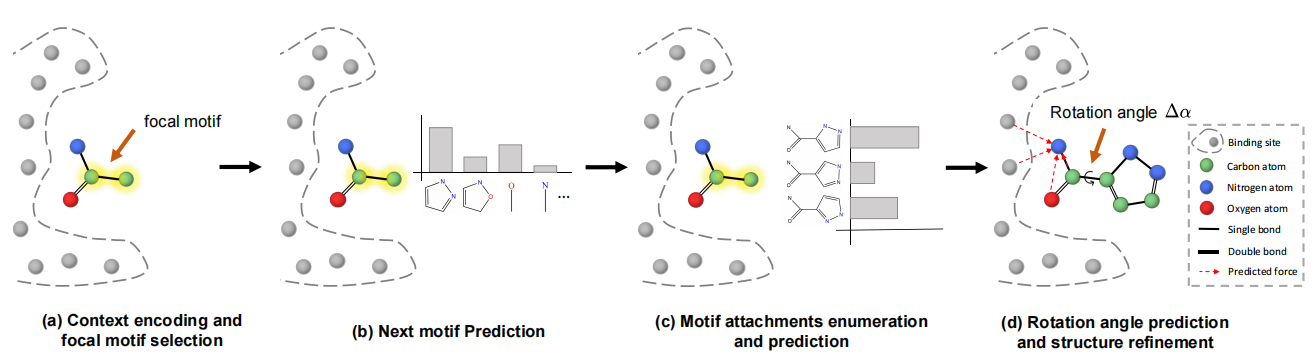
设计与特定蛋白质结合位点结合的配体分子是基于结构的药物设计的基本问题。尽管深层生成模型和几何深度学习在药物设计方面取得了巨大进展,但现有的作品要么在2D图空间中进行样品,要么无法生成具有现实子结构的有效分子。 To tackle these problems, we propose a F ragment-based L ig A nd G eneration framework ( FLAG ), to generate 3D molecules with valid and realistic substructures fragment-by-fragment.在旗帜中,通过在数据集中提取常见的分子片段(IE,基序)来构建基序词汇。在每个一代步骤中,首先使用3D图神经网络编码中间上下文信息。然后,我们的模型选择焦点基序,预测下一种基序类型,并附加新基序。键长/角可以通过化学信息素工具快速准确地确定。最后,根据预测的旋转角度和结构细化,进一步调整了分子几何形状。我们的模型不仅可以在常规指标(例如结合亲和力,QED和SA)上实现竞争性能,而且在产生具有逼真的子结构的分子方面,差距均优于基准。
conda env create -f flag_env.yaml
conda activate flag_envPlease refer to README.md in the data folder.
python build_vocab.py
python train.py
python motif_sample.py
演示:https://huggingface.co/spaces/zaixi/iclr_flag
检查点:https://drive.google.com/drive/folders/1ni-tl7yzymsfljezxatxbpuio7lvubt9?usp = drive_link
100个蛋白质靶标的生成的分子结构存储在flag_gen.pt.pt中
索引文件是test_index.pkl
@inproceedings{
zhang2023molecule,
title={Molecule Generation For Target Protein Binding with Structural Motifs},
author={ZAIXI ZHANG and Shuxin Zheng and Yaosen Min and Qi Liu},
booktitle={International Conference on Learning Representations},
year={2023},
url={https://openreview.net/forum?id=Rq13idF0F73}
}