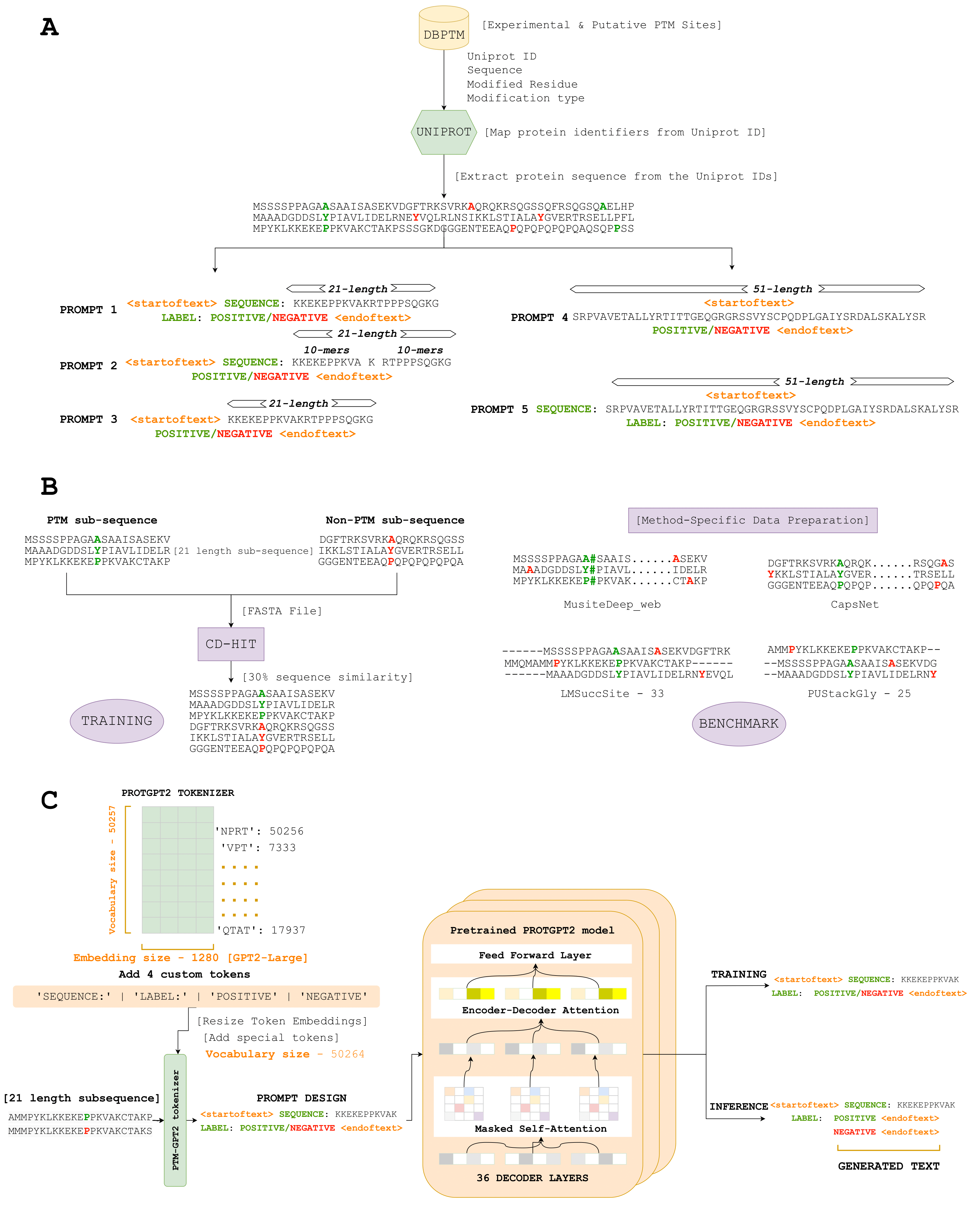
Здесь мы вводим PTMGPT2, набор моделей, способных генерировать токены, которые обозначают модифицированные последовательности белка, решающие для идентификации сайтов PTM. В основе этой платформы лежит ProTGPT2, модель авторегрессии трансформатора. Мы адаптировали ProTGPT2, используя его в качестве предварительно обученной модели, и дополнительно настраивали его для специфической задачи генерации классификационных меток для данного типа PTM. Уникально, PTMGPT2 использует архитектуру только для декодера, которая устраняет необходимость в специфической для задачи главы класса класса во время обучения. Вместо этого последний слой декодера функционирует как проекция обратно в словарное пространство, эффективно генерируя следующий возможный токен на основе изученных шаблонов между токенами в приглашении ввода.
Ссылка - (https://nsclbio.jbnu.ac.kr/gpt_model/)
Свяжитесь с нами непосредственно по адресу [email protected] для объема прогнозов и обученных моделей
Ссылка - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
Ссылка - (https://doi.org/10.5281/zenodo.11371883)
Ссылка - (https://zenodo.org/records/11362322)
Ссылка - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
Трансформеры 4.29.2
Scikit-learn 1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
• Модель: эта папка проводит образцевую модель, предназначенную для прогнозирования сайтов PTM из заданных белковых последовательностей, иллюстрируя применение PTMGPT2.
• Токенизатор: эта папка содержит образцек -токенизатор, ответственный за токенизацию белковых последовательностей, включая токены ручной работы для определенных аминокислот или мотивов.
• sepence.ipynb: Этот файл предоставляет исполняемый код для применения модели PTMGPT2 и токенизатора для прогнозирования сайтов PTM, служащий практическим руководством для пользователей для применения модели к своим наборам данных.