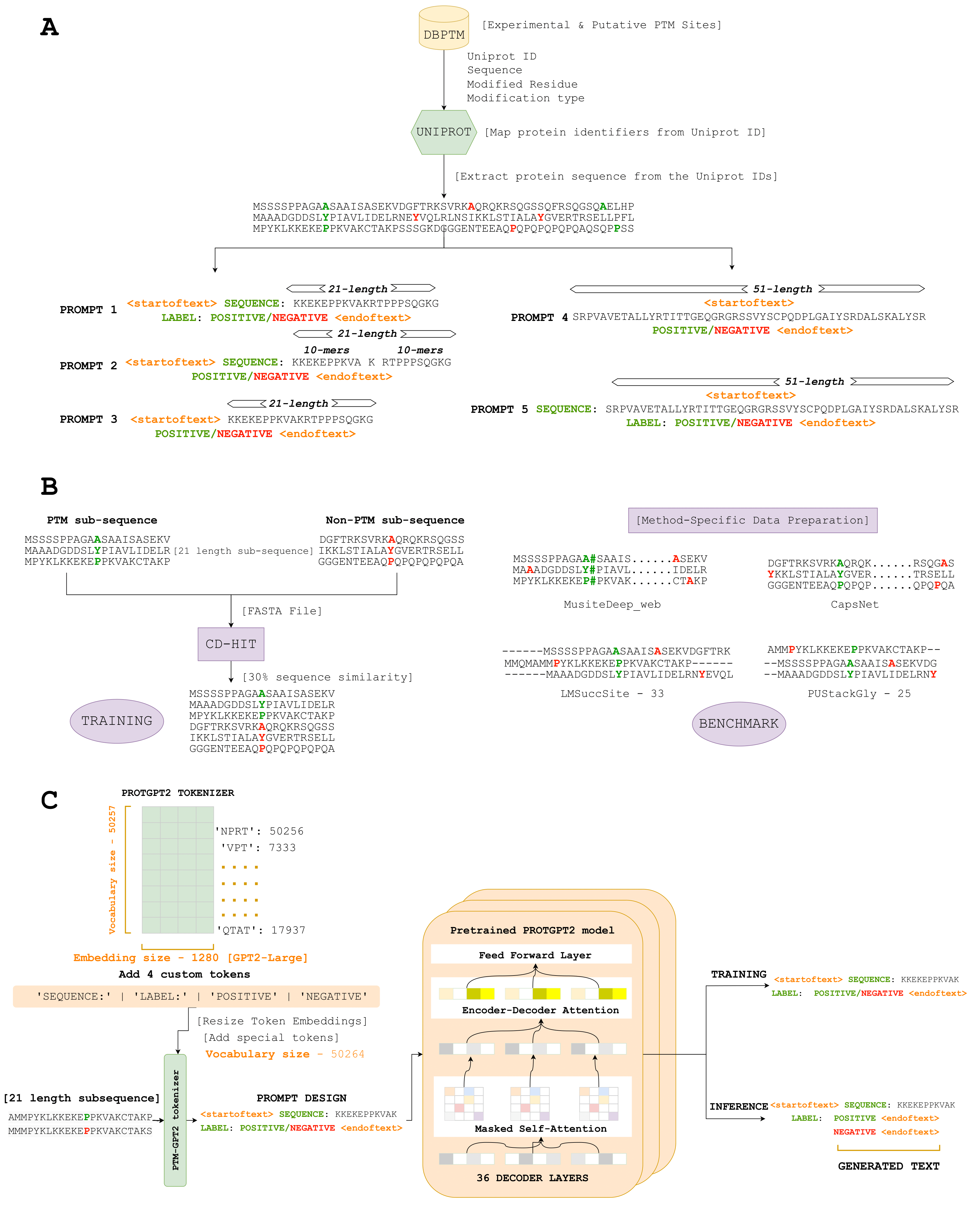
Ici, nous introduisons PTMGPT2, une suite de modèles capables de générer des jetons qui signifient des séquences de protéines modifiées, cruciale pour identifier les sites PTM. Au cœur de cette plate-forme se trouve ProtGPT2, un modèle de transformateur autorégressif. Nous avons adapté ProtGPT2, en l'utilisant comme un modèle pré-formé, et l'avons affiné pour la tâche de specification de générer des étiquettes de classification pour un type PTM donné. Uniquement, PTMGPT2 utilise une architecture de décodeur uniquement, ce qui élimine la nécessité d'une tête de classification spécifique à la tâche pendant la formation. Au lieu de cela, la couche finale du décodeur fonctionne comme une projection dans l'espace de vocabulaire, générant efficacement le jeton suivant possible en fonction des modèles appris parmi les jetons dans l'invite d'entrée.
Lien - (https://nsclbio.jbnu.ac.kr/gpt_model/)
Contactez-nous directement à [email protected] pour les prédictions en vrac et les modèles formés
Lien - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
Lien - (https://doi.org/10.5281/zenodo.11371883)
Lien - (https://zenodo.org/records/11362322)
Lien - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
Transformers 4.29.2
Scikit-learn 1.2.2
pytorch 2.0.1
Pytorch-Cuda 11.7
• Modèle: Ce dossier héberge un modèle d'échantillon conçu pour prédire les sites PTM à partir de séquences protéiques données, illustrant l'application de PTMGPT2.
• Tokenizer: Ce dossier contient un échantillon de jetons responsable des séquences de protéines de tokenisage, y compris des jetons fabriqués à la main pour des acides ou des motifs aminés spécifiques.
• Inference.ipynb: Ce fichier fournit un code exécutable pour l'application du modèle PTMGPT2 et du tokenizer pour prédire les sites PTM, servant de guide pratique pour que les utilisateurs appliquent le modèle à leurs ensembles de données.